Cosa fare quando i risultati del qPCR sono strani?
Gli esperimenti qPCR possono sembrare facili e a volte difficili. Dopo aver completato la progettazione del primer, i campioni qPCR vengono aggiunti passo dopo passo come nella PCR e l'esperimento può essere completato sulla macchina. Ci sono diversi problemi comuni negli esperimenti qPCR con il metodo della tintura e, date le possibili cause e soluzioni, speriamo che tu possa usarli per risolvere i problemi negli esperimenti qPCR.
1. Che cos'è la qPCR?
2. Curva di amplificazione qPCR anomala
3. Curva di fusione qPCR anomala
4. Informazioni per l'ordinazione
5. Prodotti correlati
6. Per quanto riguarda la lettura
1. Che cos'è la qPCR?
Da quando è stata inventata la tecnica della reazione a catena della polimerasi (PCR), la PCR è probabilmente la tecnica più ampiamente utilizzata in biologia molecolare grazie alla sua semplicità, economicità, affidabilità, rapidità e alta sensibilità. qPCR è una tecnologia sviluppata dalla tecnologia PCR. Durante il processo di amplificazione del DNA, il metodo di rilevamento della quantità totale di prodotti dopo ogni ciclo di PCR con coloranti fluorescenti non solo ha la rapidità e la sensibilità della PCR, ma ha anche una maggiore specificità e un monitoraggio elevato e in tempo reale, una quantificazione ripetibile e accurata e altri vantaggi. qPCR è una tecnologia che utilizza un dispositivo di amplificazione PCR quantitativa in tempo reale, vale a dire uno strumento qPCR, per monitorare i prodotti di amplificazione degli acidi nucleici in tempo reale durante il processo PCR. qPCR realizza l'analisi quantitativa del modello di partenza attraverso l'analisi del valore Ct e della curva standard. Nel 1992, il giapponese Higuchi propose per primo la "tecnologia PCR quantitativa fluorescente in tempo reale". Nel 1996, l'American Biological Company ha lanciato il primo strumento PCR quantitativo fluorescente al mondo, composto da un sistema di ciclo termico di amplificazione PCR, un sistema ottico di rilevamento della fluorescenza e un software applicativo e per computer. Può monitorare i prodotti di amplificazione degli acidi nucleici in tempo reale tramite coloranti fluorescenti o sonde fluorescenti. , tramite la relazione della funzione matematica, combinata con il software per l'analisi dei risultati, viene realizzato il calcolo della quantità iniziale del modello del campione da testare. Pertanto, la tecnologia PCR quantitativa fluorescente in tempo reale è stata ampiamente utilizzata. I metodi di etichettatura fluorescente per qPCR sono suddivisi in metodo mosaico di colorante fluorescente basato sul metodo di colorante SYBR Green I, metodo sonda fluorescente basato sul metodo sonda Taqman (Cycling Probe, Molecular Bracon, ecc.), metodo primer colorante quencher.
Gli esperimenti qPCR possono sembrare facili e a volte difficili. Dopo aver completato la progettazione del primer, i campioni qPCR vengono aggiunti passo dopo passo come la PCR e l'esperimento può essere completato sulla macchina. Tuttavia, molti piccoli dettagli richiedono attenzione, come nessun liquido appeso alla parete del tubo, nessuna bolla nel tubo e la necessità di ridurre al minimo il contatto diretto tra la parete del tubo, il coperchio del tubo, ecc., per ridurre gli errori sperimentali o strani risultati sperimentali. Xiaoyi ha compilato diversi problemi comuni negli esperimenti qPCR con il metodo della tintura e ha fornito le possibili cause e soluzioni, che speriamo tu possa usare per risolvere i problemi negli esperimenti qPCR.
2. Curva di amplificazione qPCR anomala
2.1 Anomalie nella fase basale del grafico logaritmico della curva di amplificazione
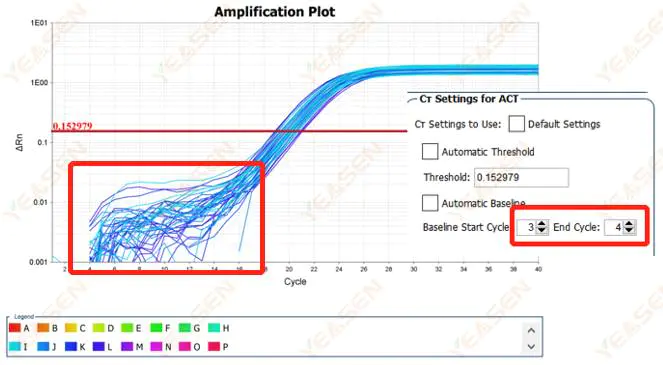
Figura 1. Grafico di amplificazione
Possibili cause:
Impostazione della linea di base non corretta.
Soluzioni:
Si consiglia di aumentare il valore finale della linea di base.Di seguito è riportato un esempio di diagramma di regolazione.

Figura 2. Grafico di amplificazione
2.2 Curva di amplificazione, grafico logaritmico, segmentazione della curva
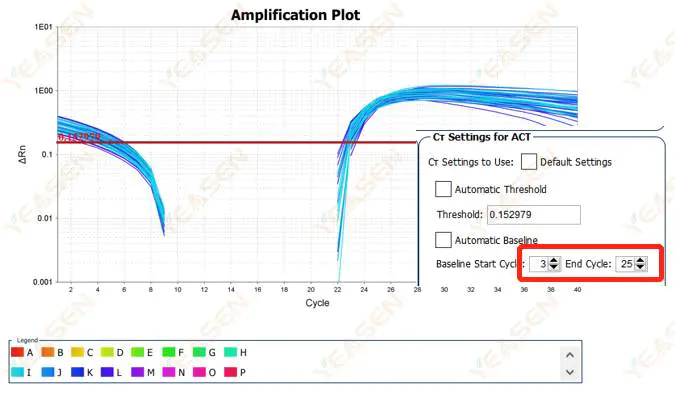
Figura 3. Grafico di amplificazione
Possibili cause:
La linea di base è impostata troppo in alto.
Soluzioni:
Si raccomanda di ridurre il valore finale della linea di base. Il diagramma di esempio di aggiustamento è il seguente.
2.3 Diagramma di linearità della curva di amplificazione non uniforme
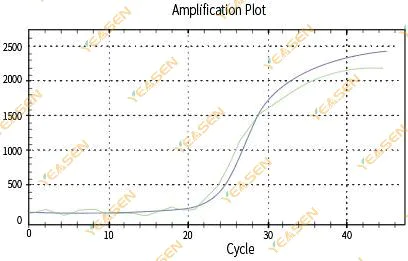
Figura 4. Grafico di amplificazione
Possibili cause:
1) La provetta di reazione PCR non era chiusa ermeticamente e la soluzione di reazione è fuoriuscita.
2)Soluzione di reazione PCR con parete sospesa.
3) Lo strumento non è calibrato (nemmeno la calibrazione automatica o la calibrazione ROX).
4) Il sistema contiene molti inibitori che causano una fluorescenza instabile.
5) L'uso eccessivo dello strumento determina una raccolta di fluorescenza instabile.
Soluzioni:
1) Premere saldamente il coperchio del tubo.
2) Mescolare accuratamente il reagente e, dopo la centrifugazione completa, inserirlo con attenzione nello strumento quantitativo.
3)Calibrare lo strumento.
4) Migliorare la purezza dell'RNA e selezionare i reagenti di trascrizione inversa appropriati.
2.4 Grafico lineare della curva amplificata a zigzag in un plateau
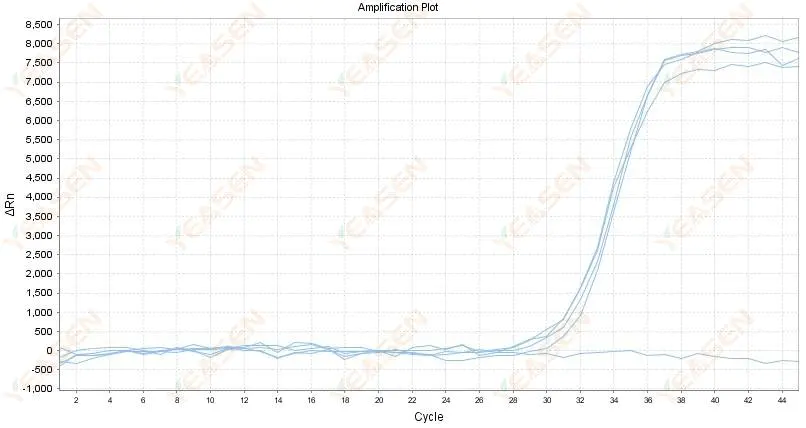
Figura 5. Grafico di amplificazione
Possibili cause:
1) La purezza dell'RNA è scarsa e sono presenti numerose impurità.
2) Lo strumento viene utilizzato per troppo tempo.
Soluzioni:
1) Riestrarre RNA di alta qualità.
2) Diluire lo stampo di RNA per ridurre la concentrazione di impurità.
3) Calibrare lo strumento.
2.5 Il grafico lineare della curva di amplificazione non riesce a raggiungere un plateau
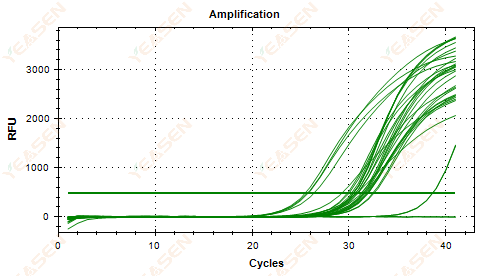
Figura 6. Grafico di amplificazione
Possibili cause:
1) Bassa concentrazione del modello (valore Ct intorno a 35).
2) Cicli di amplificazione troppo pochi.
3) Bassa efficienza di amplificazione del reagente (piccolo Ct, ma anche incapacità di raggiungere il plateau).
Soluzioni:
1) Aumentare la concentrazione del modello.
2) Aumentare il numero di cicli.
3) Aumento di Mg2+ concentrazione.
2.6 Curva di amplificazione grafico lineare plateau cedimento
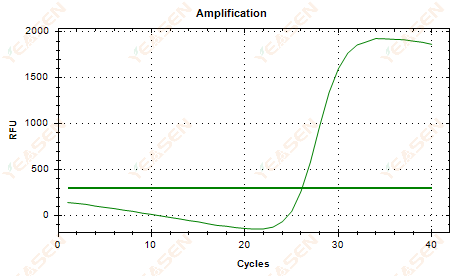
Figura 7. Grafico di amplificazione
Possibili cause:
1) Presenza di degradazione (degradazione del prodotto di amplificazione, degradazione SYBR).
2) Il tappo della provetta non è coperto correttamente e il reagente è volatilizzato.
3) La concentrazione di cDNA è troppo alta (se il valore Ct è troppo piccolo, la soglia di fluorescenza viene alzata e il cedimento è più grave).
4) Bolle d'aria nel tubo, poi scompaiono.
Soluzioni:
1) Migliora la purezza del sistema.
2) Ridurre la quantità di cDNA (diluire il modello).
3) Ridurre il valore finale della linea di base.
2.7 Valore elevato di Ct nella curva di amplificazione
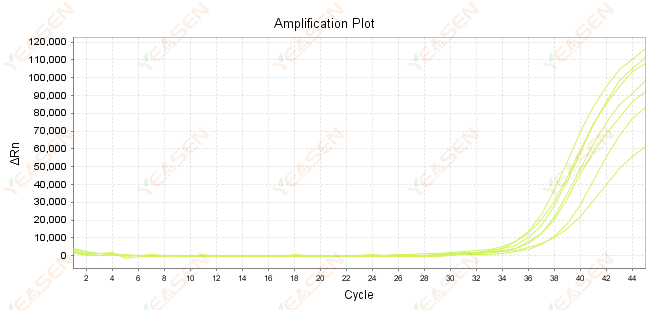
Figura 8. Grafico di amplificazione
Possibili cause:
1) Bassa quantità di modelli.
2) Bassa efficienza di amplificazione.
3) Il frammento PCR è troppo lungo.
4) Presenza di inibitori nel sistema di reazione.
Soluzioni:
1) Ridurre i tempi di diluizione o aumentare la quantità di modello in modo che il valore Ct sia il più possibile compreso tra 15 e 30.
2) Ottimizzare le condizioni di reazione, provare una procedura di amplificazione in tre fasi o riprogettare i primer.
3) La lunghezza del prodotto PCR è progettata per essere compresa tra 100 e 150 bp; non è consigliabile superare i 300 bp.
4) Riproduzione di modelli di purezza più elevata.
2.8 Scarsa ripetibilità delle curve di amplificazione
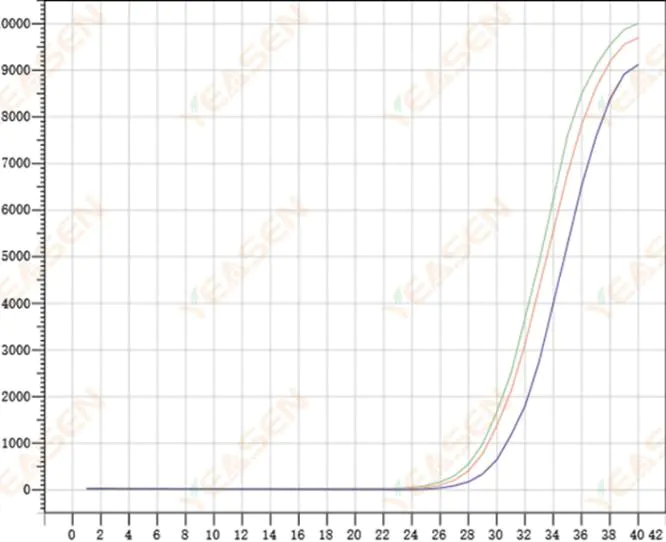
Figura 9. Grafico di amplificazione
Possibili cause:
1) Grandi errori di picco.
2) I reagenti e i sistemi non sono ben miscelati.
3) Basso numero di copie di cDNA.
4) Non è stata utilizzata alcuna calibrazione Rox.
Soluzioni:
1) Pipette di calibrazione.
2) Miscelazione accurata del sistema di reazione.
3) Una bassa concentrazione del modello, una scarsa ripetibilità, 4-6 fori composti e 1-2 deviazioni di valori maggiori possono essere opportunamente scartati.
4) È meglio usare la calibrazione Rox. Se il reagente usato non contiene Rox, il colorante di riferimento deve essere selezionato come None.
2.9 Curve di amplificazione casuali
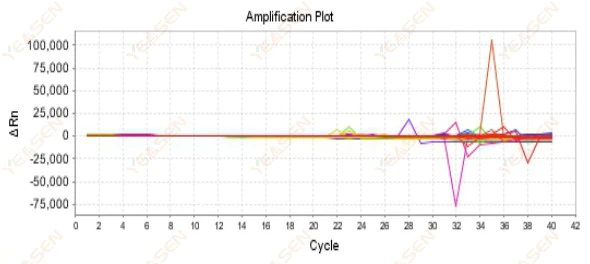
Figura 10. Grafico di amplificazione
Possibili cause:
La discrepanza tra la concentrazione di Rox e il modello.
Soluzioni:
Modificare l'impostazione del colorante di riferimento sullo strumento da ROX a NESSUNO e verificare se la curva di amplificazione torna alla normalità.
2.10 Picco NTC
2.10.1 Ct>35, curva di fusione valore Tm <80℃
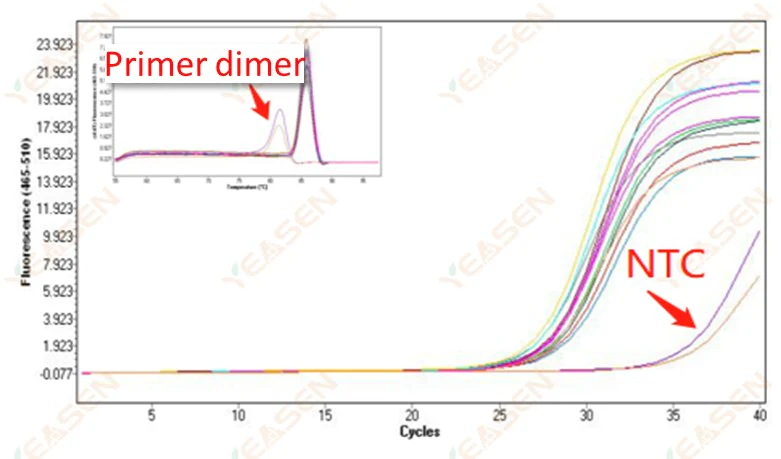
Figura 11. Grafico di amplificazione
Possibili cause:
Risultati della dimerizzazione del primer.
Soluzioni:
Primer ottimizzati.
2.10.2 Valore Ct <35, la curva di fusione NTC e la forma del picco della curva di fusione genica si sovrappongono
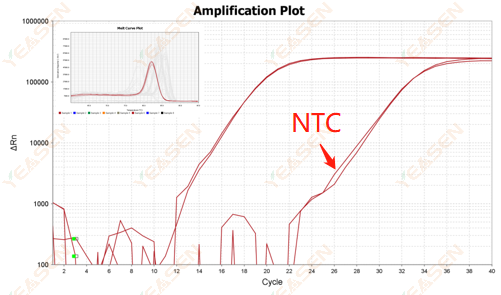
Figura 12. Grafico di amplificazione
Possibili cause:
Contaminazione del sistema di reazione.
Soluzioni:
Controllare una per una le fonti di inquinamento.
3. Curva di fusione qPCR anomala
3.1 Curva di fusione a picco singolo ma non netta
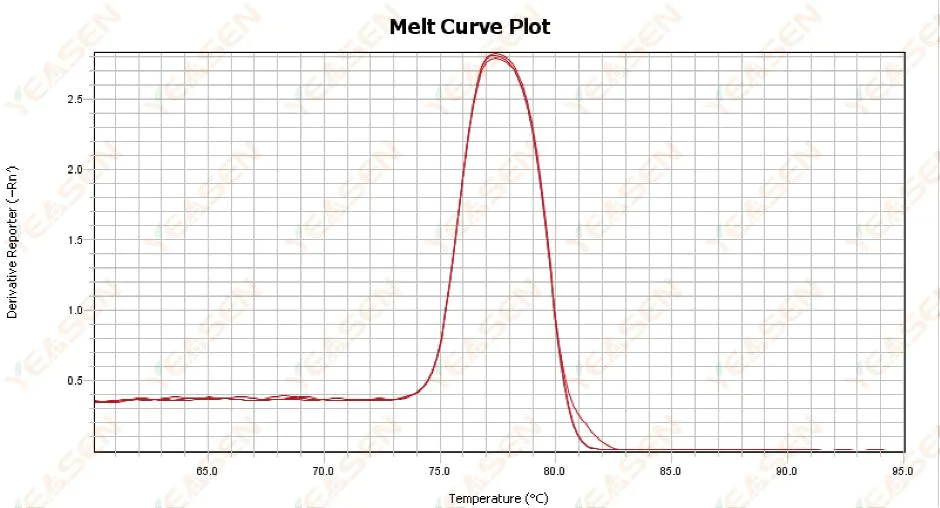
Figura 13. Grafico della curva di fusione
Possibili cause:
1) È correlato alla composizione del reagente e al modello dello strumento.
2) Presenza di amplificazione aspecifica di frammenti di dimensioni simili.
Soluzioni:
1) L'intervallo di temperatura dall'inizio del picco al picco di fine non è superiore a 7°C. I risultati sono considerati utilizzabili, ovvero picco singolo.
2) Per facilitare la determinazione è stata eseguita un'elettroforesi dell'agarosio ad alta concentrazione (ad esempio agarosio al 3%).
3.2 La curva di fusione è a doppio picco e il picco inferiore Tm è prima di 80°C
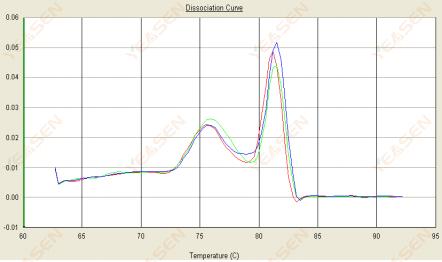
Figura 14. Curva di dissociazione
Possibili cause:
Presenza di dimeri di primer.
Soluzioni:
Aumentare la temperatura di ricottura, diminuire la concentrazione del primer o riprogettare i primer.
3.3 La curva di fusione è a doppio picco e il picco inferiore Tm è dopo 80°C
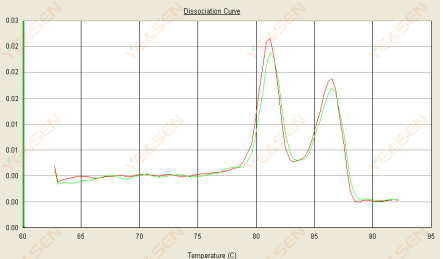
Figura 15. Curva di dissociazione
Possibili cause:
1) Una scarsa specificità del primer porta all'amplificazione di prodotti non specifici.
2) Contaminazione del gDNA.
Soluzioni:
1) Controllare la specificità del primer e riprogettarlo se scadente.
2) La conferma da parte del controllo negativo NRC, se presente, deve essere effettuata per preparare nuovamente il modello.
3.4 Picchi della curva di fusione disordinati
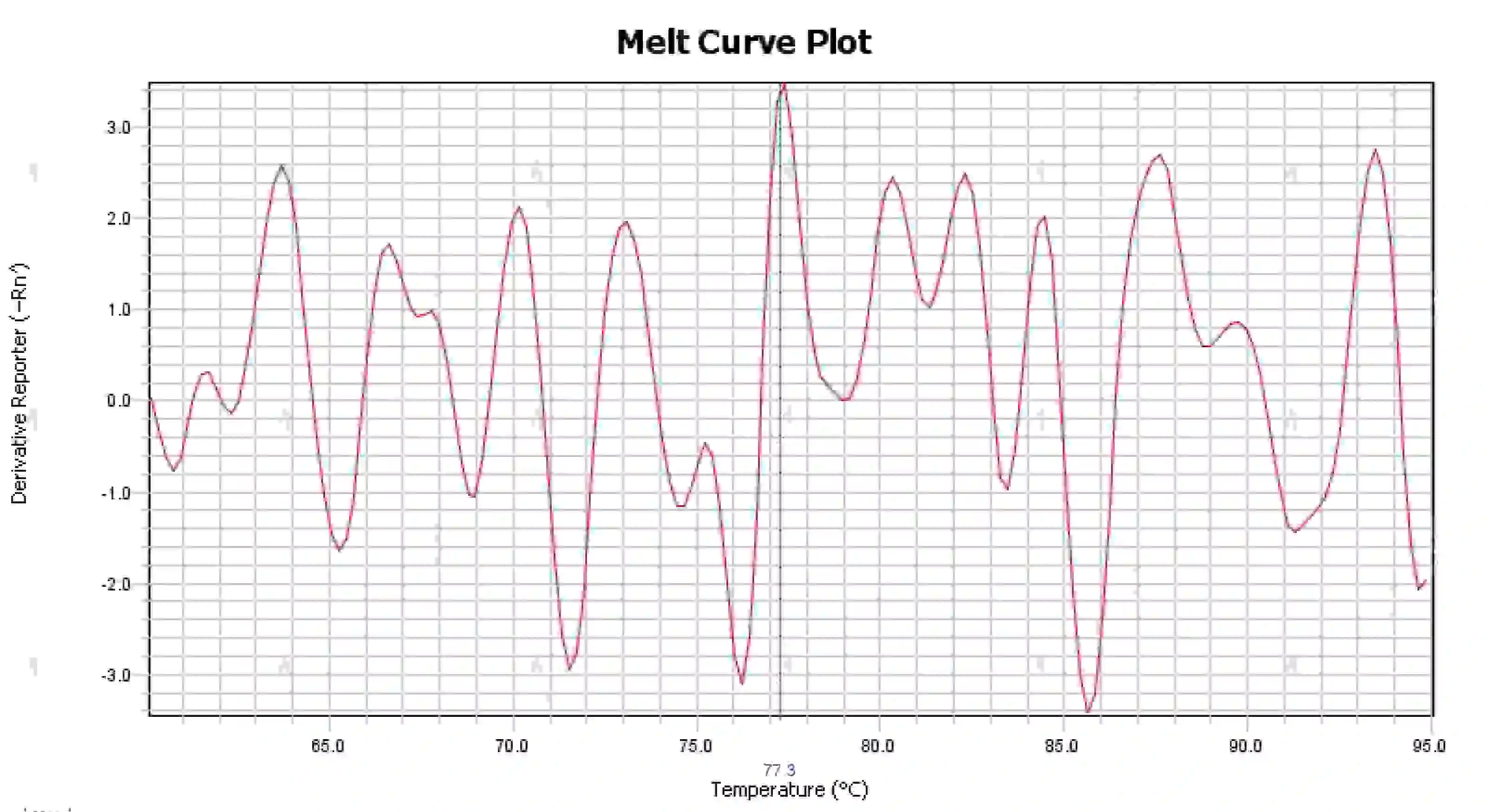
Figura 16. Grafico della curva di fusione
Possibili cause:
1) Contaminazione del sistema di reazione.
2) L'esposizione dei reagenti alla luce intensa o alle alte temperature ne provoca il malfunzionamento.
3) Lo strumento non è stato calibrato per molto tempo.
4) Mancata corrispondenza tra materiali di consumo e strumenti.
Soluzioni:
1) Combinando i risultati NTC e NRC per confermare la contaminazione, si consiglia di escludere una alla volta la contaminazione da acqua, primer, enzimi e ambiente.
2) Si consiglia di effettuare esperimenti di confronto con nuovi reagenti.
3) Si raccomanda di effettuare una regolare manutenzione della calibrazione dello strumento.
4) Confermare i requisiti dello strumento corrispondente per i materiali di consumo.
3.5 Picchi spuri all'estremità anteriore della curva di fusione
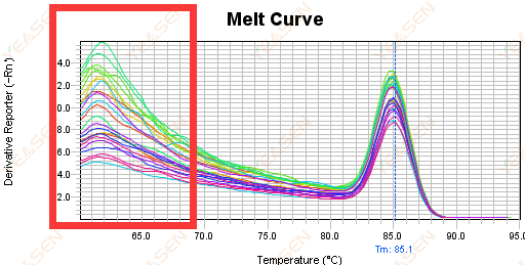
Figura 17. Grafico della curva di fusione
Possibili cause:
La concentrazione di Rox non corrisponde al modello.
Soluzioni:
Si consiglia di annullare la correzione Rox per verificare se la curva di fusione è normale.
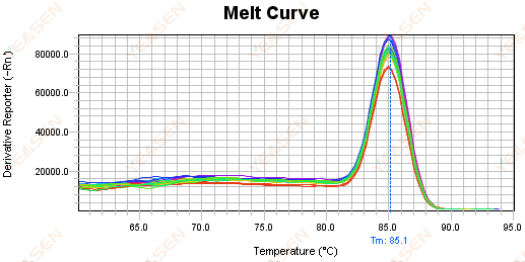
Figura 18. Grafico della curva di fusione
4. Informazioni per l'ordinazione
Tabella 1. Informazioni per l'ordinazione
| Nome del prodotto | Codice prodotto | Specificazione |
| Hieff Unicon™ Universal Blue qPCR Master Mix (a base di colorante) | 11184ES03 | 1 ml. |
| 11184ES08 | 5×1 ml. | |
| 11184ES50 | 50×1 mL | |
| 11184ES60 | 100×1 mL |
5. Prodotti correlati
I prodotti forniti da Yeasen sono i seguenti.
Tabella 2. Prodotti correlati
| Posizionamento del prodotto | Nome del prodotto | Codice prodotto |
| Rimozione del gDNA e trascrizione inversa in un unico passaggio | Digestione RT-gDNA in un unico passaggio Hifair™V SuperMix per qPCR (Richiesta di informazioni) | 11142ES |
| Master Mix ad alta sensibilità (con fase di rimozione del gDNA) | Hifair™ III 1st Strand cDNA Synthesis SuperMix per qPCR (gDNA digester plus) | 11141ES |
| Kit ad alta sensibilità (con fase di rimozione del gDNA) | Kit di sintesi del cDNA a 1° filamento Hifair™ III (digestore gDNA plus) (Richiesta di informazioni) | 11139ES |
| Master mix quantitativo ordinario (metodo sonda) | Hieff™ qPCR TaqMan Probe Master Mix (Richiesta di informazioni) | 11205ES |
6. Per quanto riguarda la lettura
DNasi I e le loro applicazioni in biomedicina
La linea guida per la selezione della trascrittasi inversa
