Come scegliere gli adattatori NGS?
L'adattatore NGS, una parte essenziale della libreria di sequenziamento di nuova generazione, svolge un ruolo nel collegare il frammento di DNA testato e la cella di flusso (chip di sequenziamento). L'efficienza del giunto è un fattore importante nel determinare la qualità e la resa della libreria. Quindi, cos'è un adattatore NGS? Quali sono i tipi comuni di adattatori NGS? E come scegliere gli adattatori NGS giusti per le tue piattaforme di sequenziamento?
1. Che cos'è NGS Adapter?
2. Quali fattori dovresti considerare quando scegli l'indice NGS?
3. Quali sono i tipi più comuni di indici?
4. Quali sono i tipi più comuni di adattatori NGS?
- Adattatore UMI
- Adattatori completi
- Adattatori incompleti
- Adattatori Tn5
5. Come scegliere gli adattatori NGS giusti per le tue piattaforme di sequenziamento?
6. Per quanto riguarda la lettura
1. Che cos'è NGS Adapter?
NGS Adapter, una serie di adattatori nel sequenziamento, è una breve sequenza nucleotidica con una sequenza nota. È legata a entrambe le estremità del frammento di acido nucleico target. Durante il sequenziamento, inizia il sequenziamento ibridandosi con la sequenza nota sulla cella Flow per combinare la libreria al chip. Quindi qual è la struttura dell'adattatore NGS?
Prendendo come esempio la piattaforma Illumina, un adattatore NGS può essere suddiviso in tre parti:
P5 e P7: sequenza combinata con le estremità P5 e P7 sulla cella di flusso per fissare la libreria sul chip di sequenziamento, facilitando la reazione del cluster tramite Bridge-PCR.
Rd1 SP e Rd2 SP (primer di sequenziamento Read1/Read2): regioni di legame dei primer di sequenziamento, che indicano la posizione in cui la sequenza inizia a essere letta.
Indice (noto anche come codice a barre): sequenza sintetica nota utilizzata per distinguere diversi campioni nel sequenziamento della libreria mista.
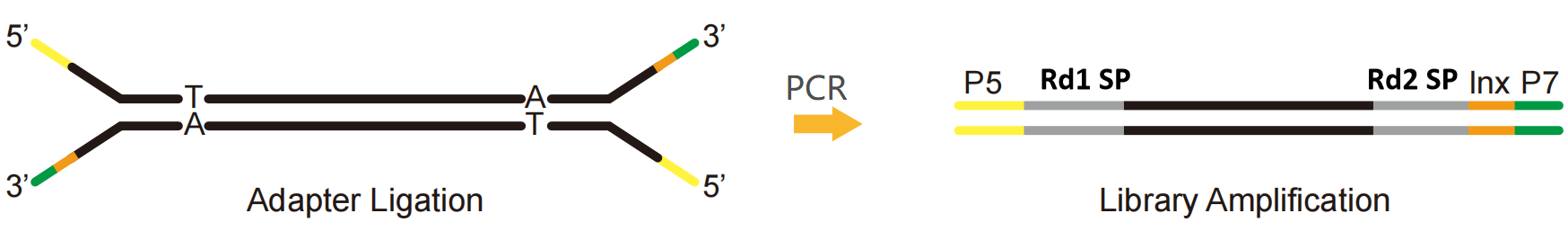
Fico. 1 Libreria di indici singoli della piattaforma Illumina

Fico. 2 Libreria di indici single-end della piattaforma MGI
Con l'aumento della produttività del sequenziamento, è possibile sequenziare più campioni contemporaneamente. Quindi è particolarmente importante distinguere campioni diversi. Come accennato in precedenza, le sequenze indice dell'adattatore NGS vengono utilizzate per differenziare vari campioni nel sequenziamento di nuova generazione (NGS). Quali fattori dovresti considerare quando scegli l'indice? Continua a leggere...
2. Quali fattori dovresti considerare quando scegli un indice?
L'indice è generalmente lungo 6nt-18nt ed è diviso in un indice singolo e un indice doppio in base al numero di indici. L'indice doppio si trova a entrambe le estremità del frammento da testare. Il bilanciamento delle basi e il bilanciamento della fluorescenza devono essere considerati quando si seleziona una combinazione di indici.
Il saldo di base si riferisce al saldo tra più indici, piuttosto che al saldo di base in un singolo indice. Deve essere considerato sia in base ai tipi di base che alla distribuzione di base. Il principio di combinazione è che devono essere incluse le quattro basi A/T/C/G nello stesso gruppo di indici e la proporzione di queste quattro basi è ravvicinata, rappresentando rispettivamente circa il 25%.
Il bilanciamento del segnale di fluorescenza si riferisce alla scelta di garantire il bilanciamento dei segnali fluorescenti quando il bilanciamento di base non può essere garantito. Nel sequencer a 4 canali nella piattaforma Illumina, dG/dT è etichettato con fluorescenza verde e dC/dA è etichettato con fluorescenza rossa. Durante il sequenziamento, per garantire il successo del sequenziamento, in ogni ciclo devono essere presenti sia segnali di fluorescenza verdi che rossi. Pertanto, quando si seleziona l'indice, è opportuno considerare l'equilibrio tra il segnale verde e quello rosso.
3. Quali sono i tipi più comuni di indici?
Gli indici doppi più comuni includono solitamente l'indice doppio univoco (UDI), l'indice doppio univoco (UDB) e l'indice doppio combinato (CDI), che riducono significativamente i salti di indice e le assegnazioni errate.
UDI&UDB: gli indici ad entrambe le estremità sono corrispondenti uno a uno, progettati in gruppi e possono essere verificati in modo incrociato ad entrambe le estremità;
Un kit di primer UDI Stubby per Illumina fornito da Yeasen (Cat#12404ES/12405ES)>>
CDI: gli indici ad entrambe le estremità possono essere combinati secondo determinati requisiti per formare una libreria di indici a doppia estremità;
Un primer 384 CDI per Illumina, Set1-Set2 fornito da Yeasen (Cat#12412ES/12413ES)>>
Per migliorare la produttività e l'efficienza di amplificazione e ridurre i costi di sequenziamento, Illumina ha introdotto la cella a flusso array (PFCT) e la tecnologia di clustering di amplificazione esclusiva (ExAmp) per Novaseq e altri sequenziatori ad alta produttività, amplificando però inavvertitamente il fenomeno di mancata corrispondenza delle etichette dei campioni e il salto di indice.

Fig. 3 I diversi modelli di strumenti Illumina adottano celle a flusso non modellate o celle a flusso modellate
Per compensare il problema dell'index hoop evidenziato dalle piattaforme di sequenziamento come HiSeq3000/4000, HiSeq X Series e NovaSeq, Illumina ha proposto la strategia di posizionare l'indice su entrambe le estremità della libreria, il che può eseguire la verifica bilaterale ed eliminare gli adattatori non corrispondenti. Quando si utilizzano gli indici univoci su entrambe le estremità, il tasso di allocazione degli errori dell'indice verrà ridotto allo 0,01%. Rispetto al precedente metodo di combinazione del gruppo di permutazione dell'indice convenzionale, l'index hopping verrà ridotto di due ordini di grandezza.
Nella costruzione di una libreria PCR-free, è disponibile un adattatore indice single-end. La mancata corrispondenza delle etichette è causata principalmente da errori di sequenziamento. Nel complesso, il tasso di mancata corrispondenza delle etichette è basso (in media 0,0004%, fino a 0,001%). Tuttavia, nella costruzione di una libreria di cattura mirata, il problema di diafonia è amplificato perché più passaggi porteranno alla mancata corrispondenza delle etichette e di solito vengono utilizzati gli adattatori UDI/UDB/CDI.
4.Quali sono i tipi più comuni di adattatori NGS?
Con lo sviluppo della tecnologia di sequenziamento, ci sono sempre più tipi di adattatori, come gli adattatori a indice singolo/doppio indice (come menzionato nella sezione 3), gli adattatori UMI, gli adattatori transposase, gli adattatori completi/incompleti, ecc., che sono adatti a una varietà di scenari applicativi. Questa parte classifica sistematicamente questi adattatori per fornirti le basi della selezione degli adattatori.
Adattatore UMI 4.1
L'adattatore Unique Molecular Identifier (UMI) è uno strumento di punta per il rilevamento di mutazioni a bassa frequenza e la quantificazione assoluta. L'UMI è una sequenza sintetica casuale con una sequenza nota. Può essere progettata come una catena nucleotidica completamente casuale, una catena nucleotidica parzialmente degenerata o una catena nucleotidica fissa.La lunghezza è solitamente 10nt (UMI a terminazione singola) o 5-8nt (UMI a doppia terminazione). La sua funzione è quella di congelare lo stato dei frammenti di DNA prima dell'amplificazione e ogni molecola di DNA corrisponde a un UMI. Pertanto, durante l'analisi della bioinformatica, può distinguere i modelli di DNA da diverse fonti, distinguere quali sono mutazioni falso-positive causate da errori casuali nel processo di amplificazione e sequenziamento PCR e quali sono effettivamente trasportate dai pazienti, in modo da filtrare il rumore di fondo, realizzare il rilevamento accurato di mutazioni a bassa frequenza ed estremamente bassa frequenza ed eseguire la quantificazione assoluta di diverse molecole di DNA. È ampiamente utilizzato nel rilevamento delle mutazioni a bassa frequenza, in particolare nel campo della ricerca sui tumori.

Fig 4 Schema dell'adattatore UMI struttura della piattaforma Illumina
4.2 Adattatori completi
Gli adattatori completi, un prodotto necessario per una libreria PCR-free, contengono tutte le sequenze richieste per i sequenziamenti, come P5, P7, RdS1 e RdS2 nella piattaforma Illumina, inoltre indicizzano le sequenze e le sequenze UMI in base ai requisiti per il sequenziamento. Con gli adattatori completi, può essere sequenziato direttamente senza introdurre altri adattatori tramite PCR. Quindi, gli adattatori completi possono essere utilizzati per creare una libreria PCR-free. Le librerie PCR-free possono ridurre il bias di amplificazione PCR, il tasso di errore e la duplicazione delle sequenze, aumentando la copertura di alcune regioni GC o AT elevate che sono ampiamente utilizzate nella ricerca sul genoma della popolazione.
Un prodotto adattatore completo per la piattaforma Illumina fornito da Yeasen (Cat#13519ES/13520ES)>>
Un prodotto adattatore completo per la piattaforma MGI fornito da Yeasen (Cat#13360ES/13361ES)>>
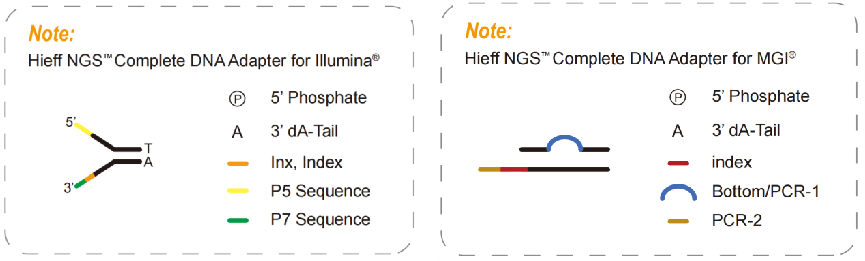
Fig. 5 Schema completo dell'adattatore
4.3 Adattatori incompleti
Gli adattatori incompleti devono introdurre altre sequenze tramite PCR dopo la legatura dell'adattatore per formare un adattatore completo. Sono caratterizzati da elevata efficienza di connessione e alta velocità di libreria effettiva. Il processo PCR è un effetto di arricchimento per la libreria completa per garantire la concentrazione della libreria effettiva e può anche introdurre indici a doppia estremità e sequenze UMI.
4.4 Adattatori Tn5
Gli adattatori Tn5 collegano parte della sequenza adattatrice a entrambe le estremità dei frammenti di DNA tramite l'attività endonucleasica di restrizione di Tn5. Eseguono la frammentazione e la legatura dell'adattatore contemporaneamente per risparmiare tempo e campioni. Infine, il resto della sequenza linker, indice, UMI e altre sequenze vengono introdotte tramite PCR per formare una libreria completa. Può essere utilizzata per creare una libreria Cut&tag.
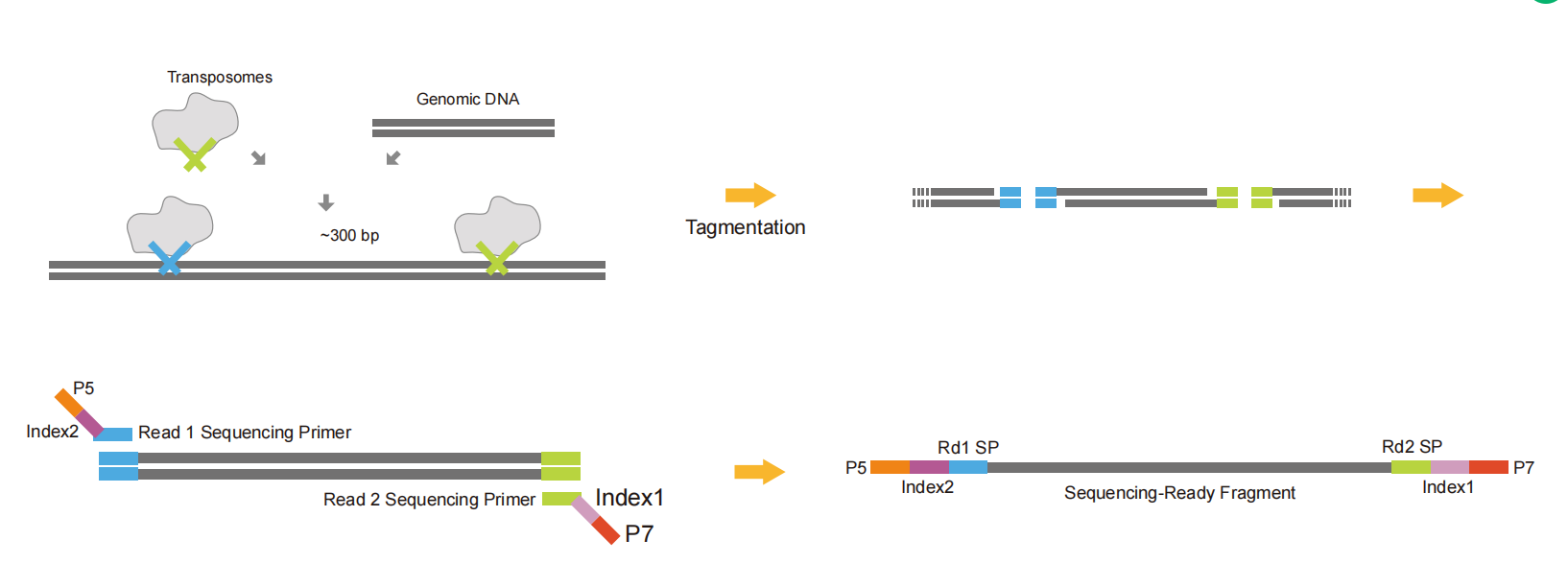
Fig.6 Diagramma schematico della costruzione della libreria dell'adattatore Tn5
5. Come scegliere gli adattatori NGS giusti per le tue piattaforme di sequenziamento?
Attualmente, ci sono due piattaforme di sequenziamento mainstream, tra cui Illumina e MGI. Yeasen, un fornitore di soluzioni complete per NGS, ha sviluppato più adattatori NGS adatti alle piattaforme Illumina o MGI.
In termini di piattaforma Illumina, gli adattatori Illumina NGS forniti da Yeasen contengono tre tipi, tra cui UDI, CDI e un singolo indice. In termini di piattaforma MGI, gli adattatori MGI NGS offerti da Yeasen hanno due tipi, che comprendono Dual UMI-UDB e indice singolo.Nella tabella seguente sono elencate le informazioni sul prodotto, inclusi i tipi di adattatori, le dimensioni disponibili e la concentrazione rispettivamente dell'adattatore e dell'innesco.
Gli adattatori NGS completi e UDI non devono preoccuparsi di problemi di accoppiamento, adatti ai clienti che desiderano semplicità d'uso; gli adattatori NGS CDI hanno meno tubi e dimensioni ridotte, adatti ai clienti che desiderano conservare e trasportare facilmente. PCR-free richiede l'uso di adattatori NGS completi.
Per Illumina
| Nel tubo | Hieff NGS® DPreparazione NA Lib 384 CDI Primer per Illumina, Set 1 (8*12, indice 96) | 12412ES |
| Hieff NGS® DPreparazione NA Lib 384 CDI Primer per Illumina, Set 2 (8*12, indice 96) | 12413ES | |
| Primer Hieff NGS® RNA Lib Prep 384 CDI per Illumina, Set 1 (indice 96) | 12414ES | |
| Primer Hieff NGS® RNA Lib Prep 384 CDI per Illumina, Set 1 (indice 96) | 12415ES | |
| Nel piatto | Kit primer Hieff NGS® Stubby UDI per Illumina (indice 1-384) Set 1-4 | 12407ES |
| Kit primer Hieff NGS® Stubby UDI per Illumina Imposta1(Piastra a 96 pozzetti, indice 1-96) Imposta 1 | 12327ES | |
| Kit primer Hieff NGS® Stubby UDI per Illumina Set2(Piastra a 96 pozzetti, indice 97-192) Imposta 2 | 12328ES | |
| Kit primer Hieff NGS® Stubby UDI per Illumina Set3(Piastra a 96 pozzetti, indice 193-288) Imposta 3 | 12329ES | |
| Kit primer Hieff NGS® Stubby UDI per Illumina Set4(Piastra a 96 pozzetti, indice 289-384) Imposta 4 | 12330ES |
Per MGI
| Capo Tecnologia NGS™ Kit adattatore UMI UDB doppio per MGI, Set1/Set2 | 96 tipi di indice | |
| Sistema NGS™ Hieff Kit adattatore completo per MGI, Set1/Set2/Set3(Richiedi informazioni) | 13360ES | 8 tipi di indice, 41-48 |
| 13361ES | 16 tipi di indice, 57-72 | |
| 13362ES | 96 tipi di indice, 1-96 |
Per quanto riguarda la lettura
Enzimi chiave coinvolti nella costruzione della libreria NGS
Quanto ne sai sulla tecnologia NGS?
Vari tipi di sfere magnetiche in NGS: sfere magnetiche DNA\RNA\mRNA
Quantificazione della libreria NGS: Qubit rapido e accurato o qPCR preciso? Tutto ciò di cui hai bisogno!
