DNase I とバイオメディカルにおける応用
デオキシリボヌクレアーゼ I (DNase I) はエンドヌクレアーゼであり、RNA の完全性を維持するだけでなく、DNA フットプリント分析、ランダム DNA ライブラリの生成、細胞溶解物またはタンパク質抽出物の粘着性の低減などにも使用されます。つまり、DNase I は、DNA の酵素切断を必要とするほぼすべてのアプリケーションで使用できます。以下は、DNase I とその特定のアプリケーションについての詳細な紹介です。
1. DNase I とは何ですか?
2. DNAフリーRNA抽出液の調製のためのDNase I
3. 試験管内転写でテンプレートDNAを除去するためのDNase I
4. rRNA除去のためのDNase I
5. DNA標識用DNase I
6. その他のアプリケーション
7. DNase I 製品選択ガイド
1. DNase I とは何ですか?
デオキシリボヌクレアーゼ I (DNase I)は、さまざまな組織や体液中に存在する一本鎖または二本鎖 DNA を消化できる非特異的エンドヌクレアーゼです。リン酸ジエステル結合を加水分解して、5'-リン酸基と 3'-OH 基を含むモノデオキシヌクレオチドおよびオリゴデオキシヌクレオチドを生成します。DNase I の最適作用 pH 範囲は 7~8 で、その活性は Ca2+ に依存し、Mn2+、Mg2+、Zn2+ などの二価金属イオンによって活性化されます。Mg2+ が存在する場合、DNase I は二本鎖 DNA の任意の部位をランダムに切断します。Mn2+ が存在する場合、DNase I は同じ部位で二本鎖 DNA を切断して、平滑末端または 1~2 ヌクレオチドの粘着末端オーバーハングを形成します。
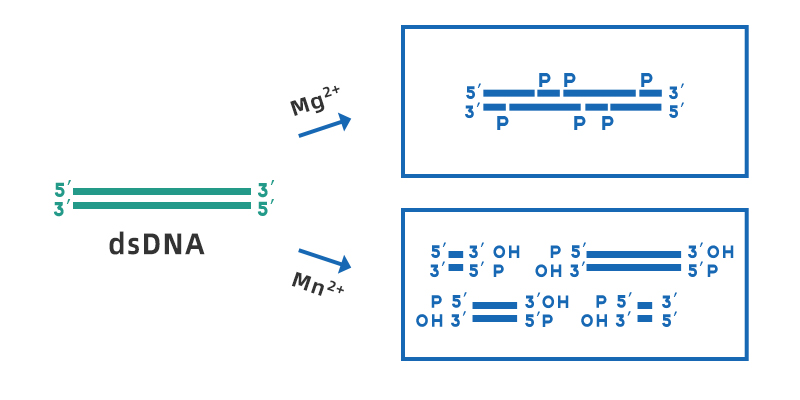
図1. Mg2+およびMn2+存在下でのDNase IによるdsDNAの切断の模式図。
DNase I による切断は一般に非特異的切断と考えられていますが、DNase I は副溝領域などの特定の配列断片に作用する可能性が高く、プリン-ピリミジン配列の切断が起こりやすい傾向があります。ただし、DNase I が異種 dsDNA に作用すると、4 つの塩基すべてが切断され、特定の塩基への影響は他の塩基の影響の 3 倍を超えることはありません。
2. DNAフリーRNA抽出液の調製のためのDNase I
生物学実験では、まず核酸を準備してRNAのさまざまな機能を研究します。しかし、細胞溶解プロセス中にDNAとRNAが一緒に放出されることが多いため、どのような抽出溶液を使用しても両者の干渉を避けることはできません。そのため、干渉を除去するには特定の酵素を使用する必要があります。高品質のRNA抽出には、サンプルから残留DNAを除去するためにDNase Iが使用されます。
DNase I は二本鎖および一本鎖 DNA をオリゴヌクレオチドおよび単一ヌクレオチドに分解することができ、RNA 調製製品中の DNA を効果的に分解することができます。次に、DNase I を停止バッファーで加熱して不活性化します。加熱プロセス中に、RNA 分子のヘアピン構造を開くことができ、これにより RNA が逆転写プロセスに直接入りやすくなります。
RNA の品質は、実験データに直接大きな影響を及ぼします。一般に、RNA 抽出中に gDNA 残留物を完全に回避することはできません。そのため、下流のアプリケーション (mRNA 発現分析、トランスクリプトーム分析など) を実行する前に、RNA サンプルを DNase I で処理して残留 gDNA を消化することが推奨されます。gDNA 消化のステップは、RNA 抽出中、RNA 抽出後、または RNA 逆転写の前に実行できます。製品の位置付けによると、
表1: RNA抽出または逆転写前のDNA除去に関連する製品のリスト
|
製品の位置付け |
製品名 |
猫 # |
|
RNA抽出 |
TRIeasy™ トータルRNA抽出試薬 [お問い合わせ] |
10606ES |
|
19221ES |
||
|
MolPure™ Plant Plus RNAキット[お問い合わせ] |
19292ES |
|
|
MolPure™ ウイルス DNA/RNA キット[お問い合わせ] |
19321ES |
|
|
gDNA除去 |
10325ES |
|
|
逆転写 |
11141ES |
|
|
定量PCR |
11184ES |
3. 試験管内転写でテンプレートDNAを除去するためのDNase I
インビトロ転写(IVT)では、主にDNAをテンプレートとして使用し、対応する基質とバッファーを使用して、インビトロ転写によってRNAを取得します。インビトロ転写実験では、T7、T3、SP6などのRNAポリメラーゼがRNA合成によく使用されます。合成されたRNAにはDNA残基が含まれている場合があります。DNA残基を除去することは、下流の実験の開発に有益です。たとえば、mRNAワクチンの開発段階では、残基の除去が重要なステップであり、下流の精製の難しさを軽減し、製品の純度を高めることができます。DNAテンプレートは通常、組換えDNase I(RNaseフリー)を使用して除去されます。mRNA合成プロセスに応じて、
|
mRNA合成プロセス |
製品名 |
猫# |
|
テンプレートの準備 |
Hieff Canace™ Plus 高忠実度 DNA ポリメラーゼ[お問い合わせ] |
10148ES |
|
10922ES |
||
|
10125ES |
||
|
FuniCut™BsaI [お問い合わせ] |
15005ES |
|
|
FuniCut™ XbaI [お問い合わせ] |
15033ES |
|
|
BspQI[問い合わせ] [問い合わせ] |
16215ES |
|
|
インビトロ転写 |
10623ES |
|
|
10624ES |
||
|
T7 RNAポリメラーゼ(50 U/μL) [お問い合わせ] |
10618ES |
|
|
10133ES |
||
|
10620ES |
||
|
10621ES |
||
|
テンプレートDNAの除去 |
10611ES |
|
|
mRNAの修飾 |
10614ES |
|
|
10612ES |
||
|
10132ES |
||
|
10619ES |
||
|
mRNAの精製 |
12602ES |
4. rRNA除去のためのDNase I
生体内では、rRNA は非常に豊富で非常に保存的であり、生物学的情報を取得する上であまり重要ではないため、RNA ライブラリの構築と配列決定では、rRNA が最初に除去されることがよくあります。現在、rRNA の除去方法は主に RNase H 消化です。酵素ベースの rRNA 除去の主な手順を図 2 に示します。 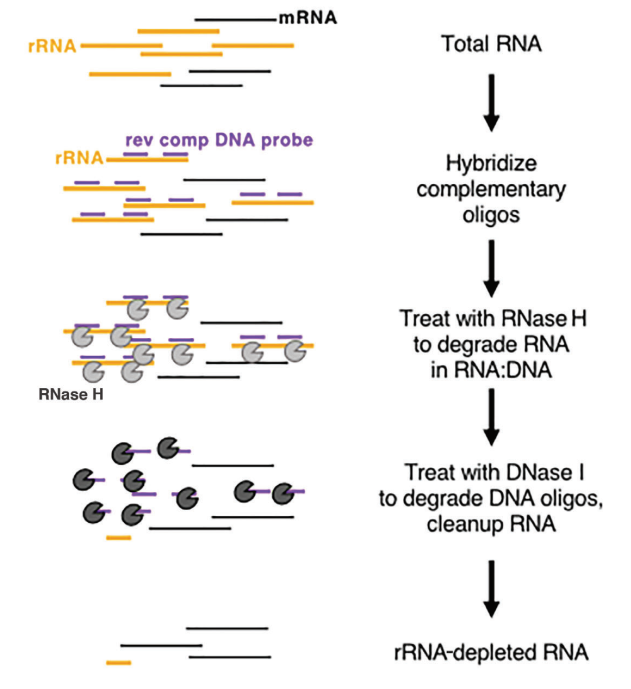
図2: 酵素ベースのrRNA除去の原理の模式図 (Baldwin, A. et al. 2021, Current Protocols)
まず、全RNAを抽出し、一本鎖DNAプローブをrRNAとハイブリダイズし、rRNA固有の一本鎖DNAプローブを設計および合成し、RNase Hを使用してハイブリダイズしたrRNAを分解し、DNase Iを使用してDNAプローブを分解します。最後に、非rRNA RNAテンプレートを残します。
|
mRNA合成プロセス |
製品名 |
猫# |
|
ヒト/マウス/ラット rRNAの枯渇 |
Hieff NGS™ MaxUp rRNA除去キット(ヒト/マウス/ラット)MaxUp [お問い合わせ] |
12253ES |
|
植物rRNAの枯渇 |
12254ES |
|
|
ヒト全RNAからのリボソームRNAおよび45S ITS/ETS領域の除去 |
12257ES |
|
|
rRNAの分解 |
12906ES |
|
|
DNAプローブの分解 |
10325ES |
5.DNA標識用DNase I
ニックトランスレーションは、研究室で最も一般的に使用されているデオキシリボ核酸プローブ標識法の 1 つです。この方法では、DNA ポリメラーゼ I のさまざまな酵素活性を利用して、標識されたデオキシリボヌクレオシド三リン酸を新しく合成された DNA 鎖に組み込みます。このようにして、高い特異的活性を持つ均一に標識された DNA プローブが合成されます。ニックトランスレーションの特徴は、高速、シンプル、慎重、高特異性、均一に標識されたプローブであり、長い二本鎖 DNA に適しています。この方法は、DNase I と E. coli DNA ポリメラーゼ I の複合作用によって実現されます。ニックトランスレーションによる DNA 標識の主な手順を図 3 に示します。
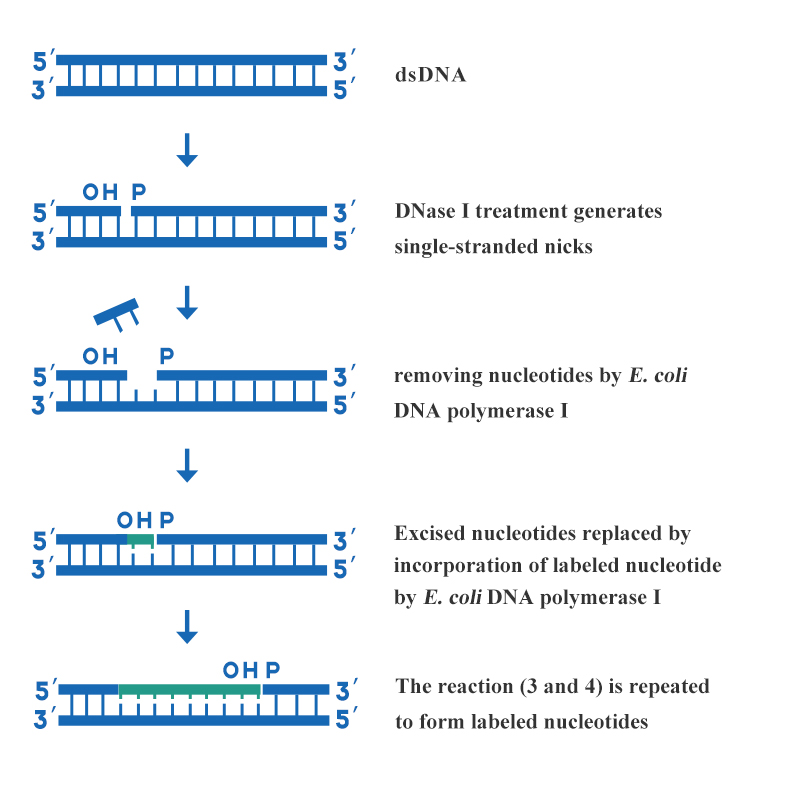
図3: ニックトランスレーションによるDNA標識の模式図
適切な濃度の DNase I を使用して、ラベル付けする二本鎖 DNA の各鎖に複数の一本鎖ギャップを作成し、ギャップに 3' ヒドロキシル末端を形成します。E. coli DNA ポリメラーゼ I の 5'→3' エキソヌクレアーゼ活性を使用して、ニックの 5' 末端からヌクレオチドを切断し、同時に E. coli DNA ポリメラーゼ I の 5'→3' ポリメラーゼ活性を使用して、ギャップの 3' 末端でラベル付けされたヌクレオチドを導入してギャップを修復します。ギャップが DNA 鎖に沿って移動すると、ラベル付けされたヌクレオチドが新しく合成された鎖に組み込まれます。
|
製品の位置付け |
製品名 |
猫# |
|
普通 |
牛膵臓由来デオキシリボヌクレアーゼI(DNase I) [問い合わせ] |
10607ES/10608ES |
|
RNaseフリー |
10325ES |
|
|
大腸菌源 |
12903ES |
6. その他のアプリケーション
上記は、よく使用されるいくつかのアプリケーションです。DNase I のその他のアプリケーションには、DNase I フットプリント アッセイや DNase I 過敏性部位などがあります。DNase I フットプリント アッセイは、DNA 上の DNA 結合タンパク質の結合部位を正確に特定できる検出方法です。タンパク質が DNA フラグメントに結合すると、結合部位が DNase I によって損傷されるのを防ぐことができ、DNA フラグメントは酵素消化後に残り (「フットプリント」)、その配列を決定できます。ゲル画像には、DNA がタンパク質に結合するバンドはありません。詳細を読むには、リンクをクリックしてください。DNase I 過敏性部位とは、クロマチンを低濃度の DNase I で処理したときに少数の特定部位で生じる切断を指し、これらの特定部位は DNase I 過敏性部位と呼ばれます。原理は、遺伝子が転写活性状態にある場合、遺伝子を含むクロマチンは不活性領域よりも DNase 分解に対して著しく敏感であるということです。詳細を読むには、リンクをクリックしてください。
7. DNase I 製品選択ガイド
2014年に設立された|
製品名(カタログ番号) |
製品の位置付け |
推奨アプリケーション |
|
牛膵臓由来DNase I(CAT#10607、10608) [お問い合わせ] |
RNase 除去、検出されず |
主にタンパク質研究に使用されます: タンパク質調製物からの DNA 除去。 |
|
組み換え DNase I (RNase フリー) (CAT#10325) |
RNaseフリー、研究用 |
さまざまな用途に最適です: RNase 感受性 cDNA ライブラリなどの RNA およびタンパク質調製物からの DNA 除去、または RT-PCR 実験用のサンプル調製。 |
|
UCF.ME™デオキシリボヌクレアーゼ I (DNase I) GMP グレード(CAT#10611) |
RNaseフリー、GMP医薬品グレード。 |
さまざまな用途に最適です: RNase 感受性 cDNA ライブラリなどの RNA およびタンパク質調製物からの DNA 除去、または RT-PCR 実験用のサンプル調製。 |
読書に関して:
mRNA in vitro合成用GMPグレード試薬
DNase I フットプリント法の原理とその生物医学的応用
参考文献
1. Baldwin A、Morris AR、Mukherjee N. RNA-seq用のヒトリボソームRNAを除去するための簡単で費用対効果が高くスケーラブルな方法[J]。Current Protocols、2021年。
2. Song C、Zhang S、Huang H. 微生物ゲノムの複製起点の同定に適した方法の選択[J]。Frontiers in Microbiology、2015、6:1049。
