Como escolher seus adaptadores NGS?
O NGS Adapter, uma parte essencial da biblioteca de sequenciamento de próxima geração, desempenha um papel na conexão do fragmento de DNA testado e da célula de fluxo (chip de sequenciamento). A eficiência da junta é um fator importante na determinação da qualidade e do rendimento da biblioteca. Então, o que é um adaptador NGS? Quais são os tipos comuns de adaptadores NGS? E como escolher os adaptadores NGS certos para suas plataformas de sequenciamento?
1. O que é o Adaptador NGS?
2. Quais fatores você deve considerar ao escolher o índice NGS?
3. Quais são os tipos comuns de índices?
4. Quais são os tipos comuns de adaptadores NGS?
- Adaptador UMI
- Adaptadores completos
- Adaptadores incompletos
- Adaptadores Tn5
5. Como escolher os adaptadores NGS certos para suas plataformas de sequenciamento?
6. Sobre a leitura
1. O que é o adaptador NGS?
O NGS Adapter, uma série de adaptadores em sequenciamento, é uma sequência curta de nucleotídeos com uma sequência conhecida. Ele é ligado a ambas as extremidades do fragmento de ácido nucleico alvo. Durante o sequenciamento, ele inicia o sequenciamento hibridizando com a sequência conhecida na célula de fluxo para combinar a biblioteca ao chip. Então, qual é a estrutura do NGS adapter?
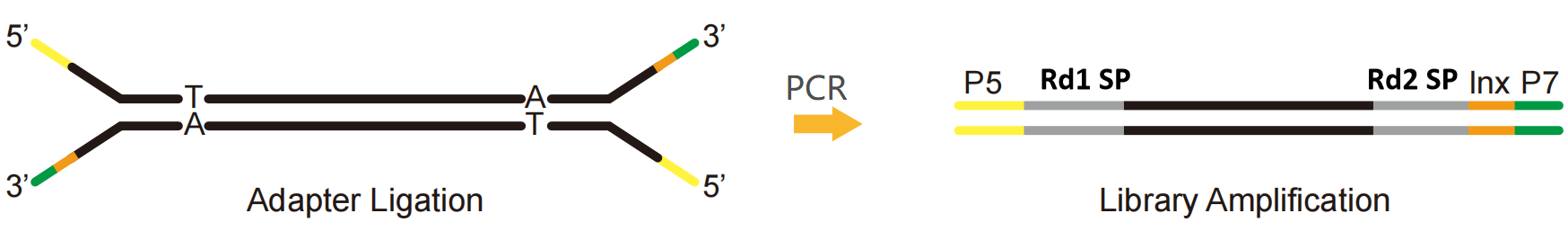
Tomando a plataforma Illumina como exemplo, um adaptador NGS pode ser dividido em três partes:
P5 e P7: Sequência combinada com as extremidades P5 e P7 na célula de fluxo para fixar a biblioteca no chip de sequenciamento, facilitando a reação do cluster por meio de Bridge-PCR.
Rd1 SP e Rd2 SP (primer de sequenciamento Read1/Read2): Regiões de ligação de primers de sequenciamento, indicando a posição onde a sequência começa a ser lida.
Índice (também conhecido como código de barras): uma sequência sintética conhecida usada para distinguir diferentes amostras no sequenciamento da biblioteca mista.
Figo. 1 Biblioteca de índice único da plataforma Illumina

Figo. 2 Biblioteca de índice single-end da plataforma MGI
Com o aumento no rendimento do sequenciamento, várias amostras podem ser sequenciadas ao mesmo tempo. Então, como distinguir amostras diversas é especialmente importante. Como mencionado antes, as sequências de índice do adaptador NGS são usadas para diferenciar várias amostras no sequenciamento de próxima geração (NGS). Quais fatores você deve considerar ao escolher o índice? Por favor, continue lendo...
2. Quais fatores você deve considerar ao escolher o índice?
O índice geralmente tem o comprimento de 6nt-18nt e é dividido em um índice único e um índice duplo de acordo com o número de índices. O índice duplo está localizado em ambas as extremidades do fragmento a ser testado. O equilíbrio de base e o equilíbrio de fluorescência devem ser considerados ao selecionar uma combinação de índice.
O saldo base se refere ao saldo entre múltiplos índices, em vez do saldo base em um único índice. Ele precisa ser considerado tanto dos tipos de base quanto da distribuição de base. O princípio da combinação é que as quatro bases A/T/C/G no mesmo grupo de índices precisam ser incluídas, e a proporção dessas quatro bases é próxima, respondendo por cerca de 25% respectivamente.
O equilíbrio do sinal de fluorescência refere-se à escolha de garantir o equilíbrio dos sinais fluorescentes quando o equilíbrio da base não pode ser garantido. No sequenciador de 4 canais na plataforma Illumina, dG/dT é rotulado com fluorescência verde, e dC/dA é rotulado com fluorescência vermelha. Durante o sequenciamento, sinais de fluorescência verde e vermelha devem existir em cada ciclo para garantir o sequenciamento bem-sucedido. Portanto, o equilíbrio entre o sinal verde e o sinal vermelho deve ser considerado ao selecionar o índice.
3. Quais são os tipos comuns de índices?
Os índices duais comuns geralmente incluem Índice Duplo Único (UDI), Código de Barras Duplo Único (UDB) e Índice Duplo Combinado (CDI), que reduzem significativamente o salto de índice e a atribuição incorreta.
UDI e UDB: os índices em ambas as extremidades são correspondentes um a um, projetados em grupos e podem ser verificados em ambas as extremidades;
Um kit de primer UDI Stubby para Illumina fornecido pela
CDI: os índices em ambas as extremidades podem ser combinados de acordo com certos requisitos para formar uma biblioteca de índices de extremidade dupla;
Um primer 384 CDI para Illumina, Set1-Set2 fornecido pela
Para melhorar a eficiência de rendimento e amplificação e reduzir o custo de sequenciamento, a Illumina introduziu a célula de fluxo de matriz (PFCT) e a tecnologia de agrupamento de amplificação exclusiva (ExAmp) para Novaseq e outros sequenciadores de alto rendimento, mas inadvertidamente amplificou o fenômeno de incompatibilidade de rótulo de amostra e salto de índice.

Fig. 3 Os diferentes modelos de instrumentos da Illumina adotam células de fluxo não padronizadas ou células de fluxo padronizadas
Para compensar o problema de index hooping destacado pelas plataformas de sequenciamento como HiSeq3000/4000, HiSeq X Series e NovaSeq, a Illumina propôs a estratégia de colocar o índice em ambas as extremidades da biblioteca, o que pode executar a verificação bilateral e eliminar os adaptadores incompatíveis. Ao usar os índices exclusivos em ambas as extremidades, a taxa de alocação de erro de índice será reduzida para 0,01%. Comparado com o método de combinação de grupo de permutação de índice convencional anterior, o index hopping será reduzido em duas ordens de magnitude.
Na construção de uma biblioteca livre de PCR, um adaptador de índice de extremidade única está disponível. A incompatibilidade de rótulo é causada principalmente por erros de sequenciamento. No geral, a taxa de incompatibilidade de rótulo é baixa (média de 0,0004%, até 0,001%). No entanto, na construção de uma biblioteca de captura direcionada, o problema de diafonia é amplificado porque várias etapas levarão à incompatibilidade de rótulo e os adaptadores UDI/UDB/CDI são geralmente usados.
4. Quais são os tipos comuns de adaptadores NGS?
Com o desenvolvimento da tecnologia de sequenciamento, há cada vez mais tipos de adaptadores, como adaptadores de índice único/índice duplo (como mencionado na seção 3), adaptadores UMI, adaptadores transposase, adaptadores completos/incompletos, etc., que são adequados para uma variedade de cenários de aplicação. Esta parte classifica sistematicamente esses adaptadores para fornecer a você a base da seleção do adaptador.
4.1 Adaptador UMI
O adaptador Unique Molecular Identifier (UMI) é uma ferramenta de ponta para detecção de mutação de baixa frequência e quantificação absoluta. O UMI é uma sequência sintética aleatória com uma sequência conhecida. Ele pode ser projetado como uma cadeia de nucleotídeos completamente aleatória, cadeia de nucleotídeos parcialmente degenerada ou cadeia de nucleotídeos fixa.O comprimento é geralmente 10nt (UMI de extremidade única) ou 5-8nt (UMI de extremidade dupla). Sua função é congelar o estado dos fragmentos de DNA antes da amplificação, e cada molécula de DNA corresponde a um UMI. Portanto, durante a análise da bioinformática, ele pode distinguir modelos de DNA de diferentes fontes, distinguir quais são mutações falso-positivas causadas por erros aleatórios no processo de amplificação e sequenciamento de PCR e quais são realmente transportadas por pacientes, de modo a filtrar o ruído de fundo, realizar a detecção precisa de mutações de baixa frequência e extremamente baixa frequência e realizar a quantificação absoluta de diferentes moléculas de DNA. É amplamente utilizado na detecção de mutações de baixa frequência, especialmente no campo da pesquisa de tumores.

Fig 4 Diagrama esquemático do adaptador UMI estrutura da plataforma Illumina
4.2 Adaptadores completos
Adaptadores completos, um produto necessário para uma biblioteca livre de PCR, contêm todas as sequências necessárias para sequenciamentos, como P5, P7, RdS1 e RdS2 na plataforma Illumina, também indexam sequências e sequências UMI de acordo com os requisitos para sequenciamento. Com os adaptadores completos, ele pode ser sequenciado diretamente sem introduzir outros adaptadores por meio de PCR. Portanto, adaptadores completos podem ser usados para construir uma biblioteca livre de PCR. As bibliotecas livres de PCR podem reduzir o viés de amplificação de PCR, a taxa de erro e a duplicação de sequência, aumentando a cobertura de algumas regiões de GC alto ou AT alto que são amplamente usadas na pesquisa do genoma populacional.
Um produto adaptador completo para a plataforma Illumina fornecido pela
Um produto adaptador completo para a plataforma MGI fornecido pela
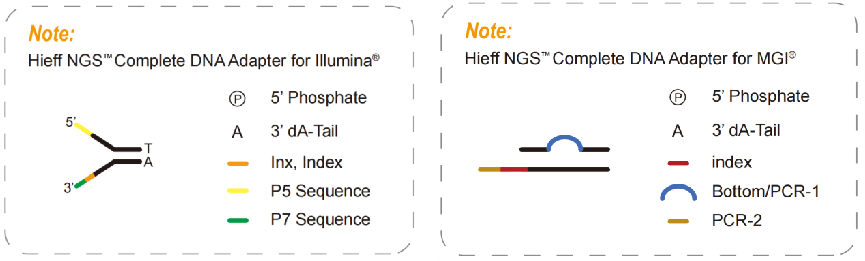
Fig. 5 Diagrama completo do adaptador
4.3 Adaptadores incompletos
Adaptadores incompletos precisam introduzir outras sequências por PCR após a ligação do adaptador para formar um adaptador completo. Eles são caracterizados por alta eficiência de conexão e alta taxa de biblioteca efetiva. O processo de PCR é um efeito de enriquecimento para a biblioteca completa para garantir a concentração da biblioteca efetiva, e também pode introduzir índices de dupla extremidade e sequências UMI.
4.4 Adaptadores Tn5
Os adaptadores Tn5 conectam parte da sequência do adaptador a ambas as extremidades dos fragmentos de DNA por meio da atividade de endonuclease de restrição do Tn5. Eles fazem a fragmentação e a ligação do adaptador realizadas simultaneamente para economizar tempo e amostras. Finalmente, o restante da sequência do linker, índice, UMI e outras sequências são introduzidas por PCR para formar uma biblioteca completa. Ela pode ser usada para construir uma biblioteca Cut&tag.
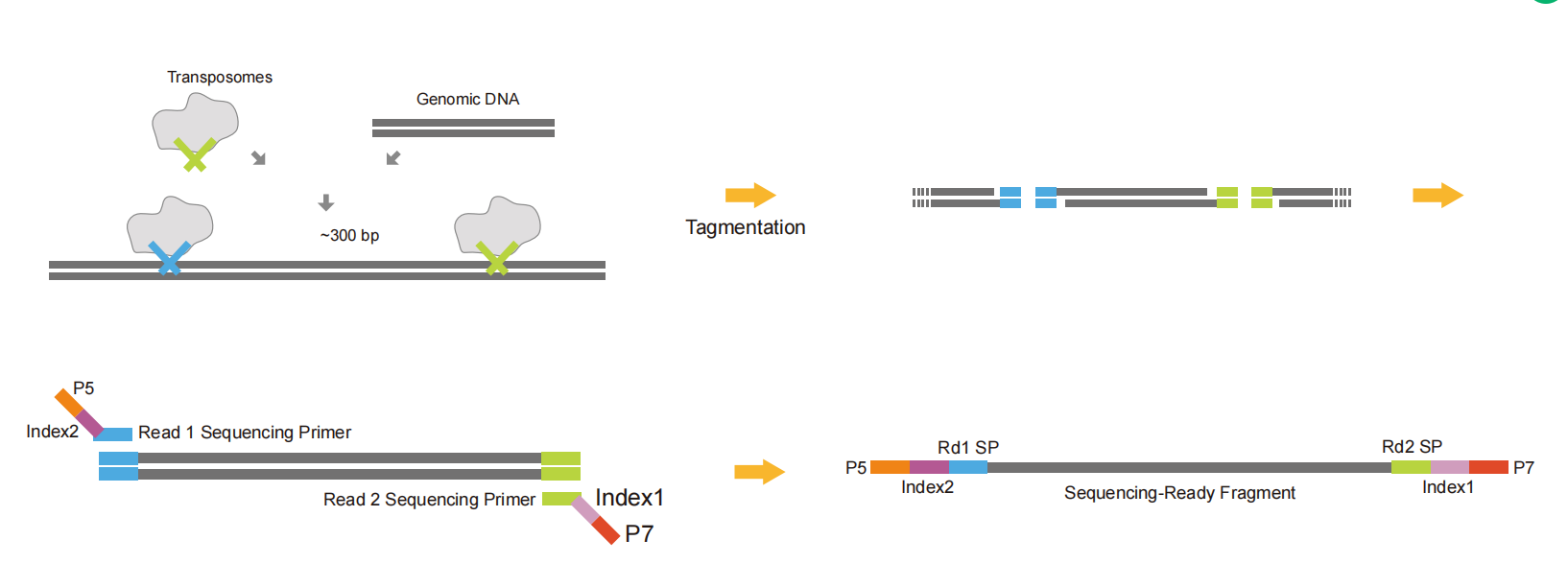
Fig.6 Diagrama esquemático da construção da biblioteca do adaptador Tn5
5. Como escolher os adaptadores NGS certos para suas plataformas de sequenciamento?
Atualmente, há duas plataformas de sequenciamento tradicionais, incluindo Illumina e MGI. A
Em termos da plataforma Illumina, os adaptadores Illumina NGS fornecidos pela
Adaptadores NGS completos e UDI não precisam se preocupar com problemas de acoplamento, adequados para clientes que desejam facilidade de uso; adaptadores NGS CDI têm menos tubos e tamanho pequeno, o que é adequado para clientes que desejam armazenar e transportar facilmente. PCR-free requer o uso de adaptadores NGS completos.
Para Illumina
| No tubo | Hieff NGS® EPreparação para a Biblioteca NA 384 CDI Primer para Illumina, Conjunto 1 (8*12, índice 96) | 12412ES |
| Hieff NGS® EPreparação para a Biblioteca NA 384 CDI Primer para Illumina, Conjunto 2 (8*12, índice 96) | 12413ES | |
| Hieff NGS® RNA Lib Prep 384 CDI Primer para Illumina, Conjunto 1 (índice 96) | 12414ES | |
| Hieff NGS® RNA Lib Prep 384 CDI Primer para Illumina, Conjunto 1 (índice 96) | 12415ES | |
| Em Prato | Kit de primer Hieff NGS® Stubby UDI para Illumina (índice 1-384) Conjunto 1-4 | 12407ES |
| Kit de primer Hieff NGS® Stubby UDI para Illumina Conjunto 1(Placa de 96 poços, índice 1-96) Conjunto 1 | 12327ES | |
| Kit de primer Hieff NGS® Stubby UDI para Illumina Conjunto 2(Placa de 96 poços, índice 97-192) Conjunto 2 | 12328ES | |
| Kit de primer Hieff NGS® Stubby UDI para Illumina Conjunto 3(Placa de 96 poços, índice 193-288) Conjunto 3 | 12329ES | |
| Kit de primer Hieff NGS® Stubby UDI para Illumina Conjunto 4(Placa de 96 poços, índice 289-384) Conjunto 4 | 12330ES |
Para MGI
| Hieff NGS™ Kit adaptador UMI UDB duplo para MGI, conjunto 1/conjunto 2 | 96 tipos de índice | |
| Hieff NGS™ Kit adaptador completo para MGI, Conjunto1/Conjunto2/Conjunto3 (Consultar) | 13360ES | 8 tipos de índice, 41-48 |
| 13361ES | 16 tipos de índice, 57-72 | |
| 13362ES | 96 tipos de índice, 1-96 |
Sobre a leitura
Principais enzimas envolvidas na construção da biblioteca NGS
O quanto você sabe sobre tecnologia relacionada a NGS?
Vários tipos de esferas magnéticas em NGS: esferas magnéticas de DNA\RNA\mRNA
Quantificação da biblioteca NGS: Qubit rápido e preciso ou qPCR preciso? Tudo necessário!
