Dyb indlæring af kerneenzymråmaterialerne, der bruges i PCR-påvisning af SARS-CoV-2
Siden marts 2022 brød den snedige Omicron-mutantstamme igen folks fredelige liv, de nye coronavirus-epidemier brød ud over hele landet og ramte 30 provinser (autonome regioner og kommuner). Som et effektivt middel til præcis forebyggelse og kontrol af SARS-CoV-2-epidemien er nukleinsyrepåvisning blevet en normal livsstil. "Har du lavet nukleinsyretesten i dag?" Det er også blevet en daglig hilsen af folk. Apropos det, ved du, hvilke kerneråmaterialer der er nødvendige i nukleinsyredetektion? Denne artikel vil introducere det kritiske kerneråmateriale i nukleinsyredetektionsenzym.
1. Nukleinsyredetektionsproces for SARS-CoV-2
2. Kerneenzymer i nukleinsyreekstraktion
3. Kerneenzymer under RT-qPCR
4. Kerneenzymer af SARS-CoV-2 nukleinsyrepåvisning fra Yeasen
1. Nukleinsyredetektionsproces for SARS-CoV-2
Et enzym er en ekstremt vigtig klasse af biokatalysatorer med høj katalytisk effektivitet og reaktionsspecificitet. De fleste biokemiske reaktioner kræver deltagelse af enzymer. I processen med nukleinsyredetektion af 2019-nCoV (vist i figur 1) spiller forskellige typer molekylære enzymer en vigtig rolle i forskellige eksperimentelle stadier såsom nukleinsyreekstraktion og RT-qPCR. Dernæst vil vi i henhold til de forskellige eksperimentelle led i nukleinsyredetektion sortere de kerneenzymråmaterialer, der bruges i nukleinsyredetektionsprocessen.
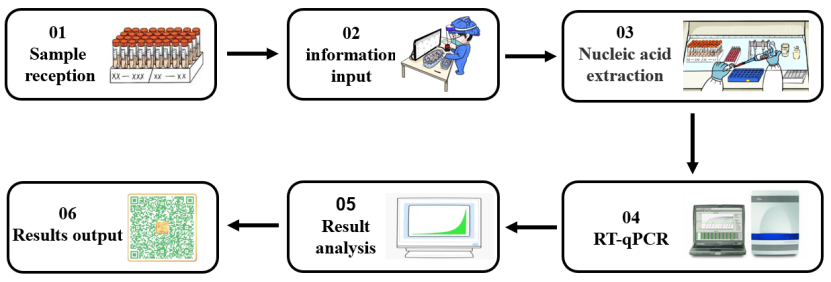
Figur 1. Nukleinsyredetektionsproces for SARS-CoV-2
2. Kerneenzymer i nukleinsyreekstraktion
Den nye coronavirus-nukleinsyreekstraktionsproces omfatter hovedsageligt to trin: lysering og oprensning. Lysis er processen med at ødelægge prøvens cellestruktur, så nukleinsyren i prøven er fri i lysissystemet; Oprensning er den fuldstændige adskillelse af nukleinsyre fra andre komponenter i lysissystemet, såsom protein, salt og andre urenheder, og reaktionsprocessen kræver deltagelse af proteinase K, deoxyribonuklease I og RNase-hæmmere.
2.1 Protease K
Proteinase K er en serinprotease med bred spaltningsaktivitet, spaltningsstederne er de carboxyterminale peptidbindinger af alifatiske og aromatiske aminosyrer (figur 2). I processen med nukleinsyreekstraktion kan proteinase K nedbryde histoner, som er tæt bundet til nukleinsyrer, fremme adskillelsen af nukleinsyrer og gøre prøvenukleinsyre lettere at ekstrahere. Derudover kan proteinase K nedbryde RNA-hydrolase (RNase) aktivitet og inhibere RNase-hydrolyse af template-RNA.
Figur 2. Skematisk diagram af proteinase K hydrolyserende peptidbindinger
Yeasen Biotech Proteinase K (Cat#10401ES) er afledt af rekombinant gærstamme, specifik aktivitet ≥30 U/mg, fri for RNase og DNase og stabil enzymaktivitet i urinstof og SDS-opløsning. Det er aktivt i et bredt pH-område (pH 4,0~12,0), velegnet til spaltning og fjernelse af proteiner, såsom in situ hybridiseringsprøvepræparation og nukleinsyreoprensning.
2.2 Deoxyribonuklease I
Deoxyribonuclease I (DNase I) kan katalysere forskellige former for DNA, målrette mod spaltning af phosphodiesterbindinger ved siden af pyrimidiner og generere polynukleotider med en phosphatgruppe i 5'-enden og en hydroxylgruppe i 3'-enden, det gennemsnitlige fordøjelsesprodukt er den mindste fordøjelses-polyte.I processen med SARS-CoV-2-nukleinsyreekstraktion bruges DNase I hovedsageligt til at fjerne genomisk kontaminering i RNA-prøver, undgå DNA-rester i RNA-skabeloner og forbedre renheden af skabeloner.
Yeasen Biotech DNase I (Cat#10325ES) er afledt af rekombinante E. coli-stammer, RNase-fri og kan bruges til behandling af forskellige RNA-prøver. det optimale arbejds-pH-område er 7,0-8,0. I nærvær af Mg2+ kan DNase I spalte et hvilket som helst sted af dobbeltstrenget DNA tilfældigt; mens i nærværelse af Mn2+DNase I kan spalte DNA dobbeltstrenget på det samme sted og danne stumpe ender eller 1-2 nukleotider overhængende klæbrige ender (vist i figur 3).
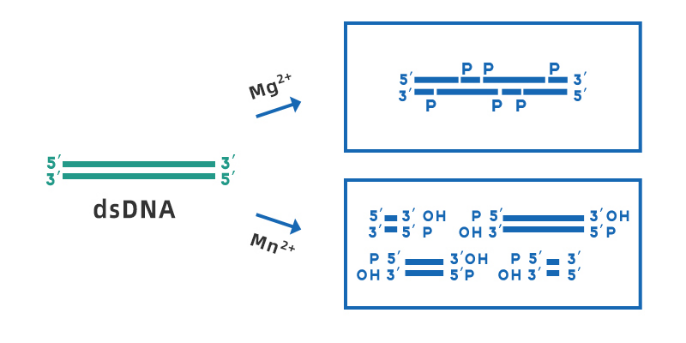
Fig. 3. Skematisk diagram af DNase I-spaltende dsDNA i nærvær af Mg2+ og Mn2+
2.3 RNase-hæmmer
I processen med SARS-CoV-2-nukleinsyredetektion kan ekstraktion og oprensning af prøvenukleinsyre eller forberedelse af det eksperimentelle reaktionssystem introducere ribonuklease (RNase)-kontamination, hvilket resulterer i nedbrydning af RNA-skabelonen. For at undgå RNase-kontamination er RNase-hæmmer påkrævet.
RNase-hæmmer er en specifik RNase-hæmmer i human placenta, som specifikt kan binde RNase til at danne et kompleks med en ikke-kovalent binding og inaktivere RNase. Yeasen Biotech Murin RNase-hæmmer (Cat#10603ES) indeholder ikke to cysteiner, der er meget følsomme over for oxidation i humane proteiner. Det har højere antioxidantaktivitet og kan i vid udstrækning hæmme forskellige typer RNaser (RNase A, B, C), mere velegnet til eksperimenter, der er følsomme over for højt dithiothreitol (DTT), såsom qPCR.
3. Kerneenzymer under RT-qPCR
Efter at nukleinsyreekstraktionen af SARS-CoV-2-prøven er afsluttet, kan nukleinsyrepåvisningen fuldføres med RT-qPCR. I processen med disse eksperimenter er DNA-polymerase, revers transkriptase og uracil-DNA-glycosylase alle essentielle kerneenzymråmaterialer.
3.1 Revers transkriptase
Efter ekstraktion og oprensning har SARS-CoV-2-RNA'et brug for revers transkriptase for at katalysere dNTP-polymerisering for at generere en cDNA-sekvens komplementær til skabelon-RNA'et (figur 4). Til RT-qPCR-reaktion skal højtemperaturresistent revers transkriptase vælges. På nuværende tidspunkt er MMLV revers transkriptase den mest udbredte, på grund af dens mangel på DNA-endonukleaseaktivitet og lave RNase H-aktivitet har den flere fordele ved anvendelsen af cDNA-kloning.
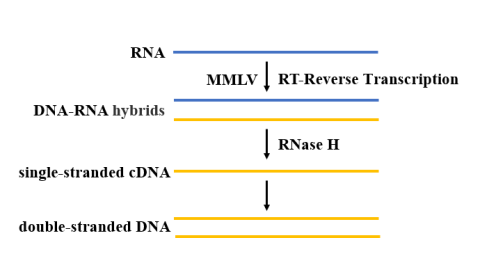
Figur 4. Skematisk diagram af omvendt transkriptionsprocessen
Yeasen Biotech Hifair™ V Omvendt transkriptase (Cat#11300ES) er en ny revers transkriptase opnået ved genteknologi. Den har god termisk stabilitet og kan modstå reaktionstemperaturer op til 60°C. Og det er det også velegnet til revers transkription af RNA-templates med komplekse sekundære strukturer. Samtidig øger enzymet affiniteten med templaten, er velegnet til revers transkription af et lille antal templates og lavkopigener og kan amplificere cDNA op til 10 kb.
3.2 DNA-polymerase
Efter at template-revers transkriptionsprocessen er afsluttet for at generere dobbeltstrenget cDNA, kræves "soul player"-DNA-polymerasen i PCR-reaktionen for at fremkomme ved polymerisering af frie deoxyribonukleotider for at forlænge DNA-kæden, og en stor mængde template-DNA amplificeres in vitro for at opnå formålet med viral nukleinsyredetektion.
Den DNA-polymerase, der almindeligvis anvendes i RT-qPCR-reaktioner, er hot-start Taq DNA-polymerase. Denne type enzym er inaktiv ved stuetemperatur.Det har først polymerisationsaktivitet efter varmstart, hvilket kan minimere genereringen af baggrundssignaler. Det løser problemerne med ikke-specifik amplifikation forårsaget af primer-dimer-generering eller mismatch i konventionelle PCR-reaktioner. På nuværende tidspunkt omfatter de almindeligt anvendte DNA-polymerase hot-start modifikationsmetoder hovedsageligt kemisk modifikation, ligandmodifikation og antistofmodifikation. Principperne for forskellige hot-start modifikationsmetoder er vist i figur 5.
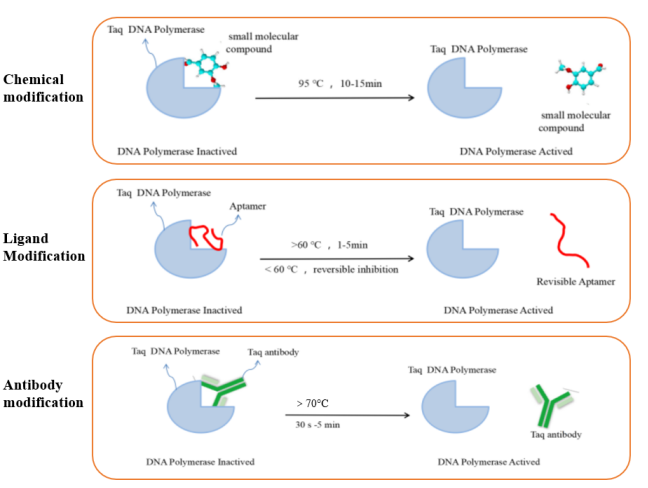
Figur 5. Skematisk diagram af forskellige typer modificerede hot-start enzymer
Yeasen Biotech UNICONTM Hotstart High Specific Taq DNA Polymerase, 5 U/μL (Cat#10726ES) er en dobbeltblokerende hot-start DNA-polymerase med høj skabelonaffinitet. Ved stuetemperatur, ikke kun 5'→3' polymeraseaktivitet er blokeret, men også 5'→3' exonukleaseaktivitet er blokeret. Denaturering ved 95°C i 30 sekunder kan fuldstændigt inaktivere det blokerende antistof og frigive DNA-polymeraseaktivitet og exonukleaseaktivitet. Den dobbeltblokerende funktion kan ikke kun effektivt forhindre uspecifik amplifikation forårsaget af fejlparringer eller primer-dimere, men også effektivt inhibere faldet i fluorescenssignal forårsaget af probe-nedbrydning. Den dobbelte garanti gør in vitro-detektionsreagenserne mere stabile under transport eller anvendelse ved stuetemperatur.
3.3 Uracil DNA glycosylase
I processen med påvisning af ny coronavirus-nukleinsyre er aerosolforurening i driftsmiljøet den mest almindelige faktor, der forårsager falsk positive PCR-resultater. Tilføjelse af UDG-enzym (Uracil DNA Glycosylase, uracil DNA-glycosylase) til amplifikationssystemet kan effektivt eliminere amplifikationsrestforurenende stoffer (for det meste i form af aerosoler) blandet i PCR-systemet for at sikre nøjagtigheden af amplifikationsresultaterne. Anti-forureningsprincippet for UDG-enzymet er vist i figur 6.
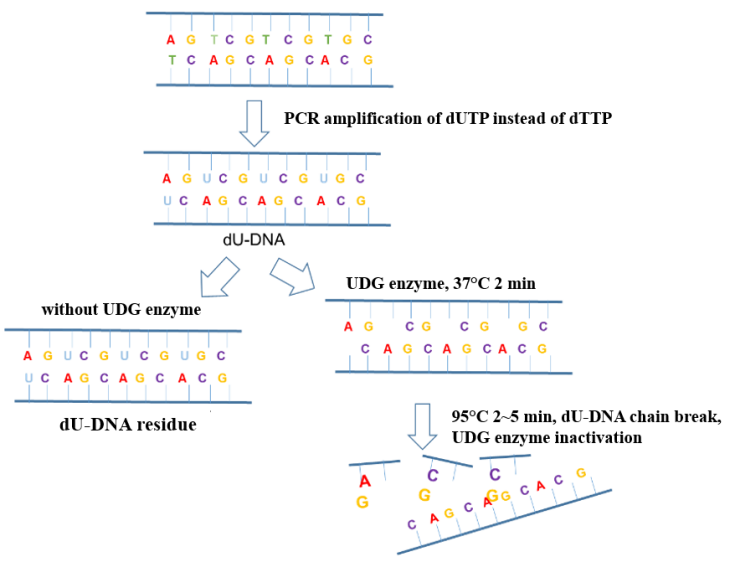
Figur 6. Skematisk diagram af anti-forureningsprincippet for UDG enzym
Yeasen Biotech Uracil DNA Glycosylase (UDG/UNG), varmelabil, 1 U/μL (Cat#10303ES) er aktiv ved 25-37°C, varmefølsom og irreversibelt inaktiveret ved 50°C i 10 minutter eller 95°C i 2 minutter. Der er ingen endonuklease- og RNase-rest, og værtsbakteriens genomrest er mindre end 10 kopier. Det kan bruges sammen med hot-start Taq DNA-polymerase for at sikre specificiteten af amplifikationsreaktionen.
4.Kerneenzymer af SARS-CoV-2 nukleinsyrepåvisning fra Yeasen
| Behandle | Beskrivelse | Produktnavn | SKU |
| Prøvebehandling | Protein fordøjelse | 10401ES | |
| RNA-ekstraktion | Rekombinant DNase I (RNase-fri) (Forespørgsel) | 10325ES | |
| RNase-hæmning | Murin RNase-hæmmer (40U/μL) | 10603ES | |
| Omvendt transskription | Velegnet til RT-qPCR | Hifair™ V Omvendt transkriptase (200U/μL) | 11300ES |
| HifairTM V omvendt transkriptase (600U/ μL) Glycerol-fri (Forespørgsel) | 11301ES | ||
| PCR-amplifikation | Hot-start DNA polymerase | 10726ES | |
| Termisk UDG | Uracil DNA Glycosylase (UDG/UNG), varmelabil, 1 U/μL | 10303ES |
Angående læsning:
Valg af omvendt transkriptase
YEASEN Varmelabil UDG——Kontroller nemt aerosolforurening
Murine RNase-hæmmere - Eliminerer RNase-kontamination med succes og bevarer RNA
