Restriktionsendonukleasen aus der Yeasen FuniCut™-Reihe, 5-minütige Verdauung mit Universalpuffer
Molekulares Klonen wird in fast jedem Labor eingesetzt. Restriktionsendonukleasen sind eine entscheidende Komponente bei Experimenten zum molekularen Klonen, aber es treten auch häufig verschiedene Probleme bei Experimenten mit Restriktionsendonukleasen auf. Häufige Probleme sind die folgenden:
- Es gibt so viele Arten von Restriktionsendonukleasen, dass die Auswahl schwerfällt.
- Fehlspaltung, zufällige Spaltung oder unvollständige enzymatische Spaltung.
- Die Verdauung erfolgt träge; sie kann eine Stunde oder sogar die ganze Nacht dauern.
- Darüber hinaus müssen bei Mehrfachenzymverdauungsexperimenten auch verschiedene Enzymverdauungsverfahren gewählt werden.
Um Ihnen ein besseres Verständnis von Restriktionsendonukleasen zu vermitteln, wird im Folgenden als Antwort auf die erste Frage kurz erklärt, was Restriktionsendonukleasen sind und wie verschiedene Endonukleasen klassifiziert werden. Die
1. Was ist eine Restriktionsendonuklease?
2. Einschränkungen Endonuclease Nomenklatur
3. Einschränkungen bei der Klassifizierung von Endonukleasen
4.
1. Was ist eine Restriktionsendonuklease?
Restriktionsendonukleasen sind eine Klasse von Enzymen, die bestimmte Nukleotidsequenzen in doppelsträngigen DNA-Molekülen erkennen und Phosphodiesterbindungen in DNA-Ketten an bestimmten Stellen durchtrennen können.
Verschiedene Restriktionsendonukleasen erkennen unterschiedliche DNA-Sequenzen. Sie können DNA innerhalb der Erkennungssequenz oder an einer Stelle unweit der Erkennungssequenz schneiden, wodurch verschiedene Produkte entstehen, wie in Abbildung 1 dargestellt. BamHI bildet klebrige Enden, KpnI bildet 3'-Klebeenden und EcoRV bildet stumpfe Enden.
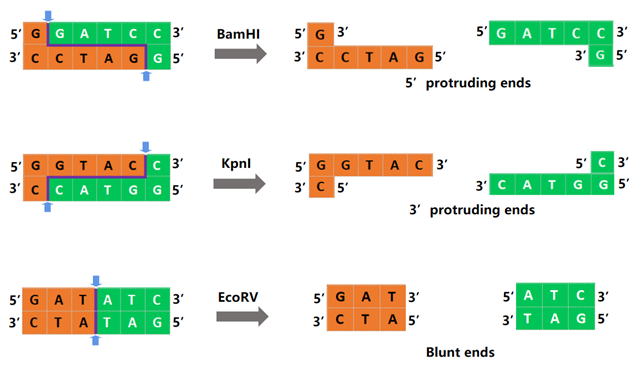
Abbildung 1. Schematische Darstellung der Verdauung von BamHI, KpnI und EcoRV
2. Einschränkungen Endonuclease Nomenklatur
Der Gattungsname, der Artname, der Bakterienstamm oder Serotyp und die Reihenfolge der Entdeckung sind die wichtigsten Faktoren, die zur Benennung von Restriktionsendonukleasen verwendet werden. Die Namensrichtlinien sind in Tabelle 1 unten am Beispiel von BstEII aufgeführt:
Tabelle 1. Nomenklatur der Restriktionsendonuklease
| Abkürzung | Vollständiger Name | Implikation |
| B | Bazillus | Erster Buchstabe des Gattungsnamens |
| st | stearothermophilus | Die ersten beiden Buchstaben des Artnamens |
| E | ET | Der erste Buchstabe des Sortennamens |
| II | Zweite Entdeckung | Die Ordnung in solchen Bakterien |
3.Einschränkungen bei der Endonukleaseklassifizierung
Je nach Komplexität der Struktur, Wirkungsweise und Unterschiedlichkeit der Cofaktoren können Restriktionsenzyme in vier Kategorien unterteilt werden. Die folgende Tabelle 2 fasst die Eigenschaften und typischen Enzyme jeder Kategorie zusammen.
Tabelle 2. Die Unterschiede zwischen verschiedenen Restriktionsendonukleasen
| Endonuklease-Klasse | Merkmale | Typische Enzymspezies |
| Typ I | 1. Erkennung und Modifikationsspaltung. 2. Es kann bestimmte DNA-Sequenzen erkennen und die Verdauungsstelle kann bis zu Tausenden von Basen unendlich weit von der Erkennungsstelle entfernt sein. 3. Aktion erfordert ATP. | EcoB, EcoK usw. |
| Typ II | 1. Hat nur die Funktion der Schnitterkennung. 2. Die Erkennungssequenz ist häufig eine kurze palindromische Sequenz (etwa 4–8 bp), und die spezifische Verdauungsstelle der Restriktionsendonuklease ist typischerweise die erkannte Sequenz. 3. Aktion erfordert Mg2+. 4. Die Art der Restriktionsenzyme, die beim molekularen Klonen am häufigsten verwendet werden. | HindIII, NotI usw. |
| Typ III | 1. Erkennung und Modifikationsverarbeitung. 2. Über trennenp Trennen Sie die Erkennungsstelle von der Verdauungsstelle. 3. Aktion erfordert ATP. | HinfIII et al. |
| Typ IV | 1. Schneidet nur methylierte DNA-Sequenzen. 2. Etwa 30 bp trennen die Erkennungsstelle von der Verdauungsstelle. | McrA, McrBC usw. |
Wie in Tabelle 3 unten angegeben, können sie basierend auf den Ähnlichkeiten und Unterschieden zwischen den Erkennungs- und Verdauungsstellen auch in drei Gruppen unterteilt werden.
Tabelle 3.Vergleichende Analyse von Isoschizomer, Neoschizomer und Isocaudarner
|
| Isoschizomer | Neoschizomer | Isocaudarner |
| Erkennungssequenz | Dasselbe | Dasselbe | anders |
| Verdauungsstellen | Dasselbe | anders | Dasselbe |
| typischer Vertreter | AgeI und BshTI: beide erkennen und spalten 5′-A↓CCGGT-3′ | SmaI (5′-CCC↓GGG-3′) und XmaI(5′-C↓CCGGG-3′) | BamHI (5'-G↓GATCC-3') und BglII (5'-A↓GATCT-3') |
Angesichts der Tatsache, dass einige Restriktionsendonukleasen auf dem Markt noch immer über Nacht verdaut werden müssen und die Produkte zu fehlerhafter Verdauung neigen, ermöglicht
4. Yeasen FuniCut™ Schnelle Restriktionsendonukleasen
Abbildung 2.
4.1 Funktionen
- Schnelle Verdauung: Die Verdauung kann in 5–15 Minuten abgeschlossen werden, was im Vergleich zu herkömmlichen Verdauungsexperimenten um mehr als 1–2 Stunden verkürzt werden kann.
- Universeller Puffer: Die Multienzym-Spaltungsreaktion wird dadurch vereinfacht, dass jede Kombination von Endonukleasen denselben Puffer verwenden kann.
- Wirkung der Enzymverdauung: vergleichbar mit N* und geeignet für dessen Puffer.
- Die direkte Elektrophorese vereinfacht den Vorgang, indem sie einen roten Puffer bereitstellt und die Verdauungsprodukte direkt zur Elektrophorese weiterleitet.
- Präzise Verdauung: Auch bei der Verdauung über Nacht ist die Aktivität der Sterne sehr gering.
4.2 Leistungsschau
4.2.1 Im FuniCut™-Puffer finden Einzel-, Doppel- und Dreifachverdauungen in 5 Minuten statt.

Abbildung 3. Einfach-, Doppel- und Dreifachverdauung mit dem FuniCut™ Der Puffer kann typischerweise in 5 Minuten fertiggestellt werden, beispielsweise die Dreifachverdauung mit EcoRI+KpnI+SmaI.
4.2.2 Die Wirkung der Enzymverdauung ist mit der von N* und T* vergleichbar und funktioniert gut mit deren Puffern.

Abbildung 4. Im Vergleich zu ähnlichen N*- und T*-Produkten ist die enzymverdauende Wirkung von
4.2.3 Übernachtverdauung mit sehr geringer Sternaktivität
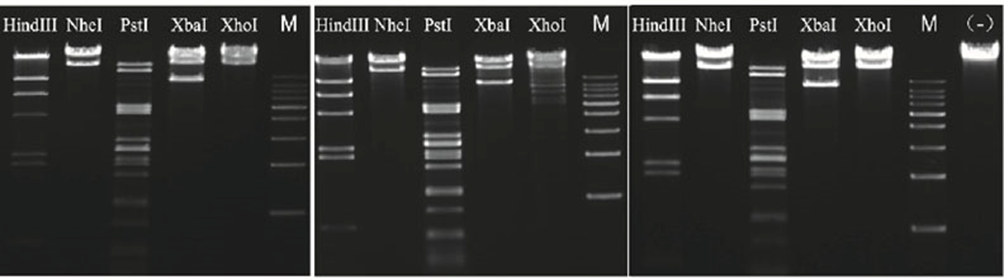
Abbildung 5. Verwenden Sie die schnellen Endonukleasen HindIII, NheI, PstI, XbaI und XhoI von
4.2.4 Hochgradig redundant und in der Lage, etwas überschüssiges Substrat zu verarbeiten.
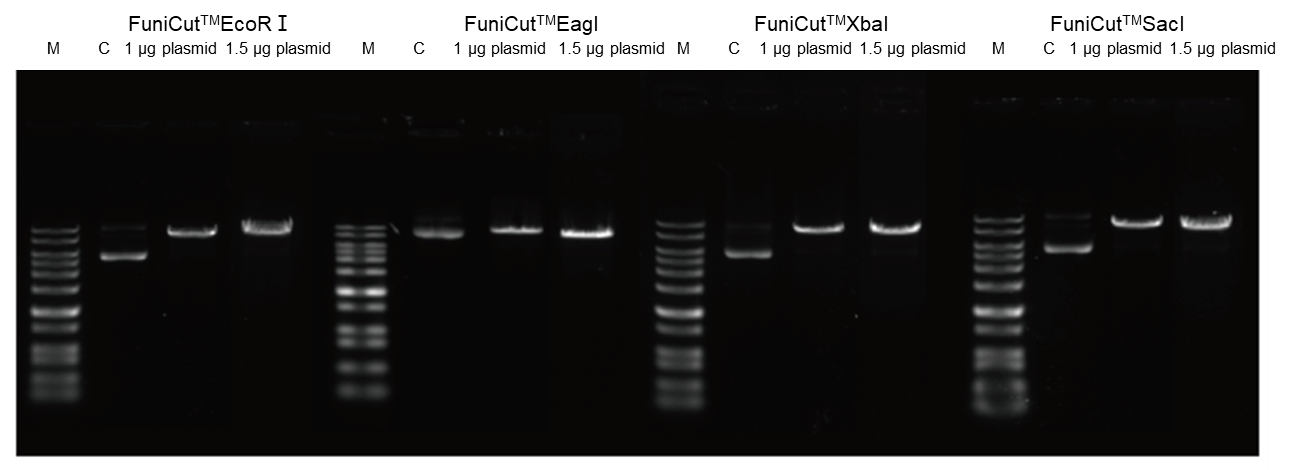
Abbildung 6. Unter Verwendung der FuniCut™ Fast Restriction Endonucleases EcoR I, Eag I, XbaI und Sac I wurden 1 μg bzw. 1,5 μg Plasmid als Substrate verwendet und 5 Minuten lang verdaut. Die verdauten Produkte wurden einer Agarosegelelektrophorese unterzogen.
Tabelle 4.Produkte und Reaktionspufferkompatibilität
| Produktname | Katze# | Erkennungssequenz | Temperatur | Thermische Deaktivierungstemperatur |
| T* Puffer | N* Puffer | Ta*-Puffer | Geschützte Base (bp) | Antworten |
| FuniCut™ AscI (erkundigen) | 15001ES50 | GG/CGCGCC | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 50T |
| FuniCut™ AvrII (erkundigen) | 15002ES25 | C/CTAGG | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 25T |
| FuniCut™ BamHI (erkundigen) | 15003ES76 | G/GATCC | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 500T |
| FuniCut™ BclI (erkundigen) | 15004ES62 | T/GATCA | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 125T |
| FuniCut™ BsaI (erkundigen) | 15005ES50 | GGTCTC (1/5) | 37 | 80 | 100 | 100 | 100 | 100 | Nicht | 50T |
| FuniCut™ BstEII (erkundigen) | 15006ES60 | G/GTNACC | 37 | 80 | 100 | 75 | 100 | 75 | Nicht | 100T |
| FuniCut™ ClaI (erkundigen) | 15007ES50 | AT/CGAT | 37 | 80 | 100 | 100 | 100 | 100 | 4 | 50T |
| FuniCut™ DpnII (erkundigen) | 15008ES50 | /GATC | 37 | 80 | 100 | 75 | 100 | 75 | Nicht | 50T |
| FuniCut™ EagI (erkundigen) | 15009ES25 | C/GGCCG | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 25T |
| FuniCut™ EcoRI (erkundigen) | 15010ES78 | G/AATTC | 37 | 80 | 100 | 100 | 100 | 100 | 5 | 600T |
| FuniCut™ EcoRV (erkundigen) | 15011ES70 | GAT/ATC | 37 | 80 | 100 | 100 | 100 | 100 | 5 | 200T |
| FuniCut™ HindIII (erkundigen) | 15012ES76 | A/AGCTT | 37 | 80 | 100 | 75 | 100 | 100 | 3 | 500T |
| FuniCut™ HpaI (erkundigen) | 15013ES50 | GTT/AAC | 37 | 80 | 100 | 100 | 100 | 50 | 1 | 50T |
| FuniCut™ KpnI (erkundigen) | 15015ES70 | GGTAC/C | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 200T |
| FuniCut™ MluI (erkundigen) | 15016ES60 | A/CGCGT | 37 | 80 | 100 | 75 | 100 | 100 | 3 | 100T |
| FuniCut™ NcoI (erkundigen) | 15018ES30 | C/CATGG | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 30T |
| FuniCut™ NdeI (erkundigen) | 15019ES70 | CA/TATG | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 200T |
| FuniCut™ NheI (erkundigen) | 15020ES30 | G/CTAGC | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 30T |
| FuniCut™ NichtI (erkundigen) | 15021ES50 | GC/GGCCGC | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 50T |
| FuniCut™ PstI (erkundigen) | 15022ES76 | CTGCA/G | 37 | NEIN | 100 | 100 | 100 | 100 | 2 | 500T |
| FuniCut™ SacI (erkundigen) | 15023ES60 | GAGCT/C | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 100T |
| FuniCut™ SalI (erkundigen) | 15024ES70 | G/TCGAC | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 200T |
| FuniCut™ SbfI (erkundigen) | 15025ES25 | CCTGCA/GG | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 25T |
| FuniCut™ SmaI (erkundigen) | 15027ES60 | CCC/GGG | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 100T |
| FuniCut™ Spiel (erkundigen) | 15028ES50 | A/CTAGT | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 50T |
| FuniCut™ SphI (erkundigen) | 15029ES50 | GCATG/C | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 50T |
| FuniCut™ SspI (erkundigen) | 15030ES56 | AAT/ATT | 37 | 80 | 100 | 50 | 100 | 100 | 2 | 60T |
| FuniCut™ StuI (erkundigen) | 15031ES60 | AGG/CCT | 37 | 80 | 100 | 100 | 100 | 100 | 1 | 100T |
| FuniCut™ TaqI (erkundigen) | 15032ES70 | T/CGA | 65 | NEIN | 100 | 100 | 100 | 100 | 4 | 200T |
| FuniCut™ XbaI (erkundigen) | 15033ES76 | T/CTAGA | 37 | 80 | 100 | 50 | 100 | 100 | 2 | 500T |
| FuniCut™ XhoI (erkundigen) | 15034ES76 | C/TCGAG | 37 | 80 | 100 | 100 | 100 | 100 | 4 | 500T |
| FuniCut™ FspI (erkundigen) | 15036ES50 | TGC/GCA | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 50T |
| FuniCut™ HinfI (erkundigen) | 15038ES76 | G/ANTC | 37 | 80 | 100 | 100 | 100 | 50 | 2 | 500T |
| FuniCut™ ApaLI (erkundigen) | 15039ES70 | G/TGCAC | 37 | 80 | 100 | 75 | 100 | 100 | 4 | 200T |
| FuniCut™ NruI (erkundigen) | 15040ES50 | Sammelkartenspiel/CGA | 37 | NEIN | 100 | 100 | 100 | 100 | 5 | 50T |
| FuniCut™ PacI (erkundigen) | 15041ES25 | TTAAT/TAA | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 25T |
| FuniCut™ PvuII (erkundigen) | 15042ES70 | CAG/CTG | 37 | NEIN | 100 | 100 | 100 | 100 | 4 | 200T |
| FuniCut™ SacII (erkundigen) | 15043ES50 | CCGC/GG | 37 | 80 | 100 | 100 | 100 | 100 | 4 | 50T |
| FuniCut™ NsiI (erkundigen) | 15044ES25 | ATGCA/T | 37 | 80 | 100 | 100 | 100 | 100 | 3 | 25T |
| FuniCut™ Esp3I (BsmBI) (erkundigen) | 15048ES30 | CGTCTC(1/5) | 37 | 80 | 100 | 100 | 100 | 100 | 5 | 30T |
| FuniCut™ BglII (erkundigen) | 15049ES60 | A/GATCT | 37 | 80 | 100 | 100 | 100 | 50 | 2 | 100T |
| FuniCut™ BstBI (erkundigen) | 15050ES60 | TT/CGAA | 37 | 80 | 100 | 100 | 100 | 100 | Nicht | 100T |
| FuniCut™ MnlI (erkundigen) | 15051ES50 | CCTC (7/6) | 37 | 80 | 100 | 100 | 100 | 100 | 2 | 50T |
CpG: Beeinflusst durch CpG-Methylierung.
EK: Beeinflusst durch EcoKI-Methylierung.
EB: Beeinflusst durch EcoBI-Methylierung.
Dcm: Beeinflusst durch Dcm-Methylierung.
Dam: Betroffen durch Dam-Methylierung.

