Hochwertige Rohstoffe für die isotherme Verstärkung – macht RT-LAMP empfindlicher und schneller!
Die isotherme Amplifikationstechnologie kann das Ziel erreichen, spezifische Nukleinsäuresequenzen unter konstanten Temperaturbedingungen zu amplifizieren. Da keine spezielle Ausrüstung erforderlich ist, die Amplifikationszeit kurz ist und die Empfindlichkeit hoch ist, hat sie gute Anwendungsaussichten bei der Vor-Ort-Erkennung und der Point-of-Care-Diagnose gezeigt und eignet sich sehr gut für die Entwicklung molekulardiagnostischer Werkzeuge.
Im Zusammenhang mit der neuen Coronavirus-Pandemie mit zunehmenden Stämmen und Infektiosität kann die Isothermal-Amplifikationstechnologie das neue Coronavirus erkennen Dies trägt dazu bei, das Problem zu lösen, dass bei manchen COVID-19-Patienten die Diagnose aufgrund der langen Nachweiszeit und der hohen Anforderungen an Nachweisgeräte und Reagenzien herkömmlicher PCR-Techniken nicht schnell bestätigt werden kann. Was ist also das Prinzip von RT-LAMP, einer der isothermen Verstärkungstechniken? Welche hochwertigen Rohstoffe kann
1. Was ist das Prinzip von RT-LAMP?
2. Primer-Design für RT-LAMP
3. Welche Rohstoffe kann
4. Leitfaden zur Produktauswahl
1. Was ist das Prinzip von RT-LAMP?
Im Jahr 2000 entwickelten die japanischen Wissenschaftler Notomi und andere eine neue Nukleinsäureamplifikationstechnologie namens Loop-Mediated Isothermal Amplification (LAMP). LAMP verwendet 4 spezifische Primer, die für 6 Regionen des Zielgens entwickelt wurden, und verwendet Strangverdrängungs-DNA-Polymerase, um 109 Kopien der Zielsequenz in zehn Minuten unter isothermen Bedingungen (60 bis 65 °C) zu amplifizieren. Die Ergebnisse wurden durch Agarosegelelektrophorese beurteilt und die positiven Ergebnisse zeigten leiterförmige Bänder. LAMP zeichnet sich durch starke Spezifität, hohe Empfindlichkeit, schnelle und einfache Bedienung und niedrige Kosten aus. Mit der kontinuierlichen Verbesserung und Perfektionierung dieser Technologie wird sie derzeit zum Nachweis verschiedener pathogener Mikroorganismen eingesetzt.
RT-LAMP ist eine Methode, bei der dem LAMP-Amplifikationssystem Reverse Transkriptase hinzugefügt wird, wodurch die direkte Erkennung viraler RNA möglich wird. Die Amplifikationseffizienz von LAMP ist extrem hoch, sodass eine große Menge Nukleinsäure mit nur einer kleinen Menge cDNA amplifiziert werden kann. Die Zeit der Reverse Transkription im Nukleinsäureamplifikationsprozess wird eingespart und die Erkennungsgeschwindigkeit von RNA wird beschleunigt.
DNA befindet sich bei etwa 65 °C in einem dynamischen Gleichgewichtszustand. Unter Einwirkung der Strangverdrängungs-DNA-Polymerase wird sie, ausgehend vom 3'-Ende des F2-Segments des FIP-Primers, mit der komplementären Sequenz der Matrizen-DNA gepaart, um die Strangverdrängungs-DNA-Synthese zu initiieren. Der F3-Primer ist am vorderen Ende von F2c komplementär zu F3c und nimmt das 3'-Ende als Ausgangspunkt zur Synthese seiner DNA, während er den vom führenden FIP-Primer synthetisierten DNA-Strang durch die Einwirkung einer Strangverdrängungs-DNA-Polymerase ersetzt und nach vorne verlängert. Der vom letzten F3-Primer synthetisierte DNA-Strang bildet mit einem Matrizen-DNA-Strang einen Doppelstrang. Der vom FIP-Primer synthetisierte DNA-Strang wird durch den F3-Primer-Strang ersetzt, um einen Einzelstrang zu erzeugen. Dieser Einzelstrang weist am 5'-Ende komplementäre F1c- und F1-Segmente auf, sodass eine Selbstbasenpaarung stattfindet und eine ringförmige Struktur bildet. Und der BIP-Primer wird hybridisiert und mit dem Einzelstrang kombiniert, und das 3'-Ende des BIP-Primers wird als Ausgangspunkt für die Synthese eines komplementären Strangs verwendet, und die Struktur wird dabei geöffnet.Anschließend werden Primer ähnlich F3 und B3 vom BIP-Primer eingefügt, es wird eine komplementäre Basenpaarung durchgeführt und ein neuer komplementärer Strang wird vom 3'-Ende als Ausgangspunkt synthetisiert. An beiden Enden der ersetzten einzelsträngigen DNA befinden sich komplementäre Sequenzen, und es kommt zur Selbstbasenpaarung, um eine kreisförmige Struktur zu bilden, sodass die gesamte Kette eine hantelähnliche Struktur aufweist. Diese Struktur ist die Ausgangsstruktur des Amplifikationszyklus der RT-LAMP-Methode.
In der hantelförmigen Struktur wird die DNA-Verlängerung durchgeführt, wobei das F1-Segment am 3'-Ende als Ausgangspunkt verwendet wird und es sich selbst als Vorlage verwendet. Und der FIP-Primer F2 hybridisiert mit dem einzelsträngigen F2c auf der Schleife, um eine neue Runde der Strangverdrängungsreaktion einzuleiten. Die aus dem F1-Segment synthetisierte doppelsträngige Nukleinsäure wird dissoziiert, und auf der Nukleinsäure bildet sich auf ähnliche Weise eine kreisförmige Struktur. Es gibt eine einzelsträngige Form von B2c auf der kreisförmigen Struktur, und B2 auf dem BIP-Primer hybridisiert damit, um eine neue Amplifikationsrunde einzuleiten, und durch denselben Prozess wird eine kreisförmige Struktur gebildet. Gemäß diesem Prozess durchlaufen komplementäre Sequenzen auf demselben Strang Paarung, Strangverlängerung und bilden schließlich Strukturen unterschiedlicher Größe.
2. Primer-Design für RT-LAMP
Das Primerdesign ist der Schlüssel zur erfolgreichen Amplifikation und Erkennung von RT-LAMP. RT-LAMP-Primer umfassen zwei äußere Primer (F3 und B3) und zwei innere Primer (FIP und BIP). F3-Primer: Der vorgelagerte äußere Primer, bestehend aus der F3-Region, ist komplementär zur F3c-Region des Zielgens. FIP-Primer: vorgelagerter innerer Primer, bestehend aus der F2-Region, die F2c-Region am 3'-Ende des Zielgens in der F2-Region ist komplementär zur F1c-Region am 5'-Ende des Zielgens. BIP-Primer: nachgelagerter innerer Primer, bestehend aus der B2-Region, die B2-Region ist komplementär zur B2c-Region am 3'-Ende des Zielgens und hat dieselbe Sequenz wie die B1c-Region am 5'-Ende des Zielgens.
Ähnlich wie bei der PCR sollten bei der Gestaltung von Primern Faktoren wie Basenzusammensetzung, GC-Gehalt und Substruktur beachtet werden. Darüber hinaus sollten die folgenden Punkte beachtet werden. Die 5'-Endabschnitte der inneren Primer FIP und BIP, d. h. F1c und B1c, sind im Allgemeinen 8–50 bp lang. Die Länge beträgt vorzugsweise 15–25 bp und der Tm-Wert ist größer als die Tm-Werte von F2 und B2 am 3'-Endabschnitt. Die 3'-Endabschnitte der inneren Primer FIP und BIP, d. h. F2 und B2, sind im Allgemeinen 8–50 bp lang. Die Länge beträgt vorzugsweise 15–25 bp und der Tm-Wert entspricht der optimalen Temperatur der im Experiment ausgewählten Bst-DNA-Polymerase. Die Längen der äußeren Primer F3 und B3 betragen im Allgemeinen 8–50 bp. Die Länge beträgt vorzugsweise 15–25 bp und ihr Tm-Wert ist kleiner als der von F2 und B2. Beim Entwurf von Primern sollten die Schleifenstruktur und die Größe der Zielsequenz berücksichtigt werden. Wenn die Anzahl der Basen in der Schleife größer als 40 bp ist und die Größe der Zielsequenz 130-200 bp beträgt, ist die Amplifikationseffizienz am höchsten.
3. Welche Rohstoffe kann Yeasen liefern?
3.1 [Neues Upgrade] Yeasen Bst Plus DNA-Polymerase
Das neu aufgerüstete Hieff™ Bst Plus DNA-Polymerase (Kat.-Nr. 14402ES, 14403ES) wird durch Expression und Reinigung des DNA-Polymerasegens von Bacillus stearothermophilus gewonnen sp dem die 5′→3′-Exonukleasedomäne in E. coli fehlt.Das Enzym verfügt über eine starke Strangverdrängungsfähigkeit und bietet die Vorteile einer hohen Empfindlichkeit, einer hohen Amplifikationseffizienz und einer hohen dUTP-Toleranz. Es kann auf Basis der isothermen Amplifikationstechnologie häufig zur Echtzeiterkennung von Krankheitserregern eingesetzt werden.
3.1.1 Schnell und hochempfindlich
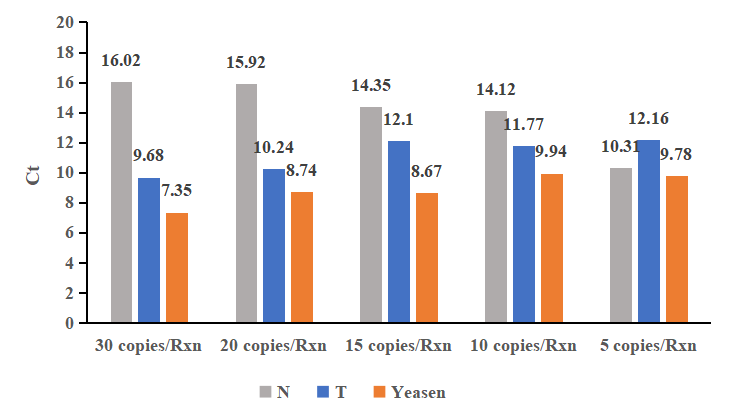
Abbildung 1. Die RT-LAMP-Reaktion wurde mit
4. Leitfaden zur Produktauswahl
Die von
Tabelle 1.Produktinformationen
| Produktpositionierung | Produktname | Katze# |
| Hochempfindliches Bst-Enzym | 14402ES | |
| Hieff™ Bst Plus DNA-Polymerase (2000 U/μl) (Erkundigen) | 14403ES | |
| Fluoreszenzfarbstoffmethode RT-Lampen-Display-Kit | 13762ES | |
| Reverse Transkriptase geeignet für RT-Lamp | Hifair™ Ⅲ Reverse Transkriptase (Erkundigen) | 11111ES |
| Hifair™ Ⅲ Reverse Transkriptase, Glycerin-frei (Erkundigen) | 11297ES | |
| Muriner RNase-Inhibitor | 10603ES | |
| 10703ES | ||
| Hitzelabiles UDG | Uracil-DNA-Glykosylase (UDG/UNG), hitzelabil, 1 U/μL (Erkundigen) | 10303ES |
| Hochreines dUTP | 10128ES |
Zum Thema Lesen:
