T7 High Yield RNA Synthesis Kit – Effiziente RNA-Synthese für die Forschung
T7 RNA-Synthese-Kit mit hoher Ausbeute optimiert das Transkriptionsreaktionssystem. Das Kit kann die einzelsträngige RNA effizient synthetisieren, indem es T7-RNA-Polymerase, die lineare doppelsträngige DNA mit der T7-Promotorsequenz als Vorlage und NTPs als Substrat zur Steuerung der DNA-Sequenz stromabwärts des Promotors verwendet. Während der Transkription können dem Substrat modifizierte Nukleotide hinzugefügt werden, um Biotin oder farbstoffmarkierte RNA herzustellen.
1. Einführung des T7 High Yield RNA Synthesis Kit
2. Grundsätze der in vitro Transkription
3. Vorteile des
4. Produktleistung
5. So verwenden Sie dieses Kit
6. Häufig gestellte Fragen
7. Bestellinformationen
1. Einführung des T7 High Yield RNA Synthesis Kit
Das T7 High Yield RNA Synthesis Kit kann sowohl lange als auch kurze Transkripte synthetisieren, und RNA kann mit 1 μg DNA-Vorlagen-Input in Mengen von 100-200 μg produziert werden. Die synthetisierte RNA kann für verschiedene nachgelagerte Anwendungen verwendet werden, wie z. B. RNA-Struktur- und Funktionsforschung, RNase-Schutz, Sondenhybridisierung, RNA-Interferenz, Mikroinjektion und in vitro Übersetzung.
2. Grundsätze der in vitro Transkription
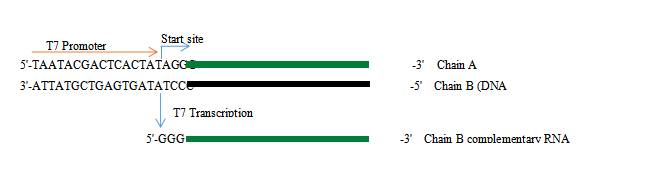
Abbildung 1. In vitro Transkriptionsprozess
3. Vorteile des Yeasen T7 High Yield RNA Synthesis Kit
Extrem hohe Ausbeute – bis zu 200 µg in 2 Stunden
Vielseitig – geeignet für 20nt-10000nt RNA-Transkripte
Vielseitig einsetzbar – erzeugt unmarkierte, markierte oder gecappte RNA
4. Produktleistung
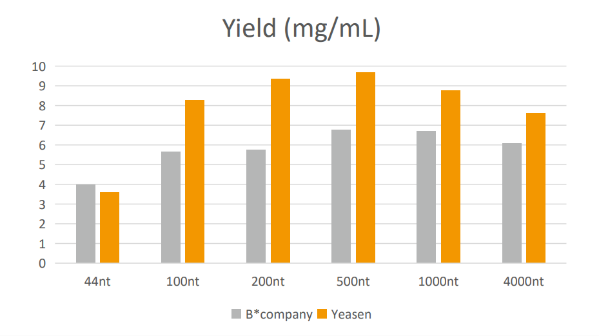
Abbildung 2. IVT-Ertragsvergleich
Hinweis: Alle Reaktionsreagenzien stammen von T7 RNA-Synthese-Kit (
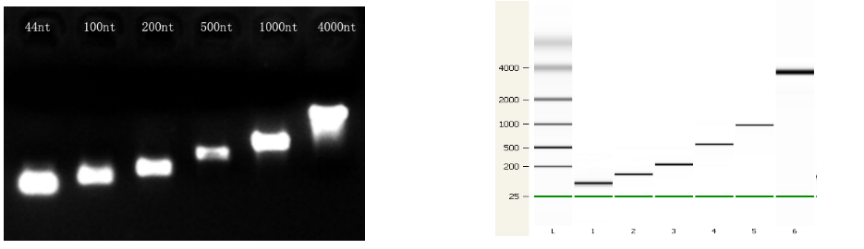
Abbildung 3. Die Qualität der Transkriptionsprodukte bei unterschiedlichen Längen
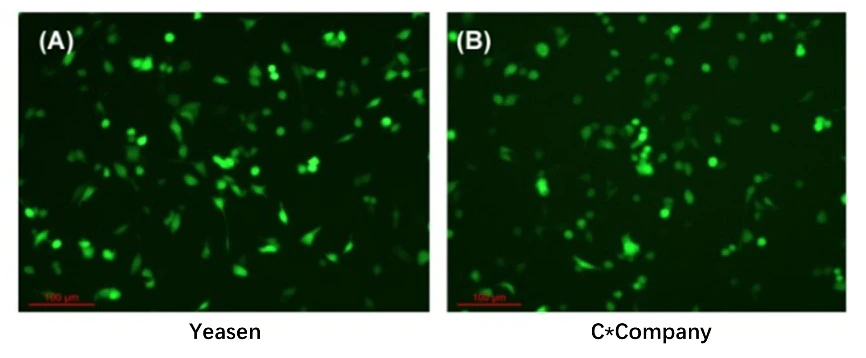
Abbildung 4. Der Expressionsgrad der transkribierten mRNA in HEK-293-Zellen
Hinweis: Die mRNA ist mit dem Vaccinia Capping Enzyme (
5. So verwenden Sie dieses Kit
5.1 Reagenzien auftauen
Zentrifugieren Sie den T7-RNA-Polymerase-Mix kurz und legen Sie ihn auf Eis. Tauen Sie 10× Transkriptionspuffer und Ribonukleotide (ATP, CTP, GTP, UTP) auf, mischen Sie und zentrifugieren Sie ihn auf den Boden des Röhrchens, stellen Sie 10× Transkriptionspuffer bei Raumtemperatur auf und legen Sie vier Arten von Ribonukleotiden auf Eis.
5.2 Bereiten Sie das Transkriptionsreaktionssystem gemäß der folgenden Tabelle vor
Tabelle 1.Herstellungsverfahren für das Transkriptionsreaktionssystem
| Komponenten | Volumen (μL) | Endkonzentration |
| RNase-freies H2O | Bis zu 20 | - |
| 10×Transkriptionspuffer | 2 | 1× |
| CTP / GTP / ATP / UTP (je 100 mM) | je 2 | Jeweils 10 mM |
| Vorlagen-DNA | 1 µg | - |
| T7 RNA-Polymerase-Mischung | 2 | - |
Notiz:
a) Die Reaktion wird bei Raumtemperatur vorbereitet. Da der 10× Transkriptionspuffer Spermidin enthält, führt eine zu hohe Spermidinkonzentration bei niedrigen Temperaturen zur Ausfällung der DNA-Vorlage.
b) Kurzes Transkript (<100 nt), 2 µg Vorlage können verwendet werden, die Transkriptionszeit erhöht sich auf 4–8 Stunden.
c) Für lange Transkripte (> 1000 nt) wird die Verwendung linearisierter Plasmidvorlagen zur Transkription empfohlen.
d) Führen Sie die Reaktion im PCR-Gerät bei geöffnetem Heißdeckel durch, um ein Verdunsten der Reaktionslösung über längere Zeit zu verhindern.
e) Das Reaktionsprodukt kann einen weißen Niederschlag aufweisen. Dies ist Magnesiumpyrophosphat, das während der Reaktion aus freiem Pyrophosphat und Magnesiumionen gebildet wird und die nachfolgenden Experimente nicht beeinflusst. Sie können EDTA hinzufügen, um es zu entfernen. Wenn Sie befürchten, dass die Zugabe von EDTA nachfolgende Experimente beeinflusst, kann der Überstand auch durch Zentrifugieren zurückgewonnen werden.
f) Die Reagenzien und Behälter sollten frei von RNase-Kontamination sein.
5.3 2 Stunden bei 37°C inkubieren
Mischen Sie die obige Reaktionslösung, zentrifugieren Sie sie kurz auf den Boden des Röhrchens und inkubieren Sie sie 2 Stunden lang bei 37 °C. Wenn die Transkriptlänge weniger als 100 nt beträgt, erhöhen Sie die Reaktionszeit auf 4–8 Stunden.
5.4 DNase I-Behandlung (optional)
Nachdem die Reaktion abgeschlossen ist, geben Sie 2 μl DNase I (RNase-frei) in jedes Röhrchen und inkubieren Sie es 15 Minuten lang bei 37 °C, um die Matrizen-DNA zu entfernen.
6. Häufig gestellte Fragen
6.1 Die Ausbeute der IVT-Reaktion ist gering
Die Qualität der Vorlage hängt eng mit der Ausbeute zusammen. Wenn die Ausbeute der Versuchsgruppe deutlich niedriger ist als die der positiven Kontrollgruppe, kann dies folgende Gründe haben:
① Die Vorlagen enthalten Komponenten, die die IVT-Reaktion hemmen.
② Mit den Vorlagen stimmt etwas nicht.
6.2 Vorschläge
① Reinigen Sie die Vorlage erneut.
② Bestätigen Sie die Konzentration und Integrität der Vorlagen.
③ Verlängern Sie die Reaktionszeit.
④ Erhöhen Sie den Eingabewert der Vorlage.
⑤Probieren Sie andere RNA-Polymerasen und entsprechende Promotoren aus.
6.3 Die Ausbeute an Kurztranskripten ist gering
Wenn die Zieltranskripte kürzer als 100 nt sind, verlängern Sie die Reaktionszeit auf 4–8 Stunden oder erhöhen Sie die Vorlageneingabe auf 2 µg in einem 20 µL-Reaktionssystem.
6.4 Es gibt unerwartet längere Transkripte
Sollte das Ergebnis der Gelelektrophorese zeigen, dass unerwartet längere Transkripte vorliegen, kann das folgende Gründe haben:
① Plasmidvorlagen sind möglicherweise nicht vollständig linearisiert.
②Vorlagen haben zusammenhängende Enden mit 3 Fuß Überhang.
③Transkripte haben eine Sekundärstruktur, die nicht vollständig denaturiert ist.
6.5 Vorschläge
①Überprüfen Sie, ob die Plasmidvorlagen vollständig linearisiert sind. Führen Sie die Plasmidlinearisierung ggf. erneut durch.
②Wählen Sie geeignete Restriktionsenzyme aus, um Plasmidvorlagen zu linearisieren und die Bildung kohäsiver Enden mit 3'-Überhängen zu vermeiden. Es ist praktikabel, Klenow-Fragmente oder T4-DNA-Polymerase zu verwenden, um bei Bedarf ein stumpfes Ende zu erzeugen.
③Verwenden Sie denaturiertes Gel, um Transkripte zu erkennen.
6.6 Es gibt unerwartet kürzere Transkripte
Sollte das Ergebnis der Gelelektrophorese unerwartet kürzere Transkripte aufweisen, kann das folgende Gründe haben:
①Es gibt eine Sequenz, die der Terminationssequenz der T7-RNA-Polymerase in Vorlagen analog ist.
②Der GC-Gehalt der Vorlagen ist hoch.
6.7 Vorschläge
①Verringern Sie die Reaktionstemperatur (z. B. um 30 °C), beachten Sie jedoch, dass eine Verringerung der Reaktionstemperatur manchmal zu geringeren Erträgen führt.
②Versuchen Sie, die IVT-Reaktion mit anderen RNA-Polymerasen zu katalysieren.
③Wenn der GC-Gehalt der Vorlagen hoch ist, stellen Sie die IVT-Reaktionstemperatur auf 42 °C ein oder fügen Sie SSB in das IVT-Reaktionssystem ein, um die Ausbeute und die Länge der Transkripte zu erhöhen.
6.8 Es kommt zum Verschmieren der Transkripte während der Gelelektrophorese
Kommt es bei der Gelelektrophorese zum Verschmieren der Transkripte, kann das folgende Gründe haben:
①Während des Versuchsbetriebs kommt es zu einer RNase-Kontamination.
②DNA-Vorlagen sind durch RNasen kontaminiert.
6.9 Vorschläge
①Stellen Sie sicher, dass alle Reagenzien mit RNase-freiem H2O formuliert sind. Verwenden Sie RNase-freie Pipettenspitzen und Eppendorf-Röhrchen und tragen Sie während des Versuchsbetriebs Einweghandschuhe und -masken aus Latex.
②DNA-Vorlagen erneut reinigen.
7. Bestellinformationen
Nachfolgend sind repräsentative Produkte aufgeführt, die von
Wir bieten auch maßgeschneiderte Dienstleistungen an. Wenn Sie an einem Produkt interessiert sind, das nicht angezeigt wird, kontaktieren Sie uns und wir werden gemeinsam mit Ihnen Ihre Anforderungen erfüllen.
Tabelle 2.Produktinformationen
Zum Thema Lesen:
GMP-Reagenzien für die mRNA-In-vitro-Synthese
