Aprendizaje profundo de las principales materias primas enzimáticas utilizadas en la Detección del SARS-CoV-2 mediante PCR
Desde marzo de 2022, la astuta cepa mutante Omicron volvió a romper la vida pacífica de las personas, las epidemias del nuevo coronavirus estallaron en todo el país y afectaron a 30 provincias (regiones autónomas y municipios). Como medio eficaz de prevención y control precisos de la epidemia de SARS-CoV-2, la detección de ácidos nucleicos se ha convertido en una forma de vida normal. "¿Te hiciste la prueba de ácido nucleico hoy?" También se ha convertido en un saludo diario de la gente. Hablando de eso, ¿sabes qué materias primas básicas se necesitan en la detección de ácidos nucleicos? Este artículo presentará la materia prima básica crítica en la enzima de detección de ácidos nucleicos.
1. Proceso de detección de ácidos nucleicos para el SARS-CoV-2
2. Enzimas fundamentales en la extracción de ácidos nucleicos
3. Enzimas centrales durante la RT-qPCR
4. Enzimas centrales de la detección de ácidos nucleicos del SARS-CoV-2 de Yeasen
1. Proceso de detección de ácidos nucleicos para el SARS-CoV-2
Una enzima es una clase extremadamente importante de biocatalizadores con alta eficiencia catalítica y especificidad de reacción. La mayoría de las reacciones bioquímicas requieren la participación de enzimas. En el proceso de detección de ácidos nucleicos de 2019-nCoV (mostrado en la Figura 1), diferentes tipos de enzimas moleculares juegan un papel importante en diferentes etapas experimentales, como la extracción de ácidos nucleicos y la RT-qPCR. A continuación, de acuerdo con los diferentes vínculos experimentales en la detección de ácidos nucleicos, clasificaremos las materias primas enzimáticas básicas utilizadas en el proceso de detección de ácidos nucleicos.
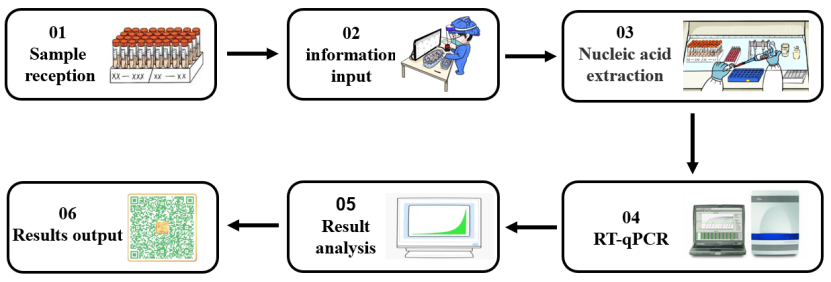
Figura 1. Proceso de detección de ácidos nucleicos para el SARS-CoV-2
2. Enzimas fundamentales en la extracción de ácidos nucleicos
El proceso de extracción de ácidos nucleicos del nuevo coronavirus incluye principalmente dos pasos: lisis y purificación. La lisis es el proceso de destrucción de la estructura celular de la muestra para que el ácido nucleico de la muestra quede libre en el sistema de lisis; la purificación es la separación completa del ácido nucleico de otros componentes en el sistema de lisis, como proteínas, sales y otras impurezas, y el proceso de reacción requiere la participación de la proteinasa K, la desoxirribonucleasa I y los inhibidores de la ARNasa.
2.1 Proteasa K
La proteinasa K es una serina proteasa con una amplia actividad de escisión; los sitios de escisión son los enlaces peptídicos carboxiterminales de aminoácidos alifáticos y aromáticos (Figura 2). En el proceso de extracción de ácidos nucleicos, la proteinasa K puede degradar las histonas que se unen fuertemente a los ácidos nucleicos, promover la separación de los ácidos nucleicos y hacer que la muestra de ácido nucleico sea más fácil de extraer. Además, la proteinasa K puede degradar la actividad de la ARN hidrolasa (ARNasa) e inhibir la hidrólisis de la ARN molde por ARNasa.
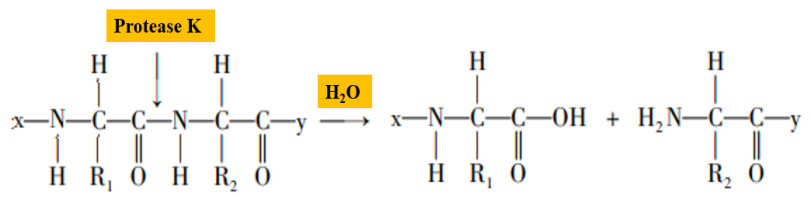
Figura 2. Diagrama esquemático de la proteinasa K que hidroliza enlaces peptídicos.
Biotecnología Yeasen Proteinasa K (Cat#10401ES) se deriva de una cepa de levadura recombinante, tiene una actividad específica ≥30 U/mg, no contiene ARNasa ni ADNasa y presenta una actividad enzimática estable en soluciones de urea y SDS. Es activa en un amplio rango de pH (pH 4,0~12,0), adecuada para la escisión y eliminación de proteínas, como la preparación de muestras de hibridación in situ y la purificación de ácidos nucleicos.
2.2 Desoxirribonucleasa I
La desoxirribonucleasa I (DNasa I) puede catalizar varias formas de ADN, apuntar a la escisión de enlaces fosfodiéster adyacentes a pirimidinas y generar polinucleótidos con un grupo fosfato en el extremo 5' y un grupo hidroxilo en el extremo 3', el producto de digestión promedio es el politetranucleótido más pequeño.En el proceso de extracción de ácido nucleico del SARS-CoV-2, la DNasa I se utiliza principalmente para eliminar la contaminación genómica en muestras de ARN, evitar residuos de ADN en las plantillas de ARN y mejorar la pureza de las plantillas.
Biotecnología Yeasen DNasa I (Cat#10325ES) se deriva de cepas de E. coli recombinantes, libre de ARNasa, y se puede utilizar para el tratamiento de varias muestras de ARN. El rango de pH de trabajo óptimo es de 7,0 a 8,0. En presencia de Mg2+, la ADNasa I puede escindir cualquier sitio de ADN bicatenario de forma aleatoria; mientras que en presencia de Mn2+La ADNasa I puede cortar ADN de doble cadena en el mismo sitio, formando extremos romos o extremos pegajosos con 1-2 nucleótidos salientes (que se muestran en la Figura 3).
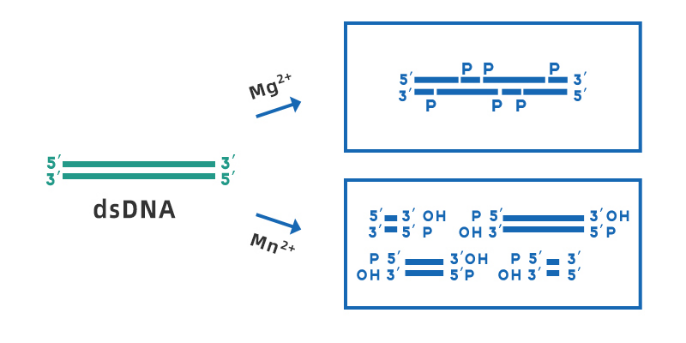
Fig. 3. Diagrama esquemático de la DNasa I que corta dsADN en presencia de Mg2+ y Mn2+
2.3 Inhibidor de la ARNasa
En el proceso de detección de ácidos nucleicos del SARS-CoV-2, la extracción y purificación de ácidos nucleicos de muestra o la preparación del sistema de reacción experimental pueden introducir contaminación por ribonucleasa (ARNasa), lo que provoca la degradación de la plantilla de ARN. Para evitar la contaminación por ARNasa, se requiere un inhibidor de ARNasa.
El inhibidor de la ARNasa es un inhibidor específico de la ARNasa en la placenta humana, que puede unirse específicamente a la ARNasa para formar un complejo con un enlace no covalente e inactivar la ARNasa. Yeasen Biotech Inhibidor de la ARNasa murina (Cat#10603ES) no contiene dos cisteínas que son muy sensibles a la oxidación en las proteínas humanas. Tiene una mayor actividad antioxidante y puede inhibir ampliamente varios tipos de ARNasas (ARNasa A, B, C), lo que lo hace más adecuado para experimentos sensibles a niveles altos de ditiotreitol (DTT), como la qPCR.
3. Enzimas centrales durante la RT-qPCR
Una vez finalizada la extracción de ácidos nucleicos de la muestra de SARS-CoV-2, se puede realizar la detección de ácidos nucleicos mediante RT-qPCR. En el proceso de estos experimentos, la ADN polimerasa, la transcriptasa inversa y la uracilo ADN glicosilasa son materias primas enzimáticas esenciales.
3.1 Transcriptasa inversa
Después de la extracción y purificación, el ARN del SARS-CoV-2 necesita una transcriptasa inversa para catalizar la polimerización de dNTP y generar una secuencia de ADNc complementaria al ARN molde (Figura 4). Para la reacción RT-qPCR, se debe seleccionar una transcriptasa inversa resistente a altas temperaturas. En la actualidad, la transcriptasa inversa MMLV es la más utilizada, debido a su falta de actividad endonucleasa de ADN y baja actividad de ARNasa H, tiene más ventajas en la aplicación de la clonación de ADNc.
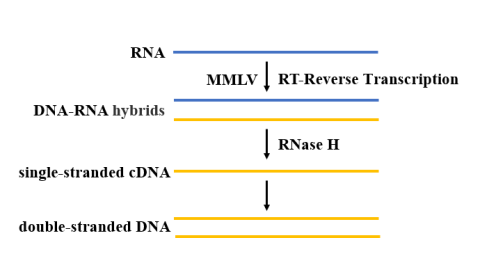
Figura 4. Diagrama esquemático del proceso de transcripción inversa.
Biotecnología Yeasen Hifair™ V Transcriptasa inversa (Cat#11300ES) es una nueva transcriptasa inversa obtenida mediante tecnología de ingeniería genética. Tiene buena estabilidad térmica y puede soportar temperaturas de reacción de hasta 60 °C. Y también es Adecuado para la transcripción inversa de plantillas de ARN con estructuras secundarias complejas. Al mismo tiempo, la enzima mejora la afinidad con la plantilla, es adecuada para la transcripción inversa de una pequeña cantidad de plantillas y genes de bajo número de copias, y puede amplificar ADNc de hasta 10 kb.
3.2 ADN polimerasa
Una vez completado el proceso de transcripción inversa de la plantilla para generar ADNc de doble cadena, se requiere que la ADN polimerasa "jugador del alma" en la reacción de PCR haga su aparición, polimerizando desoxirribonucleótidos libres para extender la cadena de ADN, y una gran cantidad de ADN plantilla se amplifica in vitro para lograr el propósito de la detección de ácidos nucleicos virales.
La ADN polimerasa que se utiliza habitualmente en las reacciones RT-qPCR es la ADN polimerasa Taq de arranque en caliente. Este tipo de enzima es inactiva a temperatura ambiente.Tiene actividad de polimerización solo después del inicio en caliente, lo que puede minimizar la generación de señales de fondo. Resuelve los problemas de amplificación no específica causada por la generación o desajuste de dímeros de cebador en las reacciones de PCR convencionales. En la actualidad, los métodos de modificación de inicio en caliente de la ADN polimerasa que se utilizan comúnmente incluyen principalmente la modificación química, la modificación de ligandos y la modificación de anticuerpos. Los principios de los diferentes métodos de modificación de inicio en caliente se muestran en la Figura 5.
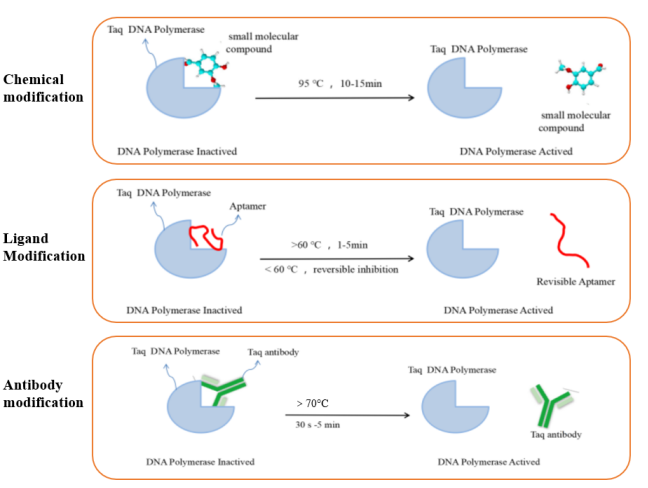
Figura 5. Diagrama esquemático de diferentes tipos de enzimas de arranque en caliente modificadas
Biotecnología Yeasen Polimerasa de ADN Taq de alta especificidad UNICONTM Hotstart, 5 U/μL (Cat#10726ES) es una ADN polimerasa de inicio en caliente de doble bloqueo con alta afinidad por la plantilla. A temperatura ambiente, no solo la región 5'→3' La actividad de la polimerasa está bloqueada, pero también el 5'→3' La actividad de la exonucleasa está bloqueada. La desnaturalización a 95 °C durante 30 segundos puede inactivar por completo el anticuerpo de bloqueo, lo que libera la actividad de la ADN polimerasa y la actividad de la exonucleasa. La función de doble bloqueo no solo puede prevenir eficazmente la amplificación no específica causada por desajustes o dímeros de cebadores, sino que también inhibe eficazmente la disminución de la señal de fluorescencia causada por la degradación de la sonda. La doble garantía hace que los reactivos de detección in vitro sean más estables durante el transporte o el uso a temperatura ambiente.
3.3 Uracilo ADN glicosilasa
En el proceso de detección de ácidos nucleicos del nuevo coronavirus, la contaminación por aerosoles en el entorno operativo es el factor más común que provoca resultados de PCR falsos positivos. La adición de la enzima UDG (uracilo ADN glicosilasa, uracilo ADN glicosilasa) al sistema de amplificación puede eliminar eficazmente los contaminantes residuales de amplificación (principalmente en forma de aerosoles) mezclados en el sistema de PCR para garantizar la precisión de los resultados de amplificación. El principio anticontaminación de la enzima UDG se muestra en la Figura 6.
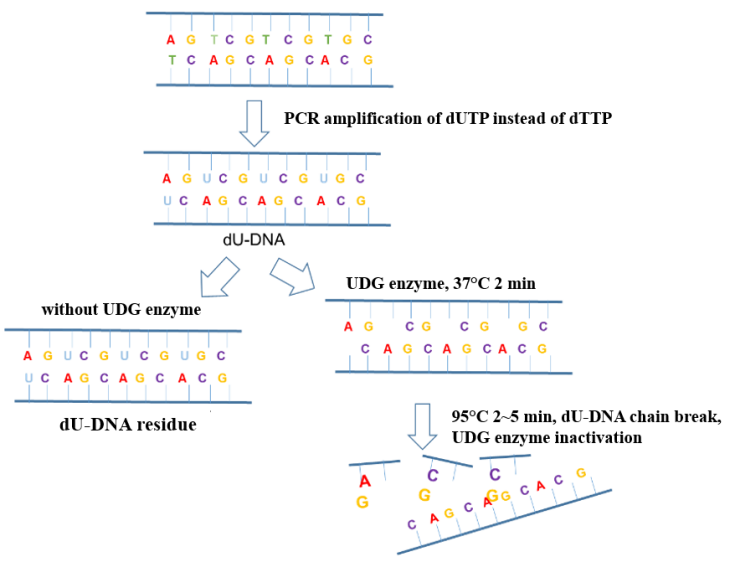
Figura 6. Diagrama esquemático del principio anticontaminación de la enzima UDG
Biotecnología Yeasen La uracilo ADN glicosilasa (UDG/UNG), termolábil, 1 U/μL (Cat#10303ES) es activa a 25-37 °C, sensible al calor y se inactiva irreversiblemente a 50 °C durante 10 min o 95 °C durante 2 min. No hay residuos de endonucleasa ni ARNasa, y el residuo del genoma de la bacteria huésped es inferior a 10 copias. Se puede utilizar junto con la ADN polimerasa Taq de arranque en caliente para garantizar la especificidad de la reacción de amplificación.
4.Enzimas centrales de la detección de ácidos nucleicos del SARS-CoV-2 de Yeasen
| Proceso | Descripción | Nombre del producto | Código SKU |
| Procesamiento de muestras | Digestión de proteínas | 10401ES | |
| Extracción de ARN | Recombinante DNasa I (Libre de ARNasa) (Consultar) | 10325ES | |
| Inhibición de la ARNasa | Inhibidor de la ARNasa murina (40 U/μL) | 10603ES | |
| Transcripción inversa | Adecuado para RT-qPCR | Hifair™ V Transcriptasa inversa (200 U/μL) | 11300ES |
| Transcriptasa inversa HifairTM V (600 U/μL) libre de glicerol (consultar) | 11301ES | ||
| Amplificación por PCR | ADN polimerasa de arranque en caliente | Polimerasa de ADN Taq de alta especificidad UNICONTM Hotstart | 10726ES |
| UDG Térmico | Uracilo ADN glicosilasa (UDG/UNG), termolábil, 1 U/μL | 10303ES |
Respecto a la lectura:
Selección de la transcriptasa inversa
YEASEN UDG termolábil: controla fácilmente la contaminación por aerosoles
Inhibidores de la ARNasa murina: eliminan con éxito la contaminación por ARNasa y preservan el ARN
