Apprentissage en profondeur des matières premières enzymatiques de base utilisées dans le Détection du SARS-CoV-2 par PCR
Depuis mars 2022, la souche mutante rusée d'Omicron a une fois de plus perturbé la vie paisible des gens, les épidémies de nouveau coronavirus ont éclaté dans tout le pays et ont touché 30 provinces (régions autonomes et municipalités). En tant que moyen efficace de prévention et de contrôle précis de l'épidémie de SRAS-CoV-2, la détection d'acide nucléique est devenue un mode de vie normal. « Avez-vous fait le test d'acide nucléique aujourd'hui ? » C'est également devenu une salutation quotidienne des gens. En parlant de cela, savez-vous quelles sont les matières premières de base nécessaires à la détection d'acide nucléique ? Cet article présentera la matière première de base essentielle de l'enzyme de détection d'acide nucléique.
1. Procédé de détection d'acide nucléique pour le SARS-CoV-2
2. Enzymes de base dans l'extraction des acides nucléiques
3. Enzymes de base pendant la RT-qPCR
4. Enzymes de base de la détection des acides nucléiques du SARS-CoV-2 par Yeasen
1. Procédé de détection des acides nucléiques du SARS-CoV-2
Une enzyme est une classe extrêmement importante de biocatalyseurs avec une efficacité catalytique et une spécificité de réaction élevées. La plupart des réactions biochimiques nécessitent la participation d'enzymes. Dans le processus de détection des acides nucléiques du 2019-nCoV (illustré dans la figure 1), différents types d'enzymes moléculaires jouent un rôle important dans différentes étapes expérimentales telles que l'extraction des acides nucléiques et la RT-qPCR. Ensuite, en fonction des différents liens expérimentaux dans la détection des acides nucléiques, nous trierons les matières premières enzymatiques de base utilisées dans le processus de détection des acides nucléiques.
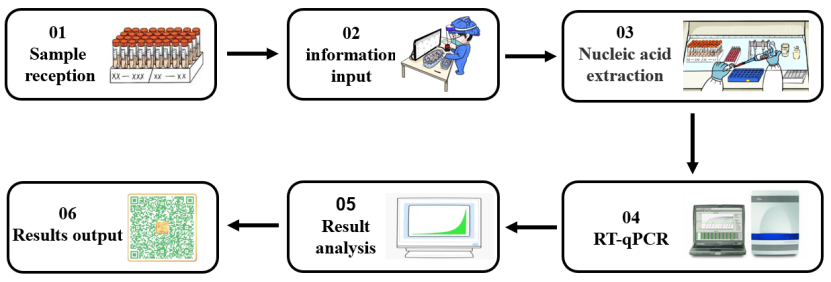
Figure 1. Processus de détection des acides nucléiques du SARS-CoV-2
2. Enzymes de base dans l'extraction des acides nucléiques
Le processus d'extraction d'acide nucléique du nouveau coronavirus comprend principalement deux étapes : la lyse et la purification. La lyse est le processus de destruction de la structure cellulaire de l'échantillon afin que l'acide nucléique de l'échantillon soit libre dans le système de lyse ; la purification est la séparation complète de l'acide nucléique des autres composants du système de lyse, tels que les protéines, le sel et d'autres impuretés, et le processus de réaction nécessite la participation de la protéinase K, de la désoxyribonucléase I et des inhibiteurs de la RNase.
2.1 Protéase K
La protéinase K est une sérine protéase à large activité de clivage, les sites de clivage étant les liaisons peptidiques carboxy-terminales des acides aminés aliphatiques et aromatiques (Figure 2). Au cours du processus d'extraction des acides nucléiques, la protéinase K peut dégrader les histones qui se lient étroitement aux acides nucléiques, favoriser la séparation des acides nucléiques et faciliter l'extraction des échantillons d'acide nucléique. De plus, la protéinase K peut dégrader l'activité de l'ARN hydrolase (RNase) et inhiber l'hydrolyse de l'ARN matrice par l'ARNase.
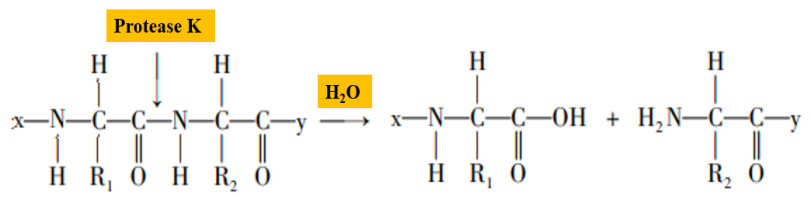
Figure 2. Schéma des liaisons peptidiques hydrolysées par la protéinase K
Yeasen Biotech Protéinase K (Cat#10401ES) est dérivé d'une souche de levure recombinante, activité spécifique ≥ 30 U/mg, exempte de RNase et de DNase, et activité enzymatique stable dans une solution d'urée et de SDS. Il est actif dans une large gamme de pH (pH 4,0~12,0), adapté au clivage et à l'élimination des protéines telles que la préparation d'échantillons d'hybridation in situ et la purification des acides nucléiques.
2.2 Désoxyribonucléase I
La désoxyribonucléase I (DNase I) peut catalyser diverses formes d'ADN, cibler le clivage des liaisons phosphodiester adjacentes aux pyrimidines et générer des polynucléotides avec un groupe phosphate à l'extrémité 5' et un groupe hydroxyle à l'extrémité 3', le produit de digestion moyen est le plus petit polytétranucléotide.Dans le processus d'extraction de l'acide nucléique du SRAS-CoV-2, la DNase I est principalement utilisée pour éliminer la contamination génomique dans les échantillons d'ARN, éviter les résidus d'ADN dans les modèles d'ARN et améliorer la pureté des modèles.
Yeasen Biotech ADNase I (Cat#10325ES) est dérivé de souches recombinantes d'E. coli, sans RNase, et peut être utilisé pour le traitement de divers échantillons d'ARN. La plage de pH de travail optimale est de 7,0 à 8,0. En présence de Mg2+, la DNase I peut cliver n'importe quel site d'ADN double brin de manière aléatoire ; tandis qu'en présence de Mn2+La DNase I peut cliver l'ADN double brin au même site, formant des extrémités franches ou des extrémités collantes surplombantes de 1 à 2 nucléotides (illustrées dans la figure 3).
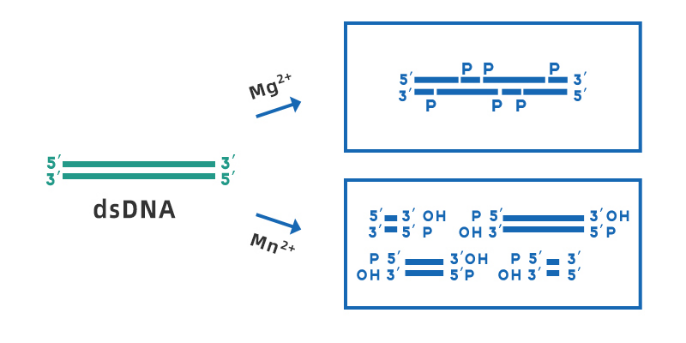
Fig 3. Schéma de la DNase I clivant l'ADN double brin en présence de Mg2+ et Mn2+
2.3 Inhibiteur de RNase
Lors de la détection de l'acide nucléique du SARS-CoV-2, l'extraction et la purification de l'échantillon d'acide nucléique ou la préparation du système de réaction expérimental peuvent entraîner une contamination par la ribonucléase (RNase), entraînant la dégradation de la matrice d'ARN. Pour éviter la contamination par la RNase, un inhibiteur de la RNase est nécessaire.
L'inhibiteur de la RNase est un inhibiteur spécifique de la RNase dans le placenta humain, qui peut se lier spécifiquement à la RNase pour former un complexe avec une liaison non covalente et inactiver la RNase. Yeasen Biotech Inhibiteur de RNase murine (Cat#10603ES) ne contient pas deux cystéines qui sont très sensibles à l'oxydation dans les protéines humaines. Il a une activité antioxydante plus élevée et peut largement inhiber divers types d'ARNases (ARNase A, B, C), plus adapté aux expériences sensibles au dithiothréitol élevé (DTT) telles que la qPCR.
3. Enzymes de base pendant la RT-qPCR
Une fois l'extraction de l'acide nucléique de l'échantillon de SARS-CoV-2 terminée, la détection de l'acide nucléique peut être réalisée par RT-qPCR. Au cours de ces expériences, l'ADN polymérase, la transcriptase inverse et l'uracile ADN glycosylase sont toutes des matières premières enzymatiques de base essentielles.
3.1 Transcriptase inverse
Après extraction et purification, l'ARN du SARS-CoV-2 a besoin de la transcriptase inverse pour catalyser la polymérisation des dNTP afin de générer une séquence d'ADNc complémentaire de l'ARN matrice (Figure 4). Pour la réaction RT-qPCR, il convient de sélectionner une transcriptase inverse résistante aux hautes températures. À l'heure actuelle, la transcriptase inverse MMLV est la plus largement utilisée, en raison de son absence d'activité endonucléase de l'ADN et de sa faible activité RNase H, elle présente davantage d'avantages dans l'application du clonage d'ADNc.
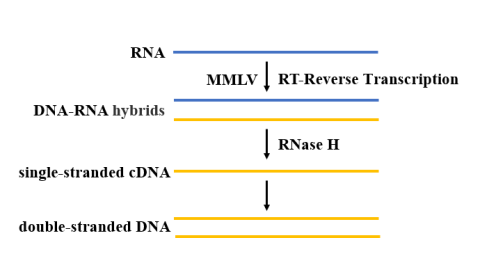
Figure 4. Schéma du processus de transcription inverse
Yeasen Biotech Hifair™ Transcriptase inverse V (Cat#11300ES) est une nouvelle transcriptase inverse obtenue par génie génétique. Elle présente une bonne stabilité thermique et peut supporter des températures de réaction allant jusqu'à 60°C. Convient à la transcription inverse de modèles d'ARN avec des structures secondaires complexes. En même temps, l'enzyme améliore l'affinité avec le modèle, convient à la transcription inverse d'un petit nombre de modèles et de gènes à faible nombre de copies, et peut amplifier l'ADNc jusqu'à 10 kb.
3.2 ADN polymérase
Une fois le processus de transcription inverse du modèle terminé pour générer l'ADNc double brin, l'ADN polymérase « joueur d'âme » dans la réaction de PCR doit faire son apparition, en polymérisant les désoxyribonucléotides libres pour étendre la chaîne d'ADN, et une grande quantité d'ADN modèle est amplifiée in vitro pour atteindre l'objectif de détection d'acide nucléique viral.
L'ADN polymérase couramment utilisée dans les réactions de RT-qPCR est l'ADN polymérase Taq à démarrage à chaud. Ce type d'enzyme est inactif à température ambiante.L'activité de polymérisation ne se produit qu'après un démarrage à chaud, ce qui peut minimiser la génération de signaux de fond. Elle résout les problèmes d'amplification non spécifique causés par la génération d'amorces-dimères ou par l'inadéquation dans les réactions de PCR conventionnelles. À l'heure actuelle, les méthodes de modification par démarrage à chaud de l'ADN polymérase couramment utilisées comprennent principalement la modification chimique, la modification du ligand et la modification de l'anticorps. Les principes des différentes méthodes de modification par démarrage à chaud sont présentés dans la figure 5.
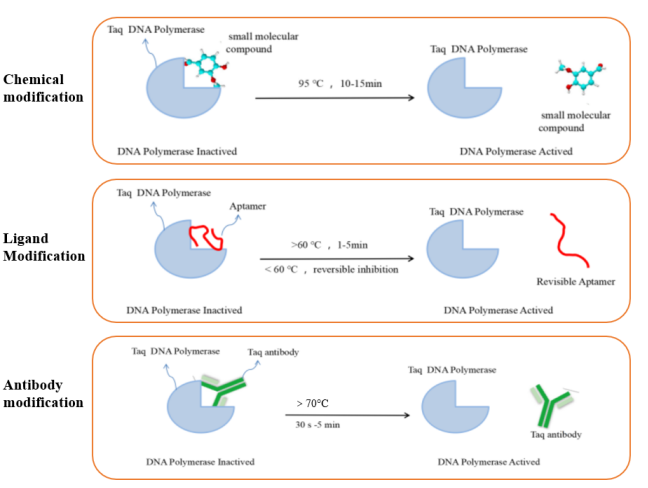
Figure 5. Schéma de principe de différents types d'enzymes de démarrage à chaud modifiées
Yeasen Biotech ADN polymérase Taq hautement spécifique à démarrage à chaud UNICONTM, 5 U/μL (Cat#10726ES) est une ADN polymérase à démarrage à chaud à double blocage avec une affinité élevée pour la matrice. À température ambiante, non seulement la séquence 5'→3' l'activité polymérase est bloquée, mais aussi le 5'→3' L'activité exonucléase est bloquée. La dénaturation à 95°C pendant 30 secondes peut complètement inactiver l'anticorps bloquant, libérant ainsi l'activité ADN polymérase et l'activité exonucléase. La fonction de double blocage peut non seulement empêcher efficacement l'amplification non spécifique causée par des mésappariements ou des dimères d'amorces, mais également inhiber efficacement la diminution du signal de fluorescence causée par la dégradation de la sonde. La double garantie rend les réactifs de détection in vitro plus stables pendant le transport ou l'utilisation à température ambiante.
3.3 Uracile ADN glycosylase
Dans le processus de détection des acides nucléiques du nouveau coronavirus, la pollution par les aérosols dans l'environnement d'exploitation est le facteur le plus courant à l'origine de résultats PCR faussement positifs. L'ajout de l'enzyme UDG (Uracil DNA Glycosylase, uracile DNA glycosylase) au système d'amplification peut éliminer efficacement les polluants résiduels d'amplification (principalement sous forme d'aérosols) mélangés dans le système PCR pour garantir l'exactitude des résultats d'amplification. Le principe anti-pollution de l'enzyme UDG est illustré à la figure 6.
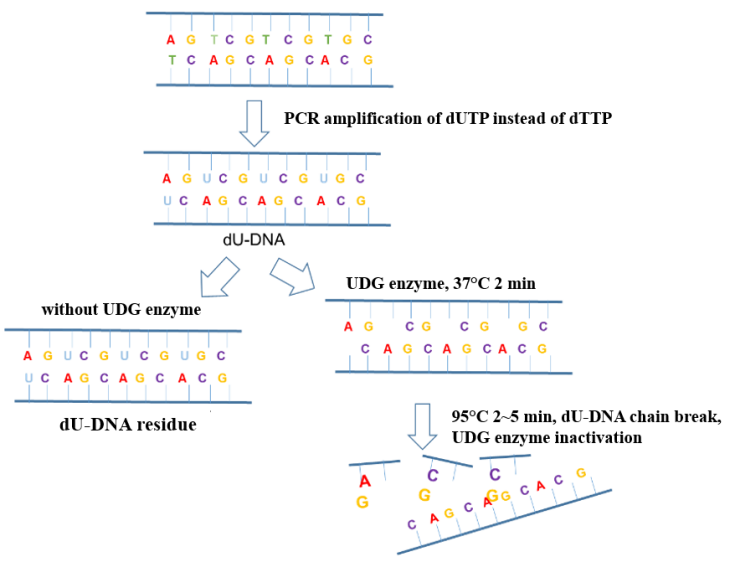
Figure 6. Schéma du principe antipollution de l'enzyme UDG
Yeasen Biotech L'Uracil DNA Glycosylase (UDG/UNG), thermolabile, 1 U/μL (Cat#10303ES) est active à 25-37°C, sensible à la chaleur et inactivée de manière irréversible à 50°C pendant 10 min ou 95°C pendant 2 min. Il n'y a pas de résidu d'endonucléase et d'ARNase, et le résidu du génome de la bactérie hôte est inférieur à 10 copies. Il peut être utilisé en conjonction avec l'ADN polymérase Taq à démarrage à chaud pour garantir la spécificité de la réaction d'amplification.
4.Enzymes de base de la détection des acides nucléiques du SARS-CoV-2 par Yeasen
| Processus | Description | Nom du produit | UGS |
| Traitement des échantillons | Digestion des protéines | 10401ES | |
| Extraction d'ARN | Recombinant ADNase I (Sans RNase) (Renseignez-vous) | 10325ES | |
| Inhibition de la RNase | Inhibiteur de RNase murine (40 U/μL) | 10603ES | |
| Transcription inverse | Adapté à la RT-qPCR | Hifair™ Transcriptase inverse V (200 U/µL) | 11300ES |
| Transcriptase inverse HifairTM V (600 U/μL) sans GLyceroL (Renseignez-vous) | 11301ES | ||
| Amplification par PCR | ADN polymérase à démarrage à chaud | ADN polymérase Taq hautement spécifique à démarrage à chaud UNICONTM | 10726ES |
| UDG thermique | Uracile ADN glycosylase (UDG/UNG), thermolabile, 1 U/μL | 10303ES |
Concernant la lecture :
Sélection de la transcriptase inverse
YEASEN UDG thermolabile — Contrôlez facilement la pollution par aérosol
Inhibiteurs de RNase murine - Éliminent avec succès la contamination par la RNase et préservent l'ARN
