Aprendizagem profunda das principais matérias-primas enzimáticas utilizadas no Detecção de PCR do SARS-CoV-2
Desde março de 2022, a astuta cepa mutante Ômicron mais uma vez quebrou a vida pacífica das pessoas, as novas epidemias de coronavírus eclodiram por todo o país e afetaram 30 províncias (regiões autônomas e municípios). Como um meio eficaz de prevenção e controle precisos da epidemia de SARS-CoV-2, a detecção de ácido nucleico se tornou um modo de vida normal. "Você fez o teste de ácido nucleico hoje?" Também se tornou uma saudação diária das pessoas. Falando nisso, você sabe quais são as principais matérias-primas necessárias na detecção de ácido nucleico? Este artigo apresentará a principal matéria-prima crítica na enzima de detecção de ácido nucleico.
1. Processo de detecção de ácido nucleico para SARS-CoV-2
2. Enzimas essenciais na extração de ácido nucleico
3. Enzimas centrais durante RT-qPCR
4. Enzimas essenciais da detecção de ácido nucleico do SARS-CoV-2 a partir de
1. Processo de detecção de ácido nucleico para SARS-CoV-2
Uma enzima é uma classe extremamente importante de biocatalisadores com alta eficiência catalítica e especificidade de reação. A maioria das reações bioquímicas requer a participação de enzimas. No processo de detecção de ácido nucleico de 2019-nCoV (mostrado na Figura 1), diferentes tipos de enzimas moleculares desempenham um papel importante em diferentes estágios experimentais, como extração de ácido nucleico e RT-qPCR. Em seguida, de acordo com os diferentes links experimentais na detecção de ácido nucleico, classificaremos as principais matérias-primas enzimáticas usadas no processo de detecção de ácido nucleico.
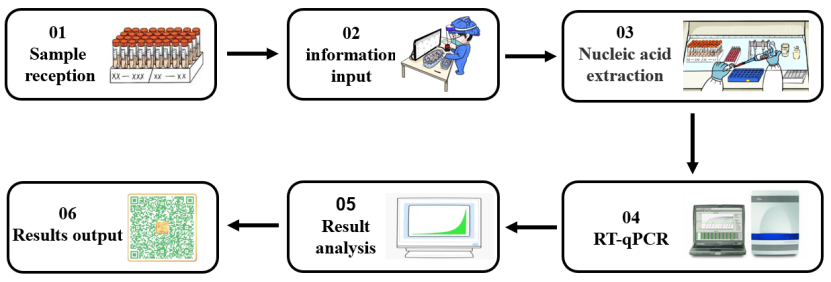
Figura 1. Processo de detecção de ácido nucleico para SARS-CoV-2
2. Enzimas essenciais na extração de ácidos nucleicos
O processo de extração de ácido nucleico do novo coronavírus inclui principalmente duas etapas: lise e purificação. Lise é o processo de destruição da estrutura celular da amostra para que o ácido nucleico na amostra fique livre no sistema de lise; Purificação é a separação completa do ácido nucleico de outros componentes no sistema de lise, como proteína, sal e outras impurezas, e o processo de reação requer a participação de inibidores de proteinase K, desoxirribonuclease I e RNase.
2.1 Protease K
Proteinase K é uma serina protease com ampla atividade de clivagem, os locais de clivagem são as ligações peptídicas carboxi-terminais de aminoácidos alifáticos e aromáticos (Figura 2). No processo de extração de ácido nucleico, a proteinase K pode degradar histonas que se ligam fortemente aos ácidos nucleicos, promovem a separação de ácidos nucleicos e tornam o ácido nucleico da amostra mais fácil de extrair. Além disso, a proteinase K pode degradar a atividade da RNA hidrolase (RNase) e inibir a hidrólise da RNase do RNA molde.
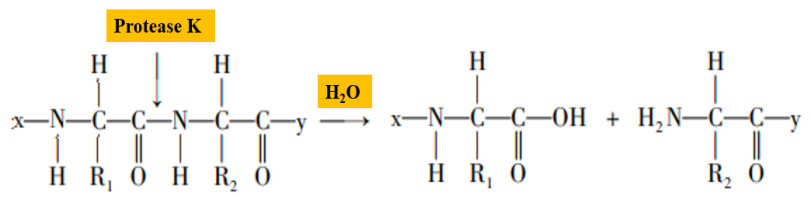
Figura 2. Diagrama esquemático da proteinase K hidrolisando ligações peptídicas
2.2 Desoxirribonuclease I
A desoxirribonuclease I (DNase I) pode catalisar várias formas de DNA, ter como alvo a clivagem de ligações fosfodiéster adjacentes às pirimidinas e gerar polinucleotídeos com um grupo fosfato na extremidade 5' e um grupo hidroxila na extremidade 3'; o produto médio da digestão é o menor politetranucleotídeo.No processo de extração de ácido nucleico do SARS-CoV-2, a DNase I é usada principalmente para remover contaminação genômica em amostras de RNA, evitar resíduos de DNA em modelos de RNA e melhorar a pureza dos modelos.
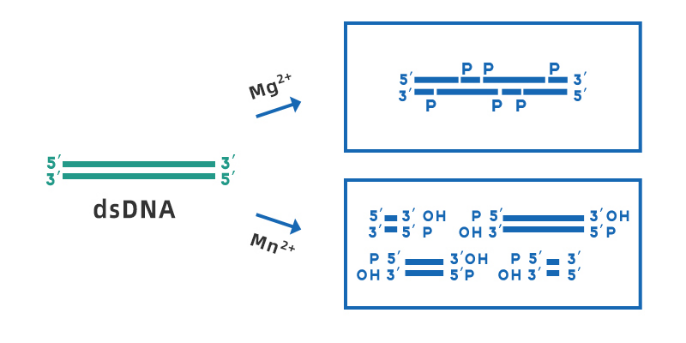
Fig 3. Diagrama esquemático da DNase I clivando dsDNA na presença de Mg2+ e Mn2+
2.3 Inibidor de RNase
No processo de detecção de ácido nucleico do SARS-CoV-2, a extração e purificação do ácido nucleico da amostra ou a preparação do sistema de reação experimental pode introduzir contaminação por ribonuclease (RNase), resultando na degradação do molde de RNA. Para evitar contaminação por RNase, o Inibidor de RNase é necessário.
O inibidor de RNase é um inibidor de RNase específico na placenta humana, que pode ligar-se especificamente à RNase para formar um complexo com uma ligação não covalente e inativar a RNase.
3. Enzimas centrais durante RT-qPCR
Após a extração de ácido nucleico da amostra de SARS-CoV-2 ser concluída, a detecção de ácido nucleico pode ser concluída por RT-qPCR. No processo desses experimentos, a DNA polimerase, a transcriptase reversa e a uracil DNA glicosilase são todas matérias-primas essenciais da enzima central.
3.1 Transcriptase reversa
Após a extração e purificação, o RNA do SARS-CoV-2 precisa da transcriptase reversa para catalisar a polimerização do dNTP para gerar uma sequência de cDNA complementar ao RNA molde (Figura 4). Para a reação de RT-qPCR, a transcriptase reversa resistente a altas temperaturas deve ser selecionada. Atualmente, a transcriptase reversa do MMLV é a mais amplamente usada, devido à sua falta de atividade de endonuclease de DNA e baixa atividade de RNase H, ela tem mais vantagens na aplicação da clonagem de cDNA.
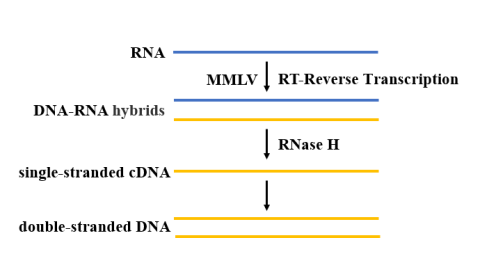
Figura 4. Diagrama esquemático do processo de transcrição reversa
3.2 DNA polimerase
Após a conclusão do processo de transcrição reversa do modelo para gerar cDNA de fita dupla, a DNA polimerase "soul player" na reação de PCR precisa aparecer, polimerizando desoxirribonucleotídeos livres para estender a cadeia de DNA, e uma grande quantidade de DNA modelo é amplificada in vitro para atingir o objetivo de detecção de ácido nucleico viral.
A DNA polimerase comumente usada em reações de RT-qPCR é a Taq DNA polimerase hot-start. Esse tipo de enzima é inativa à temperatura ambiente.Ele tem atividade de polimerização somente após partida a quente, o que pode minimizar a geração de sinais de fundo. Ele resolve os problemas de amplificação não específica causada pela geração de primer-dímero ou incompatibilidade em reações de PCR convencionais. Atualmente, os métodos de modificação de partida a quente da DNA polimerase comumente usados incluem principalmente modificação química, modificação de ligante e modificação de anticorpo. Os princípios de diferentes métodos de modificação de partida a quente são mostrados na Figura 5.
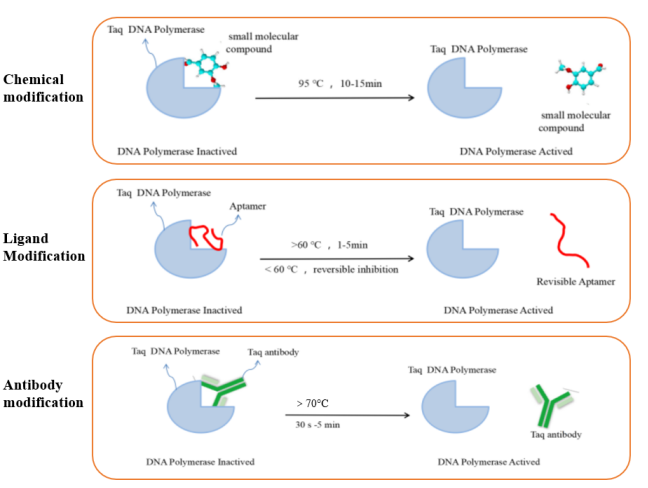
Figura 5. Diagrama esquemático de diferentes tipos de enzimas de partida a quente modificadas
3.3 Uracila DNA glicosilase
No processo de detecção de ácido nucleico do novo coronavírus, a poluição por aerossol no ambiente operacional é o fator mais comum que causa resultados falso-positivos de PCR. Adicionar a enzima UDG (Uracil DNA Glycosylase, uracil DNA glycosylase) ao sistema de amplificação pode efetivamente eliminar os poluentes residuais de amplificação (principalmente na forma de aerossóis) misturados no sistema de PCR para garantir a precisão dos resultados de amplificação. O princípio antipoluição da enzima UDG é mostrado na Figura 6.
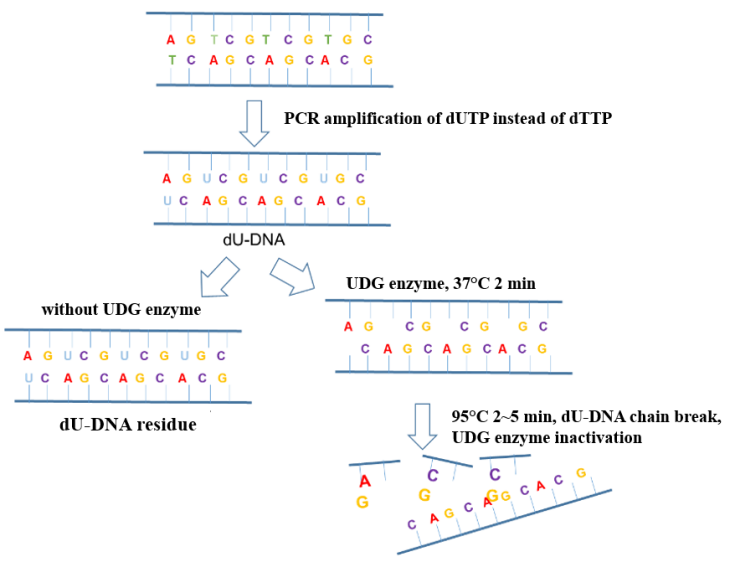
Figura 6. Diagrama esquemático do princípio antipoluição da enzima UDG
4.Enzimas essenciais da detecção de ácido nucleico do SARS-CoV-2 a partir de Yeasen
| Processo | Descrição | Nome do produto | SKU |
| Processamento de amostra | Digestão de proteínas | 10401ES | |
| Extração de RNA | Recombinante DNase I (Sem RNase) (Consultar) | 10325ES | |
| Inibição de RNase | Inibidor de RNase murina (40U/ μL) | 10603ES | |
| Transcrição reversa | Adequado para RT-qPCR | Hifair™ V Transcriptase Reversa (200U/ μL) | 11300ES |
| HifairTM V Reverse Transcriptase (600U/ μL) GlyceroL- livre (Consultar) | 11301ES | ||
| Amplificação por PCR | DNA polimerase de partida a quente | 10726ES | |
| UDG térmico | Uracil DNA glicosilase (UDG/UNG), termolábil, 1 U/μL | 10303ES |
Sobre a leitura:
Seleção de Transcriptase Reversa
YEASEN UDG termolábil ——Controla facilmente a poluição por aerossóis
Inibidores de RNase murina - eliminam com sucesso a contaminação de RNase e preservam o RNA
