เทคโนโลยีการจัดลำดับยีนประสิทธิภาพสูง ซึ่งมักเรียกกันว่าเทคโนโลยีการจัดลำดับยีนรุ่นถัดไป (NGS) ถือเป็นก้าวกระโดดครั้งสำคัญจากวิธีการจัดลำดับยีน DNA ขั้นต้น เช่น การจัดลำดับยีนแซงเกอร์ เทคโนโลยี NGS ช่วยให้สามารถจัดทำโปรไฟล์ของลำดับโมเลกุลกรดนิวคลีอิกได้หลายแสนหรือหลายล้านลำดับพร้อมกัน ข้อดีของเทคโนโลยี NGS ได้แก่ ปริมาณงานที่ยอดเยี่ยม ความคุ้มทุน ความสามารถในการปรับขนาด และการใช้งานที่หลากหลาย ทำให้เทคโนโลยี NGS เป็นเทคโนโลยีการจัดลำดับยีนที่โดดเด่นที่สุดในโลก
ขั้นตอนการจัดลำดับ NGS ประกอบด้วยขั้นตอนหลัก 4 ขั้นตอน ได้แก่ การเตรียมตัวอย่าง การสร้างไลบรารี การจัดลำดับ และการวิเคราะห์ข้อมูล สิ่งสำคัญในการสร้างไลบรารีคือการแนบลำดับอะแดปเตอร์แพลตฟอร์ม NGS ที่ได้มาตรฐานเข้ากับปลายทั้งสองด้านของดีเอ็นเอจีโนมที่แตกเป็นชิ้นเล็กชิ้นน้อย ขั้นตอนนี้มุ่งหวังที่จะสร้างโมเลกุลกรดนิวคลีอิกในไลบรารีในปริมาณมาก ซึ่งเตรียมไว้สำหรับการจัดลำดับบนเครื่องมือ NGS ผ่านการขยาย PCR การสร้างไลบรารี NGS สามารถแบ่งประเภทได้เป็นการสร้างไลบรารี DNA และการสร้างไลบรารี RNA ขึ้นอยู่กับลักษณะของตัวอย่าง เอนไซม์มีบทบาทสำคัญในการทดลองที่เชื่อมโยงกันเหล่านี้ แล้วเอนไซม์หลักตัวใดที่เกี่ยวข้องกับกระบวนการสร้างไลบรารี?
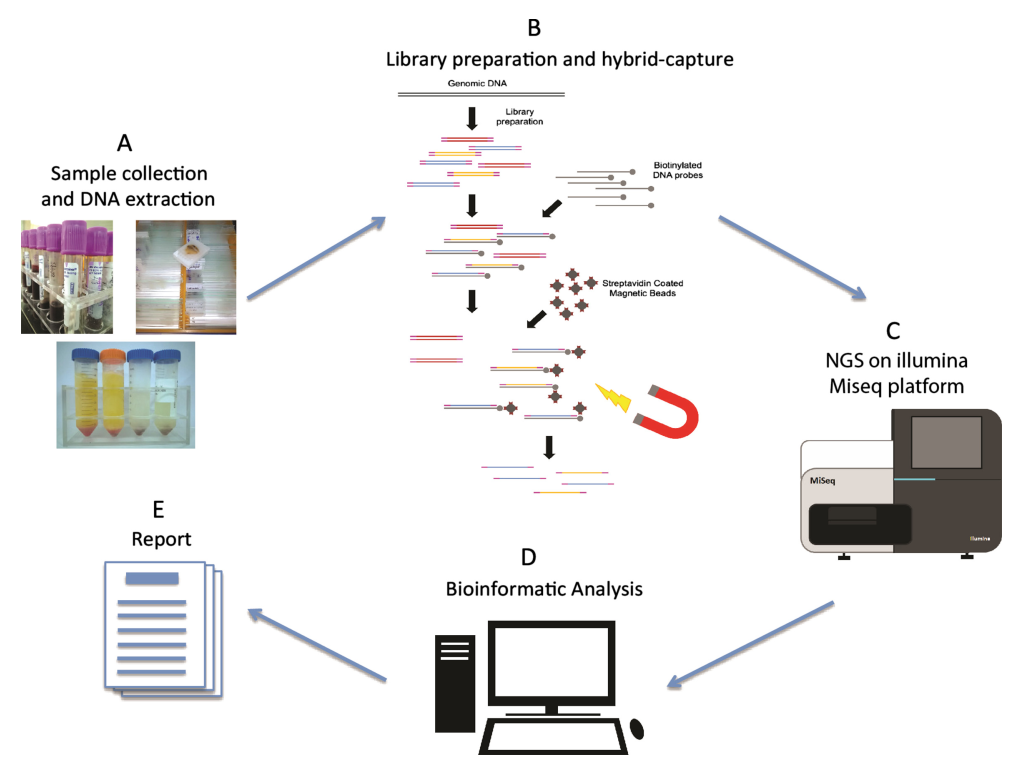
รูปที่ 1 เวิร์กโฟลว์การจัดลำดับรุ่นถัดไป[2]
1. การสร้างห้องสมุด DNA และเอนไซม์สำคัญ
2. การสร้างห้องสมุด RNA และเอนไซม์หลัก
3. แนวทางการใช้เอ็นไซม์หลัก NGS ในการสร้างไลบรารี DNA และ RNA
1. การสร้างห้องสมุด DNA และเอนไซม์สำคัญ
ในกระบวนการสร้างห้องสมุด DNA การสร้างห้องสมุดอะแดปเตอร์ผูกโคลน TA ถือเป็นเทคโนโลยีที่ใช้กันแพร่หลายที่สุดในปัจจุบัน กระบวนการสร้างห้องสมุดหลักมีดังนี้:

รูปที่ 2 กระบวนการสร้างห้องสมุด DNA (Illumina)
1.1 การแตกตัวของดีเอ็นเอ
เครื่องเรียงลำดับปัจจุบันโดยทั่วไปมีความยาวของการจัดลำดับอยู่ในช่วง 150-500 คู่เบส (bp) ดังนั้นจึงจำเป็นต้องใช้วิธีการแบ่งส่วนทางกลหรือทางเอนไซม์เพื่อแยกส่วนดีเอ็นเอจีโนมขนาดใหญ่ให้มีขนาดเล็กลง การแตกส่วนทางกลอาจทำให้สูญเสียตัวอย่างค่อนข้างมาก และต้องมีกระบวนการทำงานที่ซับซ้อนกว่า ในทางกลับกัน การย่อยด้วยเอนไซม์เป็นวิธีที่ใช้กันทั่วไปในการแยกส่วนดีเอ็นเอจีโนม เมื่อเปรียบเทียบกับวิธีการทางกล การย่อยด้วยเอนไซม์จะคุ้มทุนกว่าและตรงไปตรงมามากกว่า โดยปฏิกิริยาจะใช้เวลาเพียงระยะเวลาหนึ่งหลังจากเติมเอนไซม์สำหรับการแยกส่วนเท่านั้น
ปัจจุบัน มีชิ้นส่วนที่ใช้อยู่สองประเภทหลัก ประเภทหนึ่งอาศัยทรานสโพเซส Tn5 ตามหลักการทรานสโพซอน ในขณะที่อีกประเภทหนึ่งใช้เอนโดนิวคลีเอสผสมกัน อย่างไรก็ตาม ประสิทธิภาพของชิ้นส่วนเหล่านี้อาจได้รับอิทธิพลจากปริมาณ GC และความชอบเบสของ DNA ในทางตรงกันข้าม ชิ้นส่วนที่พัฒนาโดย
1.2 การซ่อมแซมส่วนท้าย, dA-Tailing
DNA ที่แตกเป็นชิ้นเล็กชิ้นน้อยจะสร้างปลายเหนียวขนาด 5'/3' และ DNA ปลายทู่ และปลายเหนียวทั้งหมดจะต้องถูกแปลงให้เป็นปลายทู่ รวมถึงส่วนที่ยื่นออกมา 3' ที่ถูกเอาออกและปลาย DNA ที่ยื่นออกมา 5' ที่เติมเข้าไป เมื่อใช้การผูก TA สำหรับการผูกอะแดปเตอร์ ชิ้นส่วน DNA จะต้องถูกฟอสโฟรีเลตที่ปลาย 5' ด้วย และเพิ่ม "A" ที่ปลาย 3' เพื่อให้เป็นส่วนเสริมของอะแดปเตอร์ที่มีปลายเหนียว "T"กระบวนการข้างต้นเสร็จสมบูรณ์ด้วยความร่วมมือของ T4 DNA polymerase, T4 polynucleotide kinase และ ตัก ดีเอ็นเอโพลีเมอเรส
โพลิเมอเรส DNA T4 (Cat#12901) มีกิจกรรมโพลีเมอเรสของดีเอ็นเอ 5'→3' ซึ่งสามารถเร่งปฏิกิริยาการสังเคราะห์ดีเอ็นเอตามทิศทาง 5'→3' และเติมเต็มปลายที่ยื่นออกมา 5' ในเวลาเดียวกัน เอนไซม์ยังมีกิจกรรมเอ็กโซนิวคลีเอส 3'→5' เพื่อแยกปลายที่ยื่นออกมา 3' จึงเปลี่ยนชิ้นส่วนดีเอ็นเอที่มีปลายเหนียวเป็นดีเอ็นเอปลายทู่
เนื่องจากปลาย 5' ของไพรเมอร์และอะแดปเตอร์ PCR สังเคราะห์มักเป็นกลุ่มไฮดรอกซิลแทนที่จะเป็นกลุ่มฟอสเฟต ดังนั้น จึงต้องใช้ไคเนสโพลีนิวคลีโอไทด์ T4 (Cat#12902) เพื่อเร่งปฏิกิริยาการถ่ายโอนกลุ่มฟอสเฟต ATP γ ไปยังปลาย 5' ไฮดรอกซิลของโซ่โอลิโกนิวคลีโอไทด์ในสถานะที่มี ATP เพื่อเตรียมพร้อมสำหรับขั้นตอนต่อไปของการผูกอะแดปเตอร์
เอส-แท็ก ดีเอ็นเอโพลีเมอเรส (Cat#13486) มีกิจกรรมโพลีเมอเรส 5’→3' ซึ่งสามารถสังเคราะห์ DNA จากทิศทาง 5’→3' ในขณะเดียวกัน ยังมีกิจกรรมดีออกซีไรโบนิวคลีโอไทด์ทรานสเฟอเรสซึ่งสามารถเพิ่มนิวคลีโอไทด์ "A" ลงในปลาย 3' ของผลิตภัณฑ์ PCR

รูปที่ 3 เอนไซม์หลายชนิดมีส่วนเกี่ยวข้องในกระบวนการซ่อมแซมขั้นสุดท้าย
รูปที่ 4 S-taq มีประสิทธิภาพการเติม A ให้กับเบสทั้งสี่ของ ATCG ของส่วนยีนปลาย 3' ที่ตรวจพบโดยอิเล็กโทรโฟรีซิสแบบเส้นเลือดฝอยสูงมาก
1.3 การผูกอะแดปเตอร์
อะแดปเตอร์ถือเป็นส่วนประกอบสำคัญของไลบรารี ภายในขอบเขตของการจัดลำดับ Illumina อะแดปเตอร์ประเภท Y ที่ใช้กันทั่วไปประกอบด้วยลำดับ SP P5/P7, Index และ Rd1/Rd2 ในจำนวนนี้ ลำดับ P5/P7 ทำหน้าที่จับคู่กับลำดับที่มีอยู่ในชิปการจัดลำดับ จึงยึดชิ้นส่วนที่จะวิเคราะห์ไว้กับเซลล์ไหลเพื่อดำเนินการขยายสะพาน ลำดับ Index ใช้เพื่อแยกความแตกต่างระหว่างตัวอย่างต่างๆ ภายในไลบรารีผสมที่จัดลำดับ ในขณะที่ SP Rd1/Rd2 แสดงถึงบริเวณสำหรับจับไพรเมอร์การจัดลำดับ Read1 และ Read2
สำหรับงานผูกอะแดปเตอร์ เอนไซม์ไลเกส DNA T4 (Cat#12996) เป็นตัวเลือกมาตรฐาน แสดงให้เห็นถึงความสามารถในการซ่อมแซมรอยบากสายเดี่ยวที่พบในดีเอ็นเอสายคู่และเชื่อมต่อนิวคลีโอไทด์ที่อยู่ติดกันอีกครั้ง
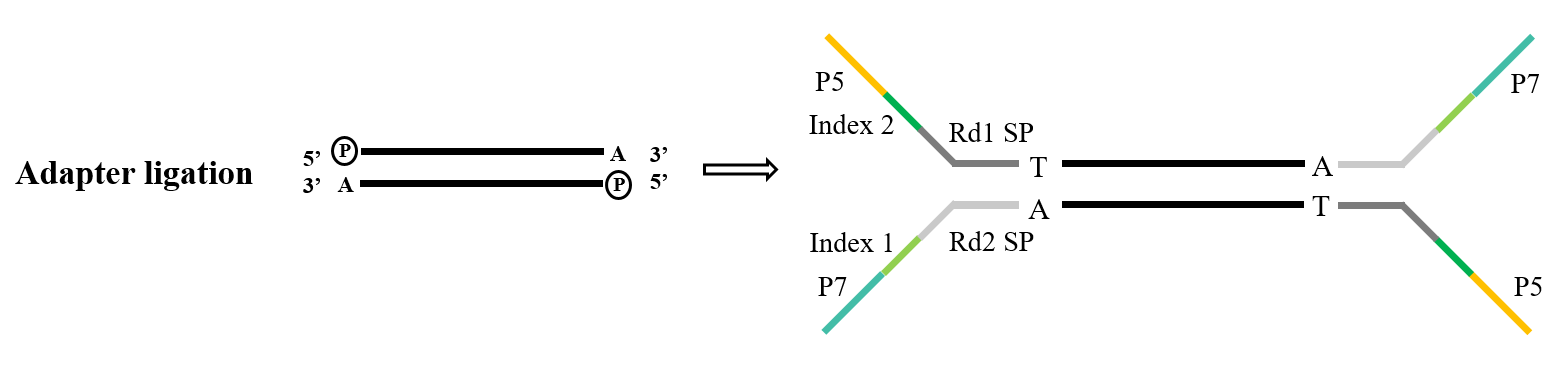
รูปที่ 5 กระบวนการเชื่อมต่ออะแดปเตอร์ทั่วไป (Illumina)

รูปที่ 6 การตรวจสอบยีนกลายพันธุ์ DNA Ligase T4 โดยการเชื่อมต่อ DNA ขนาด 170 คู่เบสกับอะแดปเตอร์ขนาด 80 คู่เบส
1.4 การขยาย PCR
รับลำดับดีเอ็นเอที่เพียงพอด้วยอะแดปเตอร์ผ่านปฏิกิริยา PCR และทำการจัดลำดับกรดนิวคลีอิกตัวอย่างให้เสร็จสมบูรณ์บนเครื่อง Hieff Canaceทีเอ็ม DNA โพลิเมอเรสแบบ Pro High-Fidelity (Cat#13476) ที่ใช้กันทั่วไปใน PCR มีกิจกรรมของโพลิเมอเรส 5'→3' และสามารถสังเคราะห์ DNA ในทิศทาง 5'→3' ได้ นอกจากนี้ ยังมีกิจกรรมของเอ็กโซนิวคลีเอส 3'→5' ซึ่งสามารถแก้ไขการรวมเบสที่ไม่ถูกต้องระหว่างกระบวนการขยาย เพื่อขยายชิ้นส่วน DNA อย่างรวดเร็วและมีความเที่ยงตรงสูง
2. การสร้างห้องสมุด RNA และเอนไซม์หลัก
การสร้างคลังข้อมูล RNA สามารถแบ่งออกได้เป็น คลังข้อมูล mRNA คลังข้อมูล LncRNA เป็นต้น โดยคลังข้อมูล RNA ทั่วไปประกอบด้วยกระบวนการต่างๆ ดังต่อไปนี้:
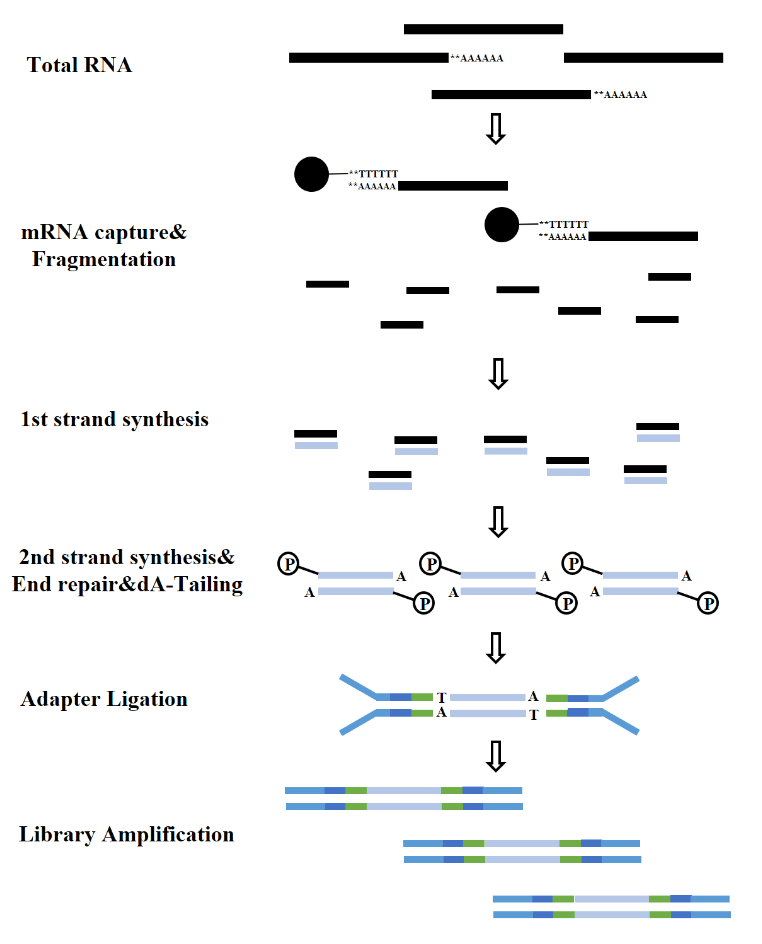
รูปที่ 7 กระบวนการสร้างคลัง mRNA (Illumina)
2.1. การเสริมสมรรถนะ RNA
ไม่ว่าจะเกี่ยวข้องกับยูคาริโอตหรือโพรคาริโอต RNA ไรโบโซม (rRNA) ถือเป็น RNA ที่มีมากที่สุด โดยคิดเป็น 80% ของเนื้อหา RNA ทั้งหมด เมื่อจัดลำดับ RNA ทั้งหมดของตัวอย่างโดยตรง ข้อมูลการจัดลำดับส่วนใหญ่จะต้องเกี่ยวข้องกับ rRNA เพื่อลดการรบกวนนี้ จำเป็นต้องใช้วิธีการเพิ่มความเข้มข้นของ RNA โดยมีวิธีการหลักสองวิธีสำหรับเรื่องนี้ ได้แก่ การเสริมความเข้มข้นของ mRNA โดยอาศัยวิธีการ oligo-dT และวิธีการลดความเข้มข้นของ rRNA
ในยูคาริโอต mRNA จะแสดงโครงสร้างโพลี(A) ที่แตกต่างกันที่ปลาย 3' ลูกปัดโอลิโก-dT สามารถนำมาใช้เพื่อจับ mRNA ทั้งหมดที่ถอดรหัสจากตัวอย่าง ทำให้เหมาะสำหรับการวิเคราะห์การถอดรหัส โดยเฉพาะกับตัวอย่าง RNA คุณภาพสูง ในทางกลับกัน วิธีการกำจัด rRNA มีข้อกำหนดที่ผ่อนปรนกว่าสำหรับคุณภาพของตัวอย่างและสามารถนำไปใช้กับตัวอย่างคุณภาพต่ำ (เช่น ตัวอย่าง FFPE) และตัวอย่าง RNA คุณภาพสูง รวมถึงตัวอย่างโพรคาริโอต วิธีการทางการค้าที่ใช้กันทั่วไปเกี่ยวข้องกับการใช้การย่อยด้วย RNase H เพื่อกำจัด rRNA โดยปฏิบัติตามขั้นตอนเฉพาะเหล่านี้:
- สังเคราะห์โพรบโอลิโคนิวคลีโอไทด์เฉพาะที่ออกแบบมาเพื่อจับกับ rRNA
- ใช้ RNase H (Cat#12906) ซึ่งมีความสามารถในการย่อยสลาย RNA ในสายไฮบริด RNA-DNA เพื่อกำจัด rRNA ที่ผูกกับโพรบอย่างเลือกสรร
- สุดท้ายย่อยโพรบ DNA ด้วย ดีเอ็นเอส ไอ (Cat#10325) ซึ่งสามารถย่อยสลายดีเอ็นเอสายเดี่ยวและสายคู่ได้อย่างมีประสิทธิภาพ โดยกำจัด rRNA ได้อย่างมีประสิทธิภาพ หากต้องการข้อมูลเพิ่มเติมเกี่ยวกับ DNase I โปรดไปที่ลิงก์นี้
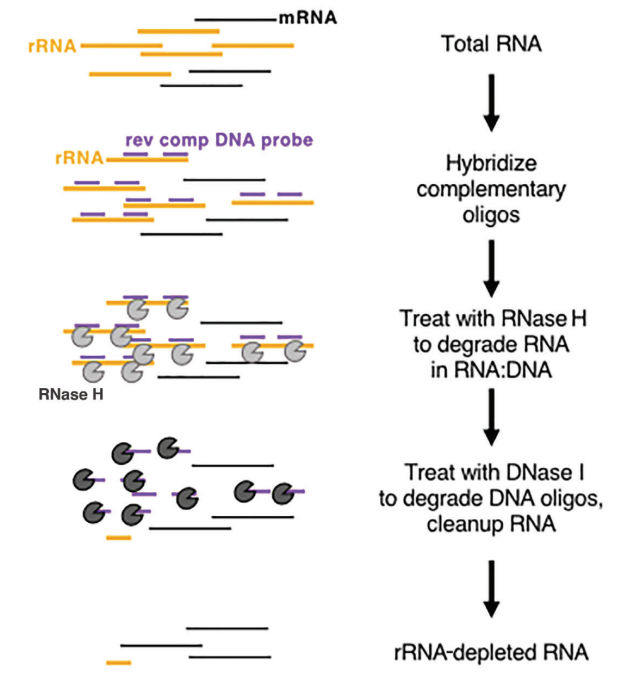
รูปที่ 8: แผนผังแสดงการลดจำนวน rRNA ที่ใช้เอนไซม์[5]
2.2 การแตกตัวของ RNA
โดยทั่วไปภายใต้การกระทำของไอออนโลหะที่มีประจุบวก 2 และอุณหภูมิสูง ชิ้นส่วนขนาดใหญ่ของ RNA จะแตกออกเป็นชิ้นส่วนเล็กๆ
2.3 การสังเคราะห์ cDNA สายที่ 1
การถอดรหัสย้อนกลับของ RNA เป้าหมายที่ได้ลงในสายแรกของ cDNA เนื่องจาก RNA ถูกย่อยสลายได้ง่ายโดย RNases ที่มีอยู่ในสิ่งแวดล้อม การใช้ สารยับยั้ง RNase (Cat#14672) ในระหว่างการถอดรหัสย้อนกลับสามารถยับยั้งการทำงานของเอนไซม์เหล่านี้และปกป้อง RNA จากการย่อยสลายของ RNase ในเวลาเดียวกัน รีเวิร์สทรานสคริปเทส (Cat#11112) ถูกใช้เพื่อถอดรหัสย้อนกลับของเทมเพลต RNA เป็น cDNA ทรานสคริปเทสย้อนกลับมีกิจกรรมดีเอ็นเอโพลีเมอเรสที่ขึ้นอยู่กับ RNA และสามารถใช้ RNA เป็นเทมเพลตเพื่อสังเคราะห์ cDNA ในทิศทาง 5'→3' สายดีเอ็นเอเดี่ยวเป็นส่วนเสริมของเทมเพลต RNA
ในช่วงวันที่ 1 การสังเคราะห์ cDNA ของสาย การรวมแอคติโนไมซินดีเข้าไปช่วยปรับปรุงการสร้างไลบรารีเฉพาะสายได้อย่างไม่ต้องสงสัย ช่วยเพิ่มความจำเพาะของสายอย่างมีนัยสำคัญ นวัตกรรมนี้ทำให้กระบวนการทดลองคล่องตัวขึ้น และทำให้นักวิจัยทำได้ง่ายขึ้น
อย่างไรก็ตาม แอกติโนไมซินดีมีข้อเสียคือเป็นพิษและต้องได้รับการปกป้องจากแสง ในปัจจุบันมีความต้องการชุดสร้างเพลทและแบบผสมสำเร็จเพิ่มมากขึ้น ความจำเป็นในการป้องกันแสงทำให้การพัฒนาชุดเพลทมีข้อจำกัด
โชคดี,

รูปที่ 9: วิศวกรรมของ MMLV เพื่อระบุ MMLV กลายพันธุ์ซึ่งอาจมีส่วนสนับสนุน Standed RNA-seq
2.4 การสังเคราะห์ cDNA สายที่ 2
cDNA สายเดี่ยวที่ผลิตขึ้นโดยการถอดรหัสย้อนกลับนั้นไม่เสถียรมาก จึงจำเป็นต้องสังเคราะห์ cDNA สายที่สองทันทีภายใต้อิทธิพลของ DNA โพลิเมอเรส I ในระหว่างการสังเคราะห์สายที่สองนี้ RNase H จะเข้ามามีบทบาทโดยการกำจัดสาย RNA ออกจากโครงสร้างไฮบริด RNA-DNA โดยทำงานร่วมกับ ดีเอ็นเอโพลีเมอเรส I (Cat#12903) เพื่ออำนวยความสะดวกในการสังเคราะห์สายที่สองของ cDNA ที่เป็นส่วนประกอบกันเอง DNA polymerase I มีกิจกรรม DNA polymerase 5’→3' และด้วยเทมเพลตและไพรเมอร์นำทาง จึงสังเคราะห์ลำดับที่เสริม cDNA สายเดี่ยวในทิศทาง 5’→3'
ขั้นตอนต่อไปในกระบวนการนี้ ได้แก่ การซ่อมแซมปลายสาย dA-Tailing การผูกอะแดปเตอร์ และการขยาย PCR ซึ่งทั้งหมดนี้ได้อธิบายไว้โดยละเอียดในขั้นตอนการสร้างห้องสมุด DNA และไม่จำเป็นต้องกล่าวถึงซ้ำอีกในที่นี้ สิ่งที่สำคัญคือ เมื่อการถอดรหัสย้อนกลับเสร็จสมบูรณ์แล้ว ไม่จำเป็นต้องแบ่งชิ้นส่วนกรดนิวคลีอิกเพิ่มเติมอีก
3. แนวทางการใช้เอ็นไซม์หลัก NGS ในการสร้างไลบรารี DNA และ RNA
ตารางที่ 1.แนวทางการใช้เอนไซม์หลัก NGS ในการสร้างไลบรารี DNA และ RNA
| พิมพ์ | การจัดวางตำแหน่งผลิตภัณฑ์ | ชื่อสินค้า | แมว# |
| ห้องสมุดอาร์เอ็นเอ การก่อสร้าง | อาร์อาร์เอ็นเอ การพร่อง/การสังเคราะห์ cDNA สายที่ 2 | 12906ES | |
| อาร์อาร์เอ็นเอ การหมดลง | 10325ES | ||
| การสังเคราะห์ cDNA สายที่ 1 | 14672ES | ||
| 11112ES | |||
| การสังเคราะห์ cDNA สายที่ 2 | 12903ES | ||
| ห้องสมุดอาร์เอ็นเอ การก่อสร้าง & ดีเอ็นเอ ห้องสมุด การก่อสร้าง | การซ่อมแซมส่วนท้าย | 12901ES | |
| 12902ES | |||
| dA-หาง | 13486ES | ||
| การผูกอะแดปเตอร์ | 10301ES | ||
| พีซีอาร์ การขยายเสียง | มิกซ์ความเที่ยงตรงสูง Super Canace® II 2× สำหรับการขยายเสียงห้องสมุด | 12621ES |
ตารางที่2.ห้องสมุด DNA และ RNA ชุดเตรียมตัว
| ชื่อ | แมว# | หมายเหตุ | |
| ดีเอ็นเอ | ชุดเตรียมห้องสมุด DNA ของ Hieff NGS | 13577ES | วิธีการเนื้องอก/กลไก |
| ชุดเตรียมไลบรารี DNA OnePot Pro ของ Hieff NGS V2 | 12194ES | วิธีเนื้องอก/เอนไซม์ | |
| ไฮฟ์ NGS วันพ็อต ชุดเตรียมห้องสมุด DNA II สำหรับ Illumina | 13490ES | พาธเจน/เอ็นไซม์ติก/เวลาปกติ (140นาที) | |
| ชุดเตรียมไลบรารี DNA แฟลช OnePot ของ Hieff NGS | 12316ES | พาธเจน/เอ็นไซม์ติก/อัลตราฟาสต์ (100นาที) | |
| ชุดเตรียมร่วมห้องสมุด Hieff NGS DNA&RNA V2 | 12305ES | Pathgen/ Enzymetic/ DNA & RNA Co-Prep | |
| อาร์เอ็นเอ | ชุดเตรียมไลบรารี mRNA แบบดูอัลโหมดของ Hieff NGS Ultima | 12308ES | ไม่มีเม็ดแม่เหล็กโอลิโกดีที 11 หลอด |
| ชุดเตรียมไลบรารี mRNA แบบดูอัลโหมดของ Hieff NGS Ultima | 12309ES | โอลิโก ดีที แม่เหล็ก บีดส์ พลัส 14 หลอด | |
| ชุดเตรียมไลบรารี RNA แบบดูอัลโหมด Hieff NGS® Ultima | 12310ES | เวอร์ชั่นพรีมิกซ์ 5 หลอด | |
| ชุดเตรียมไลบรารี RNA Hieff NGS ® EvoMax (เวอร์ชันผสมล่วงหน้า) (แอกติโนไมซิน ดี ฟรี) | 12340ES | เวอร์ชันพรีมิกซ์ (แอกติโนไมซิน ดี ฟรี) | |
| ชุดกำจัด rRNA ของ Hieff NGS® MaxUp (พืช) | 12254ES | ปลูก | |
| ชุดกำจัด rRNA ของมนุษย์ Hieff NGS® MaxUp (rRNA และ ITS/ETS) | 12257ES | มนุษย์ |
อ้างอิง:
[1] Mardis, Elaine R. แพลตฟอร์มการจัดลำดับรุ่นถัดไป[J]. Annual Review of Analytical Chemistry, 2013, 6(1):287-303
[2] Gulilat M, Lamb T, Teft WA และคณะ การจัดลำดับยีนรุ่นถัดไปแบบกำหนดเป้าหมายเป็นเครื่องมือสำหรับการแพทย์ที่แม่นยำ[J] BMC Medical Genomics, 2019, 12(1):81
[3] Lundberg KS, Dan DS, Adams M และคณะ การขยายสัญญาณความเที่ยงตรงสูงโดยใช้ DNA โพลิเมอเรสที่ทนความร้อนซึ่งแยกได้จาก Pyrococcus furiosus[J] Gene, 1991, 108(1):1-6.
[4] Miyazaki K. การแยกชิ้นส่วนของดีเอ็นเอแบบสุ่มด้วยเอ็นโดนิวคลีเอส V: การประยุกต์ใช้ในการสับเปลี่ยนดีเอ็นเอ[J] Nucleic Acids Research, 2002, 30(24):e139
[5] Baldwin A, Morris AR, Mukherjee N. วิธีการที่ง่าย คุ้มต้นทุน และปรับขนาดได้เพื่อลดปริมาณ RNA ไรโบโซมของมนุษย์สำหรับ RNA-seq[J] Current Protocols, 2021, 1(6):e176

