DNase I và ứng dụng của chúng trong Y sinh học
Deoxyribonuclease I (DNase I) là một endonuclease, ứng dụng của nó không chỉ để duy trì tính toàn vẹn của RNA mà còn để phân tích dấu chân DNA, tạo ra các thư viện DNA ngẫu nhiên, giảm độ dính trong dịch phân giải tế bào hoặc chiết xuất protein, v.v. Nói một cách ngắn gọn, DNase I có thể được sử dụng trong hầu hết mọi ứng dụng đòi hỏi phải cắt DNA bằng enzyme. Sau đây là phần giới thiệu chi tiết về DNase I và ứng dụng cụ thể của nó.
1. DNase I là gì?
2. DNase I để chuẩn bị chiết xuất RNA không có DNA
3. DNase I cho phiên mã trong ống nghiệm để loại bỏ DNA khuôn mẫu
4. DNase I để loại bỏ rRNA
5. DNase I để gắn nhãn DNA
6. Các ứng dụng khác
7. Hướng dẫn lựa chọn sản phẩm DNase I
1. DNase I là gì?
Deoxyribonuclease I (DNase I) là một endonuclease không đặc hiệu có thể tiêu hóa DNA mạch đơn hoặc mạch kép, có trong các mô và dịch cơ thể khác nhau. Nó có thể thủy phân các liên kết phosphodiester để tạo ra các mono- và oligodeoxynucleotide chứa nhóm 5'-phosphate và nhóm 3'-OH. Khoảng pH hoạt động tối ưu của DNase I là 7-8, hoạt động của nó phụ thuộc vào Ca2+ và có thể được hoạt hóa bởi các ion kim loại hóa trị hai như Mn2+, Mg2+, Zn2+, v.v. Khi có Mg2+, DNase I cắt ngẫu nhiên bất kỳ vị trí nào của DNA mạch kép; khi có Mn2+, DNase I có thể cắt DNA mạch kép tại cùng một vị trí để tạo thành một đầu tù hoặc một đầu dính 1-2 nucleotide nhô ra.
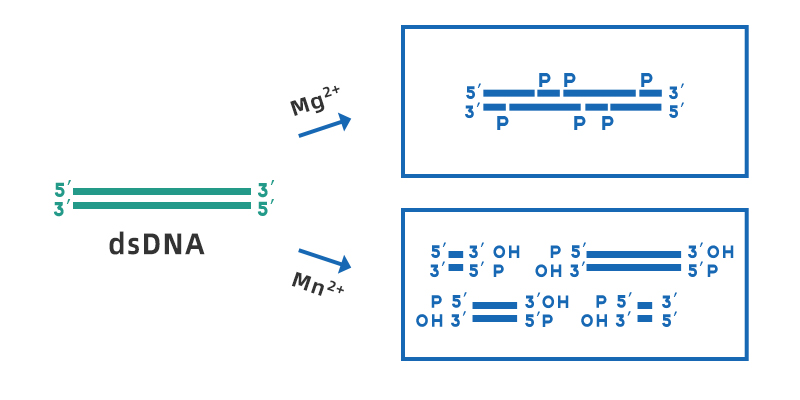
Hình 1. Sơ đồ phân cắt dsDNA bởi DNase I khi có mặt Mg2+ và Mn2+.
Mặc dù sự cắt DNase I thường được coi là sự cắt không đặc hiệu, DNase I có nhiều khả năng tác động lên một số đoạn trình tự nhất định, chẳng hạn như vùng rãnh nhỏ, và dễ bị cắt các trình tự purine-pyrimidine hơn. Tuy nhiên, khi DNase I tác động lên dsDNA không đồng nhất, cả bốn bazơ sẽ bị cắt và tác động lên một bazơ cụ thể sẽ không lớn hơn 3 lần so với các bazơ khác.
2. DNase I để chuẩn bị chiết xuất RNA không có DNA
Trong các thí nghiệm sinh học, bước đầu tiên là chuẩn bị axit nucleic để nghiên cứu các chức năng khác nhau của RNA. Tuy nhiên, vì DNA và RNA thường được giải phóng cùng nhau trong quá trình ly giải tế bào, nên sự can thiệp vào cả hai không thể tránh khỏi bất kể sử dụng dung dịch chiết xuất nào, do đó cần sử dụng các enzyme cụ thể để loại bỏ sự can thiệp. Để chiết xuất RNA chất lượng cao, DNase I được sử dụng để loại bỏ DNA còn sót lại khỏi mẫu.
DNase I có thể phân hủy DNA mạch kép và mạch đơn thành oligonucleotide và nucleotide đơn, và DNA trong sản phẩm chuẩn bị RNA có thể bị phân hủy hiệu quả. DNase I sau đó bị bất hoạt bằng cách đun nóng với đệm dừng. Trong quá trình đun nóng, cấu trúc kẹp tóc của phân tử RNA có thể được mở ra, tạo điều kiện cho RNA đi trực tiếp vào quá trình phiên mã ngược.
Chất lượng của RNA sẽ ảnh hưởng trực tiếp đến dữ liệu thực nghiệm ở mức độ lớn. Nhìn chung, không thể tránh hoàn toàn các dư lượng gDNA trong quá trình chiết xuất RNA, do đó, người ta thường khuyến nghị xử lý các mẫu RNA bằng DNase I để tiêu hóa gDNA còn sót lại trước khi thử nghiệm các ứng dụng hạ nguồn (ví dụ: phân tích biểu hiện mRNA, phân tích phiên mã, v.v.). Bước tiêu hóa gDNA có thể được thực hiện trong quá trình chiết xuất RNA, sau khi chiết xuất RNA hoặc trước khi phiên mã ngược RNA.Theo định vị sản phẩm, các sản phẩm được cung cấp bởi
Bảng 1: Danh sách các sản phẩm liên quan đến việc loại bỏ DNA của quá trình chiết xuất RNA hoặc trước khi phiên mã ngược
| Định vị sản phẩm | Tên sản phẩm | Con mèo # |
| Chiết xuất RNA | Thuốc thử chiết xuất RNA tổng số TRIeasy™ [hỏi thăm] | 10606ES |
| 19221ES | ||
| Bộ dụng cụ RNA MolPure™ Plant Plus [hỏi thăm] | 19292ES | |
| Bộ xét nghiệm DNA/RNA virus MolPure™ [hỏi thăm] | 19321ES | |
| loại bỏ gDNA | 10325ES | |
| Phiên mã ngược | Hifair™Ⅲ1st Strand cDNA Synthesis SuperMix cho qPCR (máy tiêu hóa gDNA cộng thêm) | 11141ES |
| qPCR | 11184ES |
3. DNase I cho phiên mã trong ống nghiệm để loại bỏ DNA khuôn mẫu
Phiên mã trong ống nghiệm (IVT) chủ yếu sử dụng DNA làm khuôn mẫu, cùng với các chất nền và chất đệm tương ứng để thu được RNA thông qua phiên mã trong ống nghiệm. Trong các thí nghiệm phiên mã trong ống nghiệm, RNA polymerase như T7, T3 và SP6 thường được sử dụng để tổng hợp RNA. RNA tổng hợp có thể có các dư lượng DNA. Việc loại bỏ các dư lượng DNA có lợi cho việc phát triển các thí nghiệm hạ nguồn. Ví dụ, trong giai đoạn phát triển vắc-xin mRNA, việc loại bỏ các dư lượng là một bước quan trọng, có thể làm giảm khó khăn trong quá trình tinh chế hạ nguồn và tăng độ tinh khiết của sản phẩm. Khuôn mẫu DNA thường được loại bỏ bằng cách sử dụng DNase I tái tổ hợp (không có RNase).Theo quá trình tổng hợp mRNA, các sản phẩm được cung cấp bởi
| quá trình tổng hợp mRNA | Tên sản phẩm | Con mèo# |
| Chuẩn bị mẫu | DNA Polymerase độ trung thực cao Hieff Canace™ Plus [hỏi thăm] | 10148ES |
| 10922ES | ||
| 10125ES | ||
| FuniCut™BsaI [hỏi thăm] | 15005ES | |
| FuniCut™ XbaI [hỏi thăm] | 15033ES | |
| BspQI[yêu cầu] [hỏi thăm] | 16215ES | |
| Phiên mã trong ống nghiệm | 10623ES | |
| 10624ES | ||
| T7 RNA polymerase (50 U/μL)[hỏi thăm] | 10618ES | |
| 10133ES | ||
| 10620ES | ||
| 10621ES | ||
| Loại bỏ mẫu DNA | 10611ES | |
| sửa đổi mRNA | 10614ES | |
| 10612ES | ||
| 10132ES | ||
| 10619ES | ||
| tinh chế mRNA | 12602ES |
4. DNase I để loại bỏ rRNA
Trong cơ thể sống, rRNA rất phong phú và rất bảo thủ, điều này không có nhiều ý nghĩa trong việc thu thập thông tin sinh học, do đó rRNA thường bị loại bỏ đầu tiên trong quá trình xây dựng thư viện RNA và giải trình tự.Hiện nay, phương pháp loại bỏ rRNA chủ yếu là tiêu hóa bằng RNase H. Các bước chính của quá trình loại bỏ rRNA dựa trên enzyme được thể hiện trong hình 2: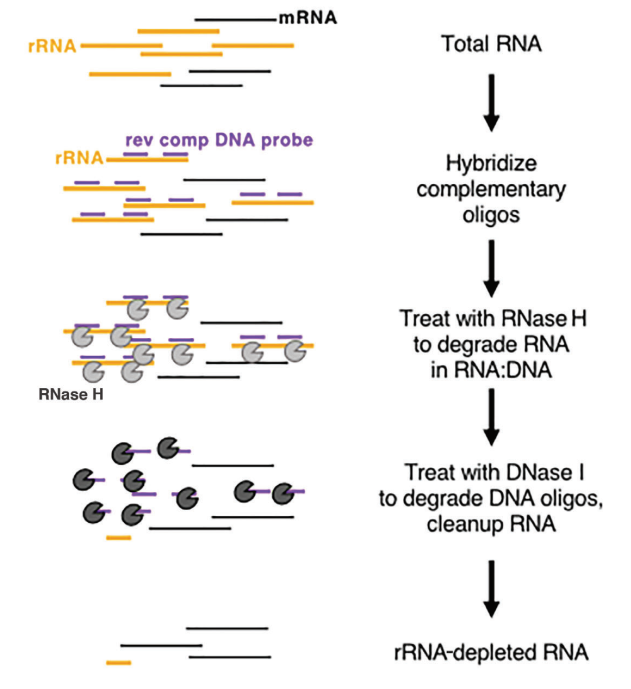
Hình 2: Sơ đồ nguyên lý của sự suy giảm rRNA dựa trên enzyme (Baldwin, A. et al. 2021, Giao thức hiện tại)
Đầu tiên, trích xuất tổng RNA, sau đó lai đầu dò DNA mạch đơn với rRNA, thiết kế và tổng hợp đầu dò DNA mạch đơn đặc hiệu rRNA, sau đó sử dụng RNase H để phân hủy rRNA đã lai và sử dụng DNase I để phân hủy đầu dò DNA. Cuối cùng, để lại khuôn mẫu RNA không phải rRNA. Các sản phẩm liên quan đến việc loại bỏ rRNA được cung cấp bởi
| quá trình tổng hợp mRNA | Tên sản phẩm | Con mèo# |
| Con người/Chuột/Chuột cống Sự suy giảm rRNA | Hieff NGS™ Bộ dụng cụ làm suy giảm rRNA MaxUp (Người/Chuột/Chuột cống) MaxUp [hỏi thăm] | 12253ES |
| Sự suy giảm rRNA thực vật | 12254ES | |
| Loại bỏ RNA ribosome và vùng 45S ITS/ETS khỏi tổng RNA của con người | Hieff NGS™ Bộ dụng cụ làm suy giảm rRNA của con người MaxUp (rRNA & ITS/ETS) | 12257ES |
| Sự phân hủy của rRNA | 12906ES | |
| Sự suy thoái của đầu dò DNA | 10325ES |
5. DNase I để gắn nhãn DNA
Dịch mã nick là một trong những phương pháp đánh dấu đầu dò axit deoxyribonucleic được sử dụng phổ biến nhất trong phòng thí nghiệm. Phương pháp này sử dụng nhiều hoạt động enzym khác nhau của DNA polymerase I để kết hợp deoxyribonucleoside triphosphate được đánh dấu vào chuỗi DNA mới tổng hợp. Do đó, các đầu dò DNA được đánh dấu đồng nhất cho hoạt động đặc hiệu cao được tổng hợp. Các đặc điểm của dịch mã nick là nhanh, đơn giản, thận trọng, độ đặc hiệu cao và đầu dò được đánh dấu đồng nhất, phù hợp với DNA sợi đôi dài hơn.Phương pháp này được thực hiện nhờ hoạt động kết hợp của DNase I và E. coli DNA Polymerase I. Các bước chính của quá trình gắn nhãn DNA bằng dịch mã nick được thể hiện trong hình 3:
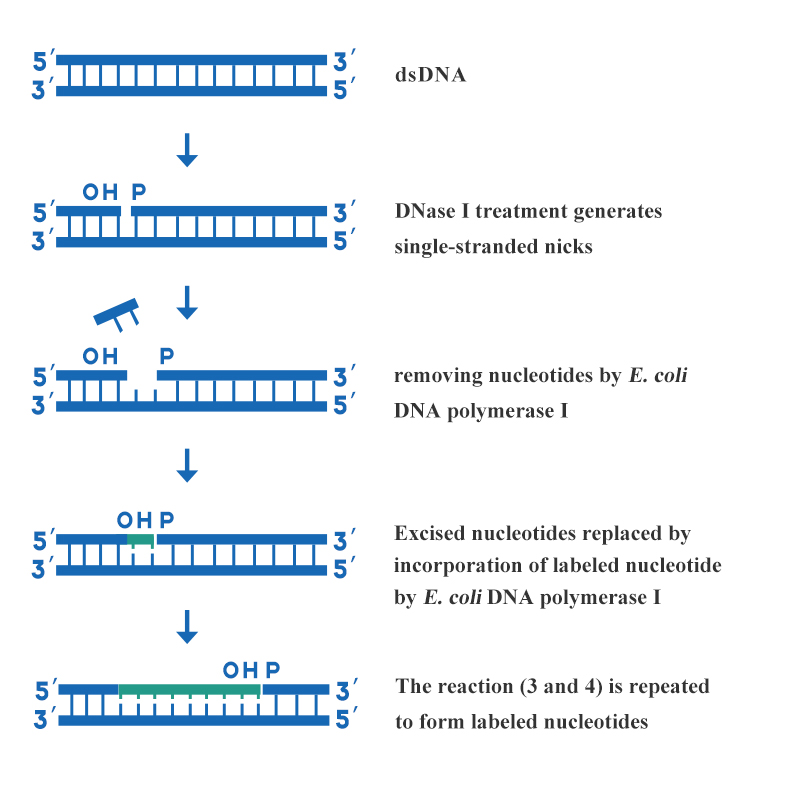
Hình 3: Sơ đồ đánh dấu DNA bằng cách dịch mã nick
Nồng độ DNase I thích hợp được sử dụng để tạo ra một số khoảng trống sợi đơn trên mỗi sợi của DNA sợi đôi cần được đánh dấu và đầu cuối hydroxyl 3' được hình thành tại khoảng trống. Sử dụng hoạt động exonuclease 5'→3' của E. coli DNA Polymerase I để cắt một nucleotide từ đầu 5' của vết khía, đồng thời hoạt động polymerase 5'→3' của E. coli DNA Polymerase I đưa vào một nucleotide được đánh dấu bằng đầu 3' của khoảng trống để sửa chữa khoảng trống. Khi khoảng trống di chuyển dọc theo sợi DNA, các nucleotide được đánh dấu được kết hợp vào sợi mới tổng hợp.Các sản phẩm liên quan đến nhãn DNA được cung cấp bởi
| Vị trí sản phẩm | Tên sản phẩm | Con mèo# |
| Bình thường | Deoxyribonuclease I (DNase I) từ tuyến tụy bò [hỏi thăm] | 10607ES/10608ES |
| Không có RNase | 10325ES | |
| Vi khuẩn E.coli nguồn | 12903ES |
6. Các ứng dụng khác
Trên đây là một số ứng dụng thường dùng. Các ứng dụng khác của DNase I bao gồm các ứng dụng sau, chẳng hạn như xét nghiệm dấu chân DNase I và các vị trí quá nhạy cảm với DNase I. Xét nghiệm dấu chân DNase I là phương pháp phát hiện có thể xác định chính xác các vị trí liên kết của protein liên kết DNA trên DNA. Khi một protein liên kết với một đoạn DNA, nó có thể bảo vệ vị trí liên kết khỏi bị DNase I làm hỏng và các đoạn DNA sẽ được để lại sau khi tiêu hóa bằng enzyme ("dấu chân") và trình tự của nó có thể được xác định. Trong hình ảnh gel, không có dải nào mà DNA liên kết với protein. Để đọc thêm hãy nhấp vào liên kết.Các vị trí quá nhạy cảm với DNase I đề cập đến các vết cắt ở một số ít các vị trí cụ thể khi chromatin được xử lý bằng DNase I thấp và các vị trí cụ thể này được gọi là các vị trí quá nhạy cảm với DNase I. Nguyên tắc là khi một gen ở trạng thái hoạt động phiên mã, chromatin chứa gen đó nhạy cảm hơn đáng kể với sự phân hủy DNase so với vùng không hoạt động. Để đọc thêm, hãy nhấp vào liên kết.
7. Hướng dẫn lựa chọn sản phẩm DNase I
| Tên sản phẩm (Mã số Cat) | Vị trí sản phẩm | Ứng dụng được đề xuất |
| DNase I từ tuyến tụy bò (CAT#10607,10608)[hỏi thăm] | RNase đã loại bỏ, không phát hiện được | Chủ yếu được sử dụng trong nghiên cứu protein: Loại bỏ DNA khỏi chế phẩm protein. |
| DNase I tái tổ hợp (không có RNase)(MÃ SỐ CAT#10325) | Không có RNase, dùng cho nghiên cứu | Thích hợp cho nhiều ứng dụng: Loại bỏ DNA khỏi các chế phẩm RNA và protein như thư viện cDNA nhạy cảm với RNase hoặc chuẩn bị mẫu cho các thí nghiệm RT-PCR. |
| UCF.ME™Deoxyribonuclease I (DNase I) đạt chuẩn GMP(MÃ SỐ 10611) | Không chứa RNase, đạt tiêu chuẩn dược phẩm GMP. | Thích hợp cho nhiều ứng dụng: Loại bỏ DNA khỏi các chế phẩm RNA và protein như thư viện cDNA nhạy cảm với RNase hoặc chuẩn bị mẫu cho các thí nghiệm RT-PCR. |
Về việc đọc:
Thuốc thử đạt chuẩn GMP cho tổng hợp mRNA trong ống nghiệm
Nguyên tắc của DNase I footprinting và các ứng dụng y sinh của nó
Tài liệu tham khảo
1. Baldwin A, Morris AR, Mukherjee N. Một phương pháp dễ dàng, tiết kiệm chi phí và có thể mở rộng để làm cạn kiệt RNA ribosome của con người cho RNA-seq[J]. Giao thức hiện tại, 2021.
2. Song C, Zhang S, Huang H. Chọn phương pháp phù hợp để xác định nguồn gốc sao chép trong bộ gen vi khuẩn [J]. Frontiers in Microbiology, 2015, 6:1049.
