DNase I und ihre Anwendungen in der Biomedizin
Desoxyribonuklease I (DNase I) ist eine Endonuklease, die nicht nur zur Aufrechterhaltung der RNA-Integrität verwendet wird, sondern auch zur DNA-Footprint-Analyse, zur Generierung zufälliger DNA-Bibliotheken, zur Verringerung der Klebrigkeit in Zelllysaten oder Proteinextrakten usw. Kurz gesagt kann DNase I in fast allen Anwendungen eingesetzt werden, die eine enzymatische Spaltung von DNA erfordern. Im Folgenden finden Sie eine detaillierte Einführung in DNase I und ihre spezifische Anwendung.
1. Was ist DNase I?
2. DNase I zur Herstellung der DNA-freien RNA-Extraktion
3. DNase I für die In-vitro-Transkription zur Entfernung der Template-DNA
4. DNase I zur rRNA-Entfernung
5. DNase I zur DNA-Markierung
6. Andere Anwendungen
7. Leitfaden zur DNase I-Produktauswahl
1. Was ist DNase I?
Desoxyribonuklease I (DNase I) ist eine unspezifische Endonuklease, die einzel- oder doppelsträngige DNA verdauen kann, die in verschiedenen Geweben und Körperflüssigkeiten vorkommt. Sie kann Phosphodiesterbindungen hydrolysieren, um Mono- und Oligodesoxynukleotide zu produzieren, die eine 5'-Phosphatgruppe und eine 3'-OH-Gruppe enthalten. Der optimale pH-Arbeitsbereich von DNase I liegt bei 7-8, ihre Aktivität hängt von Ca2+ ab und kann durch zweiwertige Metallionen wie Mn2+, Mg2+, Zn2+ usw. aktiviert werden. In Gegenwart von Mg2+ schert DNase I zufällig jede Stelle doppelsträngiger DNA; in Gegenwart von Mn2+ kann DNase I doppelsträngige DNA an derselben Stelle scheren, um ein stumpfes Ende oder einen Überhang mit 1-2 Nukleotiden langen klebrigen Enden zu bilden.
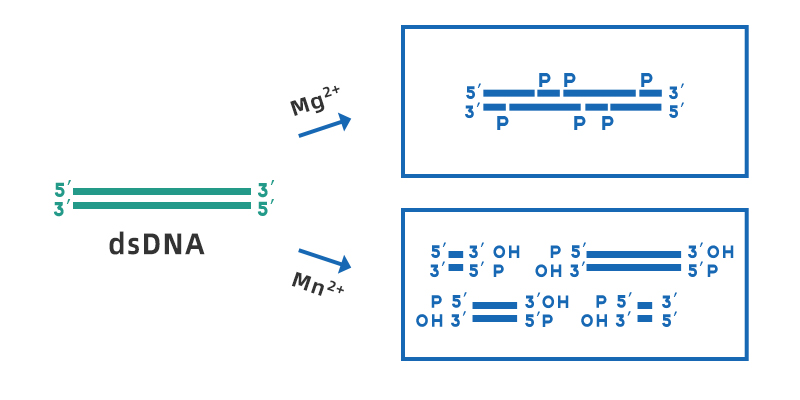
Abbildung 1. Schematische Darstellung der Spaltung von dsDNA durch DNase I in Gegenwart von Mg2+ und Mn2+.
Obwohl DNase I-Spaltungen im Allgemeinen als unspezifische Spaltungen gelten, wirkt DNase I eher auf bestimmte Sequenzfragmente, wie etwa die Minor-Furch-Region, und neigt eher zu Spaltungen von Purin-Pyrimidin-Sequenzen. Wenn DNase I jedoch auf heterogene dsDNA einwirkt, werden alle vier Basen gespalten, und die Wirkung auf eine bestimmte Base ist nicht mehr als dreimal größer als die auf andere Basen.
2. DNase I zur Herstellung der DNA-freien RNA-Extraktion
Bei biologischen Experimenten besteht der erste Schritt darin, Nukleinsäure vorzubereiten, um verschiedene Funktionen von RNA zu untersuchen. Da DNA und RNA jedoch während des Zelllyseprozesses häufig gemeinsam freigesetzt werden, lässt sich die Interferenz zwischen beiden nicht vermeiden, egal welche Extraktionslösung verwendet wird. Daher müssen spezielle Enzyme verwendet werden, um die Interferenz zu beseitigen. Für eine qualitativ hochwertige RNA-Extraktion wird DNase I verwendet, um restliche DNA aus der Probe zu entfernen.
DNase I kann doppelsträngige und einzelsträngige DNA zu Oligonukleotiden und Einzelnukleotiden abbauen, und die DNA im RNA-Präparationsprodukt kann effektiv abgebaut werden. DNase I wird dann durch Erhitzen mit einem Stopppuffer inaktiviert. Während des Erhitzungsprozesses kann die Haarnadelstruktur des RNA-Moleküls geöffnet werden, was den direkten Eintritt der RNA in den Reverse-Transkriptionsprozess erleichtert.
Die Qualität der RNA hat einen großen direkten Einfluss auf die experimentellen Daten. Im Allgemeinen können gDNA-Rückstände während der RNA-Extraktion nicht vollständig vermieden werden. Daher wird im Allgemeinen empfohlen, RNA-Proben mit DNase I zu behandeln, um restliche gDNA zu verdauen, bevor sie in nachgelagerte Anwendungen (z. B. mRNA-Expressionsanalyse, Transkriptomanalyse usw.) überführt werden. Der Schritt des Verdauens von gDNA kann während der RNA-Extraktion, nach der RNA-Extraktion oder vor der Reverse-Transkription von RNA durchgeführt werden.Entsprechend der Produktpositionierung sind die von
Tabelle 1: Liste der Produkte zur DNA-Entfernung bei der RNA-Extraktion oder vor der Reversen Transkription
| Produktpositionierung | Produktname | Katze # |
| RNA-Extraktion | TRIeasy™ Gesamt-RNA-Extraktionsreagenz [erkundigen] | 10606ES |
| 19221ES | ||
| MolPure™ Plant Plus RNA Kit [erkundigen] | 19292ES | |
| MolPure™ Virales DNA/RNA-Kit [erkundigen] | 19321ES | |
| gDNA-Entfernung | 10325ES | |
| Rücktranskription | Hifair™Ⅲ1. Strang cDNA Synthese SuperMix für qPCR (gDNA Digester plus) | 11141ES |
| qPCR | 11184ES |
3. DNase I für die In-vitro-Transkription zur Entfernung der Template-DNA
Bei der In-vitro-Transkription (IVT) wird hauptsächlich DNA als Vorlage sowie entsprechende Substrate und Puffer verwendet, um durch In-vitro-Transkription RNA zu erhalten. Bei In-vitro-Transkriptionsexperimenten werden häufig RNA-Polymerasen wie T7, T3 und SP6 zur RNA-Synthese verwendet. Die synthetisierte RNA kann DNA-Reste enthalten. Die Beseitigung der DNA-Reste ist für die Entwicklung nachfolgender Experimente von Vorteil. Beispielsweise ist in der Entwicklungsphase eines mRNA-Impfstoffs die Entfernung von Resten ein entscheidender Schritt, der die Schwierigkeit der nachfolgenden Reinigung verringern und die Reinheit des Produkts erhöhen kann. Die DNA-Vorlage wird normalerweise mithilfe von rekombinanter DNase I (RNase-frei) entfernt.Gemäß dem mRNA-Syntheseprozess sind die von
| mRNA-Syntheseprozess | Produktname | Katze# |
| Vorlagenvorbereitung | Hieff Canace™ Plus hochpräzise DNA-Polymerase [erkundigen] | 10148ES |
| 10922ES | ||
| 10125ES | ||
| FuniCut™BsaI [erkundigen] | 15005ES | |
| FuniCut™ XbaI [erkundigen] | 15033ES | |
| BspQI[anfragen] [erkundigen] | 16215ES | |
| In-vitro-Transkription | 10623ES | |
| 10624ES | ||
| T7-RNA-Polymerase (50 U/μL)[erkundigen] | 10618ES | |
| 10133ES | ||
| 10620ES | ||
| 10621ES | ||
| Entfernen der Vorlagen-DNA | 10611ES | |
| mRNA-Modifikation | 10614ES | |
| 10612ES | ||
| 10132ES | ||
| 10619ES | ||
| mRNA-Reinigung | 12602ES |
4. DNase I zur rRNA-Entfernung
In vivo ist rRNA in großen Mengen vorhanden und sehr konservativ, was für die Gewinnung biologischer Informationen kaum von Bedeutung ist. Daher wird rRNA bei der Konstruktion und Sequenzierung von RNA-Bibliotheken häufig zuerst entfernt.Zurzeit wird rRNA hauptsächlich durch RNase H-Verdauung entfernt. Die wichtigsten Schritte der enzymatischen rRNA-Depletion sind in Abbildung 2 dargestellt: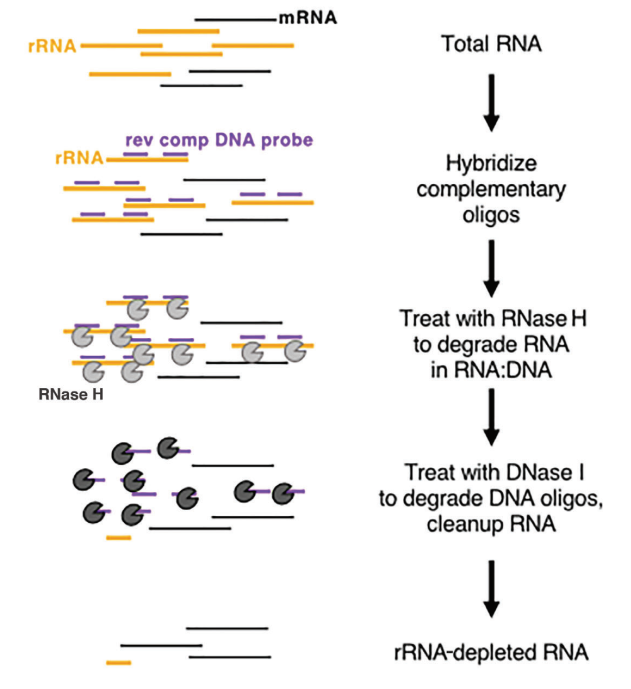
Abbildung 2: Schematische Darstellung des Prinzips der enzymatischen rRNA-Depletion (Baldwin, A. et al. 2021, Current Protocols)
Zuerst wird die gesamte RNA extrahiert, dann wird die einzelsträngige DNA-Sonde mit rRNA hybridisiert, dann wird eine rRNA-spezifische einzelsträngige DNA-Sonde entworfen und synthetisiert, dann wird die hybridisierte rRNA mit RNase H abgebaut und die DNA-Sonde mit DNase I abgebaut. Schließlich bleibt die nicht-rRNA-RNA-Vorlage übrig. Die von
| mRNA-Syntheseprozess | Produktname | Katze# |
| Mensch/Maus/Ratte rRNA-Dezimierung | Hieff NGS™ MaxUp rRNA-Depletionskit (Mensch/Maus/Ratte) MaxUp [erkundigen] | 12253ES |
| rRNA-Dezimierung bei Pflanzen | 12254ES | |
| Entfernung ribosomaler RNA und 45S ITS/ETS-Regionen aus menschlicher Gesamt-RNA | Hieff NGS™ MaxUp Human rRNA Depletion Kit (rRNA und ITS/ETS) | 12257ES |
| Abbau von rRNA | 12906ES | |
| Abbau von DNA-Sonden | 10325ES |
5.DNase I zur DNA-Markierung
Die Nick-Translation ist eine der im Labor am häufigsten verwendeten Markierungsmethoden für Desoxyribonukleinsäuresonden. Diese Methode nutzt verschiedene enzymatische Aktivitäten der DNA-Polymerase I, um markierte Desoxyribonukleosidtriphosphate in neu synthetisierte DNA-Ketten einzubauen. Auf diese Weise werden einheitlich markierte DNA-Sonden mit hoher spezifischer Aktivität synthetisiert. Die Merkmale der Nick-Translation sind schnelle, einfache, gezielte, hochspezifische und einheitlich markierte Sonden, die für längere doppelsträngige DNA geeignet sind.Die Methode wird durch die kombinierte Wirkung von DNase I und E. coli DNA-Polymerase I realisiert. Die wichtigsten Schritte der DNA-Markierung durch Nick-Translation sind in Abbildung 3 dargestellt:
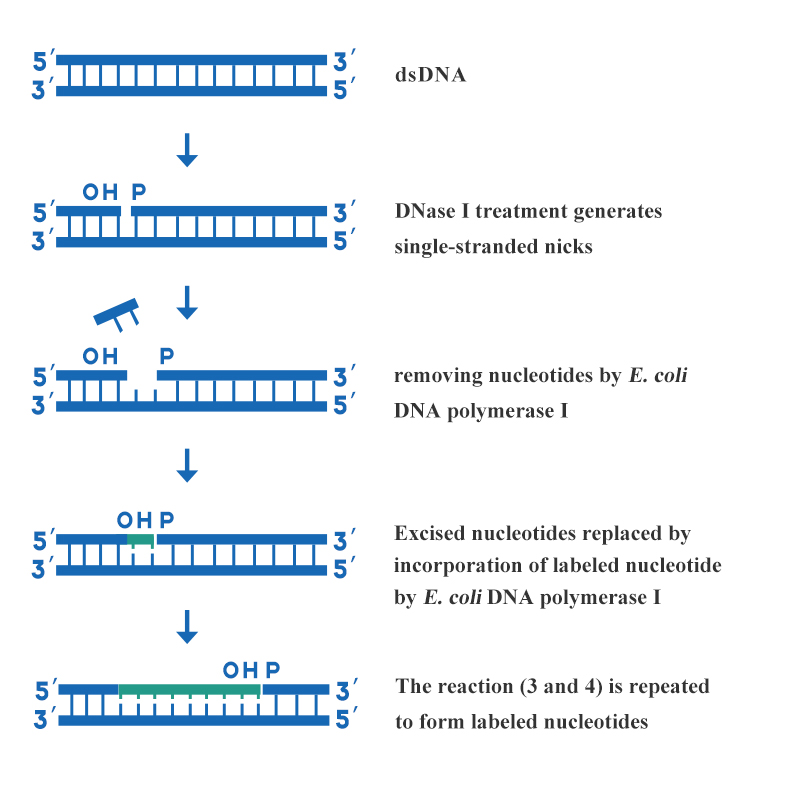
Abbildung 3: Schematische Darstellung der DNA-Markierung durch Nick-Translation
Eine geeignete Konzentration von DNase I wird verwendet, um mehrere einzelsträngige Lücken auf jedem Strang der zu markierenden doppelsträngigen DNA zu erzeugen, und das 3'-Hydroxyl-Terminus wird an der Lücke gebildet. Verwenden Sie die 5'→3'-Exonuklease-Aktivität von E. coli DNA-Polymerase I, um ein Nukleotid vom 5'-Ende des Einschnitts abzuschneiden, und führen Sie gleichzeitig mit der 5'→3'-Polymerase-Aktivität von E. coli DNA-Polymerase I ein mit dem 3'-Ende der Lücke markiertes Nukleotid ein, um die Lücke zu reparieren. Während sich die Lücke entlang des DNA-Strangs bewegt, werden die markierten Nukleotide in den neu synthetisierten Strang eingebaut.Die von
| Produktpositionierung | Produktname | Katze# |
| Normal | Desoxyribonuklease I (DNase I) aus Rinderpankreas [erkundigen] | 10607ES/10608ES |
| RNase-frei | 10325ES | |
| E.coli Quelle | 12903ES |
6. Andere Anwendungen
Oben sind einige häufig verwendete Anwendungen aufgeführt. Weitere Anwendungen von DNase I sind die folgenden, wie z. B. der DNase I-Footprinting-Test und DNase I-hypersensitive Stellen. Der DNase I-Footprinting-Test ist eine Nachweismethode, mit der die Bindungsstellen von DNA-bindenden Proteinen auf DNA genau identifiziert werden können. Wenn ein Protein an ein DNA-Fragment bindet, kann es die Bindungsstelle vor Beschädigung durch DNase I schützen. Die DNA-Fragmente bleiben nach der enzymatischen Verdauung („Footprint“) zurück und ihre Sequenz kann bestimmt werden. Im Gelbild gibt es keine Bänder, wo die DNA an das Protein bindet. Um mehr zu lesen, klicken Sie auf den Link.DNase I-hypersensitive Stellen beziehen sich auf Spaltungen an einer kleinen Anzahl spezifischer Stellen, wenn Chromatin mit wenig DNase I behandelt wird. Diese spezifischen Stellen werden als DNase I-hypersensitive Stellen bezeichnet. Das Prinzip besteht darin, dass, wenn sich ein Gen in einem transkriptionell aktiven Zustand befindet, das Chromatin, das das Gen enthält, deutlich empfindlicher auf DNase-Abbau reagiert als die inaktive Region. Klicken Sie auf den Link, um mehr zu lesen.
7. Leitfaden zur DNase I-Produktauswahl
| Produktname (Kat.-Nr.) | Produktpositionierung | Empfohlene Anwendungen |
| DNase I aus Rinderpankreas (CAT#10607,10608)[erkundigen] | RNase entfernt, nicht erkannt | Hauptsächlich verwendet in der Proteinforschung: DNA-Entfernung aus Proteinpräparaten. |
| Rekombinante DNase I (RNase-frei)(KAT#10325) | RNase-frei, für die Forschung | Ideal für eine Vielzahl von Anwendungen: DNA-Entfernung aus RNA- und Proteinpräparaten wie RNase-sensitiven cDNA-Bibliotheken oder Probenvorbereitung für RT-PCR-Experimente. |
| UCF.ME™Desoxyribonuklease I (DNase I) GMP-Qualität(KAT#10611) | RNase-frei, GMP-pharmazeutische Qualität. | Ideal für eine Vielzahl von Anwendungen: DNA-Entfernung aus RNA- und Proteinpräparaten wie RNase-sensitiven cDNA-Bibliotheken oder Probenvorbereitung für RT-PCR-Experimente. |
Zum Thema Lesen:
GMP-Reagenzien für die mRNA-In-vitro-Synthese
Die Prinzipien des DNase I-Footprintings und seine biomedizinischen Anwendungen
Verweise
1. Baldwin A, Morris AR, Mukherjee N. Eine einfache, kostengünstige und skalierbare Methode zur Depletion menschlicher ribosomaler RNA für RNA-Sequenzierung[J]. Aktuelle Protokolle, 2021.
2. Song C, Zhang S, Huang H. Auswahl einer geeigneten Methode zur Identifizierung von Replikationsursprüngen in mikrobiellen Genomen[J]. Frontiers in Microbiology, 2015, 6:1049.
