Was tun, wenn die qPCR-Ergebnisse seltsam sind?
qPCR-Experimente können sich einfach anfühlen, manchmal aber auch schwierig. Nachdem das Primer-Design abgeschlossen ist, werden qPCR-Proben wie bei einer PCR Schritt für Schritt hinzugefügt und das Experiment kann auf der Maschine abgeschlossen werden. Bei qPCR-Experimenten mit der Farbstoffmethode treten mehrere häufige Probleme auf. Angesichts der möglichen Ursachen und Lösungen hoffen wir, dass Sie diese zur Lösung der Probleme bei qPCR-Experimenten verwenden können.
1. Was ist qPCR?
2. Abnormale qPCR-Amplifikationskurve
3. Abnormale qPCR-Schmelzkurve
4. Bestellinformationen
5. Verwandte Produkte
6. Zum Thema Lesen
1. Was ist qPCR?
Seit der Erfindung der Polymerase-Kettenreaktion (PCR) ist die PCR aufgrund ihrer Einfachheit, Kostengünstigkeit, Zuverlässigkeit, Schnelligkeit und hohen Empfindlichkeit wahrscheinlich die am weitesten verbreitete Technik in der Molekularbiologie. qPCR ist eine Technologie, die von der PCR-Technologie entwickelt wurde. Während des DNA-Amplifikationsprozesses weist die Methode zum Nachweis der Gesamtmenge der Produkte nach jedem PCR-Zyklus mit Fluoreszenzfarbstoffen nicht nur die Schnelligkeit und Empfindlichkeit der PCR auf, sondern bietet auch eine höhere Spezifität und Höhe, Echtzeitüberwachung, wiederholbare und genaue Quantifizierung und andere Vorteile. qPCR ist eine Technologie, die ein quantitatives Echtzeit-PCR-Amplifikationsgerät, nämlich ein qPCR-Instrument, verwendet, um Nukleinsäureamplifikationsprodukte während des PCR-Prozesses in Echtzeit zu überwachen. qPCR realisiert die quantitative Analyse der Ausgangsvorlage durch die Analyse des Ct-Werts und der Standardkurve. 1992 schlug der Japaner Higuchi erstmals die „Echtzeit-Fluoreszenz-quantitative PCR-Technologie“ vor. 1996 brachte die American Biological Company das weltweit erste Fluoreszenz-Quantitative-PCR-Gerät auf den Markt, das aus einem thermischen PCR-Amplifikationssystem, einem optischen Fluoreszenzdetektionssystem sowie Computer- und Anwendungssoftware besteht. Es kann Nukleinsäureamplifikationsprodukte in Echtzeit durch Fluoreszenzfarbstoffe oder Fluoreszenzsonden überwachen. Durch die mathematische Funktionsbeziehung, kombiniert mit der Software zur Ergebnisanalyse, wird die Berechnung der anfänglichen Vorlagenmenge der zu testenden Probe realisiert. Daher wird die Echtzeit-Fluoreszenz-Quantitative-PCR-Technologie weithin eingesetzt. Fluoreszenzmarkierungsmethoden für qPCR werden in Fluoreszenzfarbstoffmosaikmethoden basierend auf der SYBR Green I-Farbstoffmethode, Fluoreszenzsondenmethoden basierend auf der Taqman-Sondenmethode (Cycling Probe, Molecular Bracon usw.) und Quencher-Farbstoffprimermethoden unterteilt.
qPCR-Experimente können sich einfach anfühlen, manchmal aber auch schwierig. Nachdem das Primer-Design abgeschlossen ist, werden qPCR-Proben wie bei einer PCR Schritt für Schritt hinzugefügt und das Experiment kann auf der Maschine abgeschlossen werden. Allerdings müssen viele kleine Details beachtet werden, z. B. dass keine Flüssigkeit an der Rohrwand hängt, keine Blasen im Rohr sind und der direkte Kontakt zwischen Rohrwand, Rohrabdeckung usw. minimiert werden muss, um Versuchsfehler oder seltsame Versuchsergebnisse zu vermeiden. Xiaoyi hat mehrere häufige Probleme bei qPCR-Experimenten mit der Farbstoffmethode zusammengestellt und die möglichen Ursachen und Lösungen angegeben, die Ihnen hoffentlich bei der Lösung der Probleme bei qPCR-Experimenten helfen.
2. Abnormale qPCR-Amplifikationskurve
2.1 Auffälligkeiten in der Baseline-Phase der logarithmischen Darstellung der Amplifikationskurve
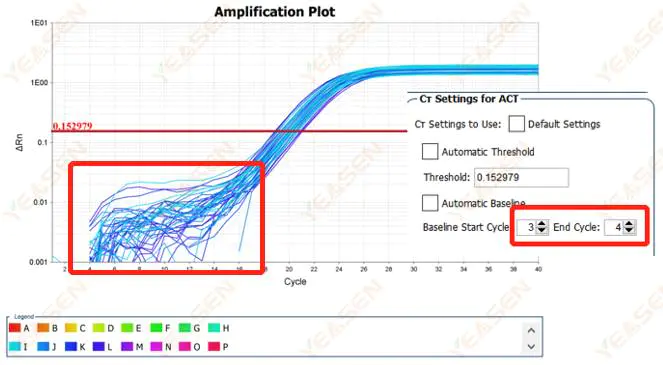
Abbildung 1. Amplifikationsdiagramm
Mögliche Ursachen:
Falsche Basislinieneinstellung.
Lösungen:
Es wird empfohlen, den Endpunkt der Basislinie zu erhöhen.Das Anpassungsbeispieldiagramm ist wie folgt.

Abbildung 2. Amplifikationsdiagramm
2.2 Segmentierung der Amplifikationskurve im Log-Plot
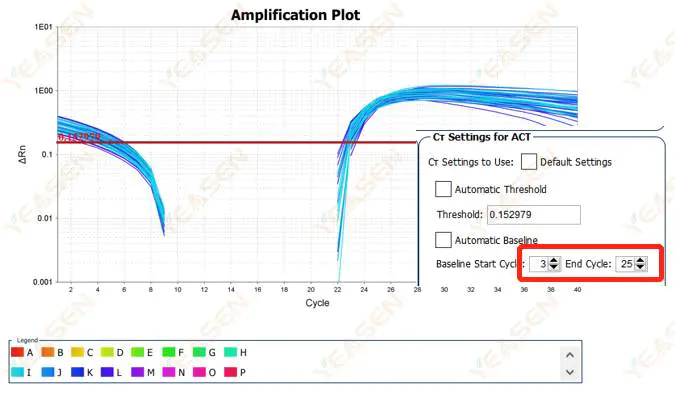
Abbildung 3. Amplifikationsdiagramm
Mögliche Ursachen:
Die Grundlinie ist zu hoch eingestellt.
Lösungen:
Es wird empfohlen, den Endpunktwert der Basislinie zu reduzieren. Das Anpassungsbeispieldiagramm ist wie folgt.
2.3 Ungleichmäßiges Linearitätsdiagramm der Verstärkungskurve
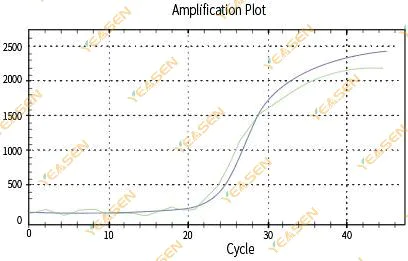
Abbildung 4. Amplifikationsdiagramm
Mögliche Ursachen:
1) Das PCR-Reaktionsröhrchen war nicht fest verschlossen und die Reaktionslösung ist ausgelaufen.
2)PCR-Reaktionslösung mit hängender Wand.
3) Das Instrument ist nicht kalibriert (einschließlich automatischer Kalibrierung oder ROX-Kalibrierung).
4) Das System enthält viele Inhibitoren, was zu einer instabilen Fluoreszenz führt.
5) Übermäßiger Gebrauch des Instruments führt zu einer instabilen Fluoreszenzsammlung.
Lösungen:
1)Drücken Sie die Rohrabdeckung fest.
2) Mischen Sie das Reagenz gründlich und geben Sie es nach vollständiger Zentrifugation vorsichtig in das quantitative Instrument.
3)Kalibrieren Sie das Instrument.
4) Verbessern Sie die RNA-Reinheit und wählen Sie geeignete Reagenzien für die Reverse-Transkription aus.
2.4 Verstärkte Kurve linearer Graph Zickzack auf einem Plateau
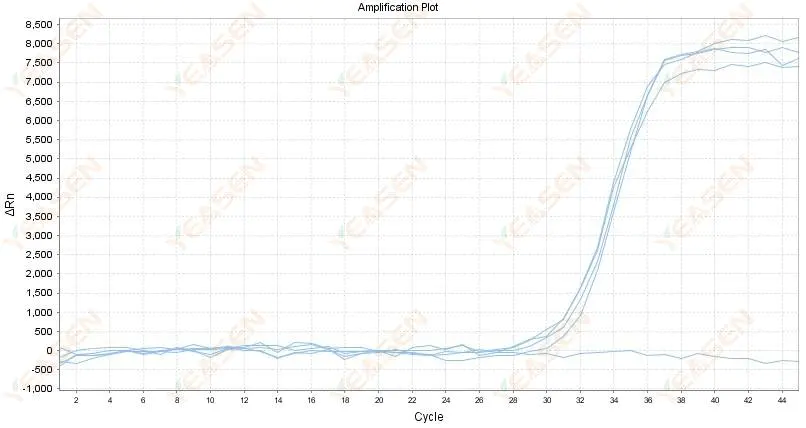
Abbildung 5. Amplifikationsdiagramm
Mögliche Ursachen:
1) Die Reinheit der RNA ist mangelhaft und es gibt viele Verunreinigungen.
2) Das Instrument wird zu lange verwendet.
Lösungen:
1) Extrahieren Sie hochwertige RNA erneut.
2) Verdünnen Sie die RNA-Vorlage, um die Konzentration der Verunreinigung zu reduzieren.
3) Kalibrieren Sie das Instrument.
2.5 Linearer Verlauf der Amplifikationskurve erreicht kein Plateau
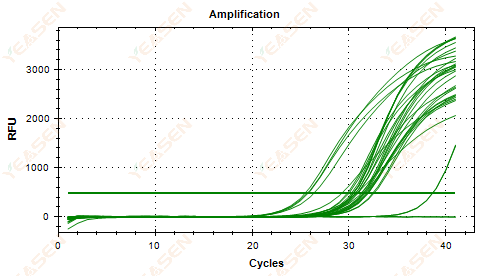
Abbildung 6. Amplifikationsdiagramm
Mögliche Ursachen:
1) Niedrige Template-Konzentration (Ct-Wert ca. 35).
2) Zu wenige Amplifikationszyklen.
3) Geringe Effizienz der Reagenzverstärkung (kleiner Ct, kann aber auch kein Plateau erreichen).
Lösungen:
1) Erhöhen Sie die Konzentration der Vorlage.
2) Erhöhen Sie die Anzahl der Zyklen.
3) Erhöhung des Mg2+ Konzentration.
2.6 Verstärkungskurve linearer Verlauf Plateau Absacken
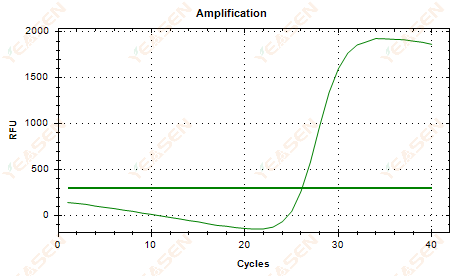
Abbildung 7. Amplifikationsdiagramm
Mögliche Ursachen:
1) Vorhandensein von Degradation (Degradation des Amplifikationsprodukts, SYBR-Degradation).
2) Der Röhrchendeckel ist nicht richtig verschlossen und das Reagenz verflüchtigt sich.
3) Die cDNA-Konzentration ist zu hoch (wenn der Ct-Wert zu niedrig ist, wird die Fluoreszenzschwelle zu hoch angesetzt und das Absacken ist stärker).
4) Luftblasen im Rohr, dann verschwinden sie.
Lösungen:
1) Verbessern Sie die Systemreinheit.
2) Reduzieren Sie die cDNA-Menge (verdünnen Sie die Vorlage).
3) Verringern Sie den Endpunkt der Basislinie.
2.7 Großer Ct-Wert in der Amplifikationskurve
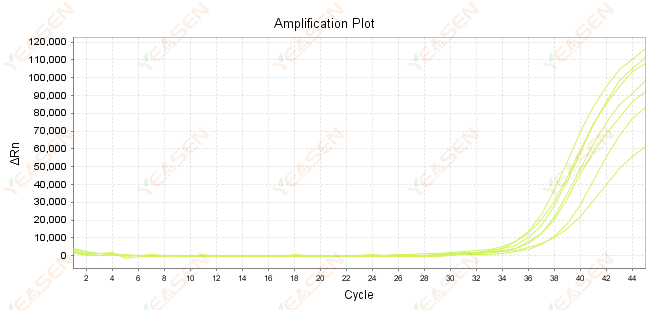
Abbildung 8. Amplifikationsdiagramm
Mögliche Ursachen:
1) Geringe Anzahl an Vorlagen.
2) Geringe Verstärkungseffizienz.
3) Das PCR-Fragment ist zu lang.
4) Vorhandensein von Inhibitoren im Reaktionssystem.
Lösungen:
1) Reduzieren Sie die Verdünnungszeiten oder erhöhen Sie die Vorlagemenge, sodass der Ct-Wert möglichst zwischen 15 und 30 liegt.
2) Optimieren Sie die Reaktionsbedingungen, versuchen Sie ein dreistufiges Amplifikationsverfahren oder entwerfen Sie Primer neu.
3) Die PCR-Produktlänge beträgt 100–150 bp, es wird jedoch empfohlen, 300 bp nicht zu überschreiten.
4) Reproduktion von Vorlagen mit höherer Reinheit.
2.8 Schlechte Wiederholbarkeit von Amplifikationskurven
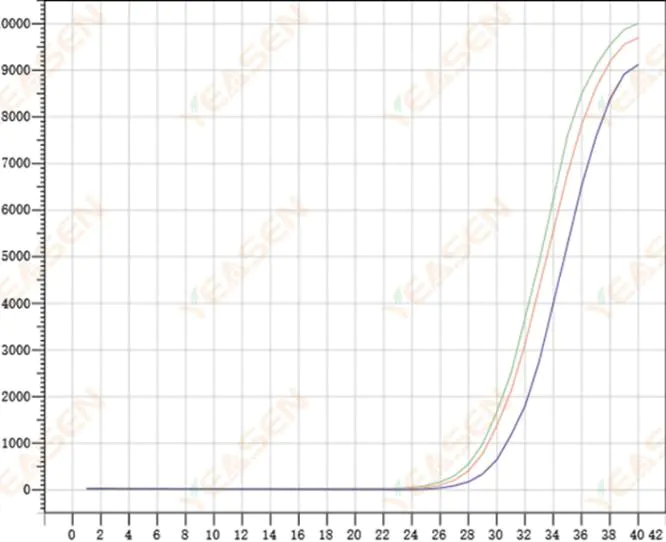
Abbildung 9. Amplifikationsdiagramm
Mögliche Ursachen:
1) Große Spike-Fehler.
2) Reagenzien und Systeme sind nicht gut gemischt.
3) Geringe Kopienzahl der cDNA.
4) Es wurde keine Rox-Kalibrierung verwendet.
Lösungen:
1) Pipetten kalibrieren.
2) Gründliche Durchmischung des Reaktionssystems.
3) Eine niedrige Vorlagenkonzentration, eine schlechte Wiederholbarkeit, 4–6 zusammengesetzte Löcher und 1–2 Abweichungen von größeren Werten können entsprechend verworfen werden.
4) Am besten verwenden Sie die Rox-Kalibrierung. Wenn das verwendete Reagenz kein Rox enthält, muss der Referenzfarbstoff als Keine ausgewählt werden.
2.9 Zufällige Verstärkungskurven
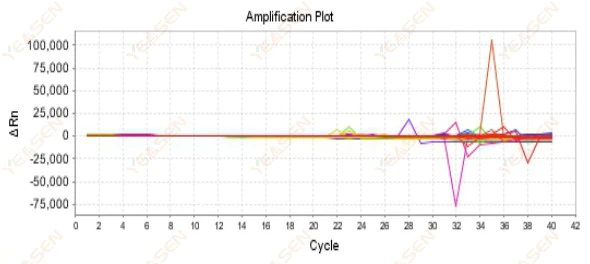
Abbildung 10. Amplifikationsdiagramm
Mögliche Ursachen:
Die Nichtübereinstimmung zwischen Rox-Konzentration und Modell.
Lösungen:
Ändern Sie die Referenzfarbstoffeinstellung am Instrument von ROX auf NONE und prüfen Sie, ob die Amplifikationskurve wieder normal wird.
2.10 NTC-Spitzenwert
2.10.1 Ct>35, Schmelzkurve Tm-Wert <80℃
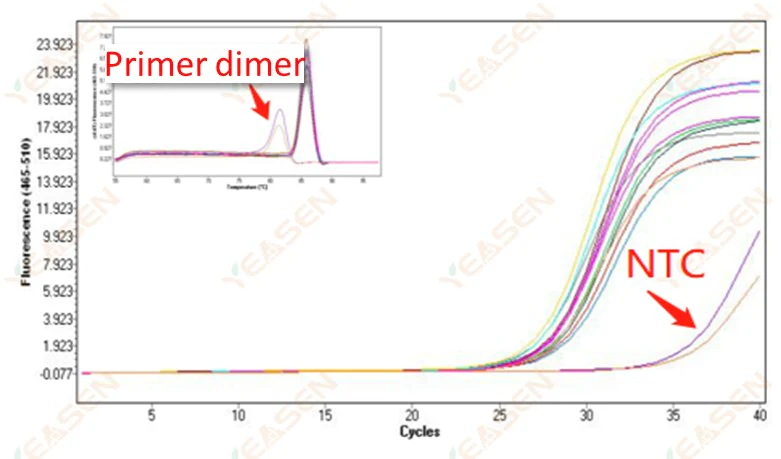
Abbildung 11. Amplifikationsdiagramm
Mögliche Ursachen:
Ergebnisse der Primer-Dimerisierung.
Lösungen:
Optimierte Primer.
2.10.2 Ct-Wert <35, NTC-Schmelzkurve und Gen-Schmelzkurven-Peakform überlappen sich
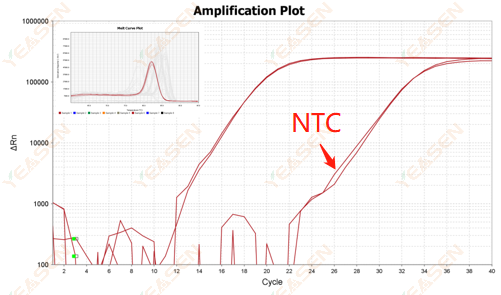
Abbildung 12. Amplifikationsdiagramm
Mögliche Ursachen:
Kontamination des Reaktionssystems.
Lösungen:
Überprüfen Sie die Verschmutzungsquellen einzeln.
3. Abnormale qPCR-Schmelzkurve
3.1 Schmelzkurve mit einem Peak, aber nicht scharf
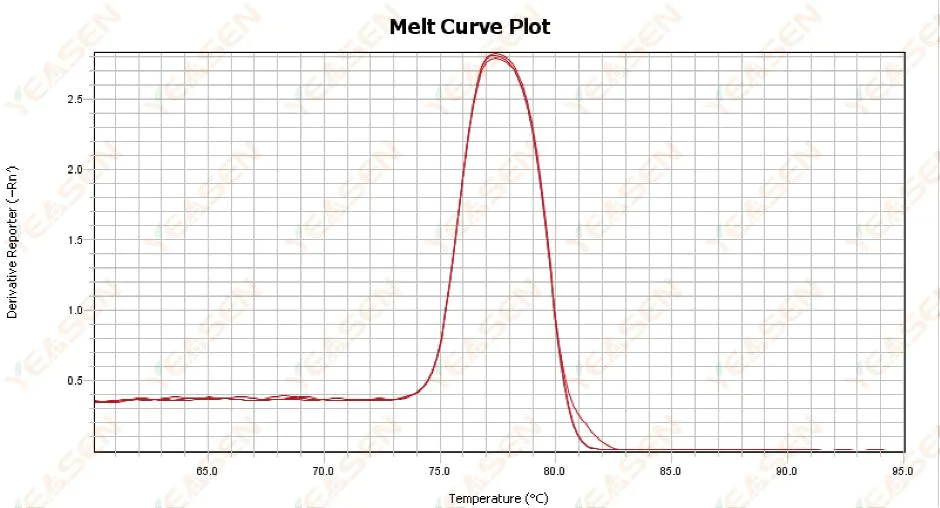
Abbildung 13. Schmelzkurvendiagramm
Mögliche Ursachen:
1) Es hängt mit der Reagenzzusammensetzung und dem Instrumentenmodell zusammen.
2) Vorhandensein einer nichtspezifischen Amplifikation von Fragmenten ähnlicher Größe.
Lösungen:
1) Die Temperaturspanne zwischen Peakbeginn und Peakende beträgt nicht mehr als 7 °C. Die Ergebnisse gelten als verwertbar, d. h. als einzelner Peak.
2) Zur Unterstützung der Bestimmung wurde eine Hochkonzentrations-Agarose-Elektrophorese (z. B. 3 % Agarose) durchgeführt.
3.2 Die Schmelzkurve ist zweispitzig und der untere Peak Tm liegt vor 80°C
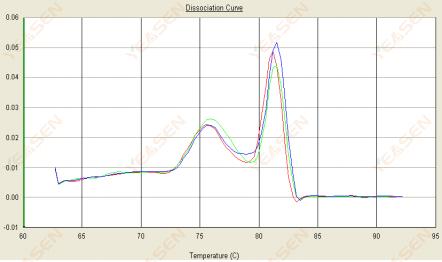
Abbildung 14. Dissoziationskurve
Mögliche Ursachen:
Vorhandensein von Primerdimeren.
Lösungen:
Erhöhen Sie die Annealing-Temperatur, verringern Sie die Primer-Konzentration oder gestalten Sie die Primer neu.
3.3 Die Schmelzkurve hat zwei Gipfel und der untere Gipfel Tm liegt nach 80°C
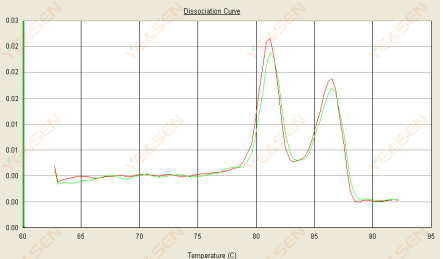
Abbildung 15. Dissoziationskurve
Mögliche Ursachen:
1) Eine schlechte Primer-Spezifität führt zur Amplifikation unspezifischer Produkte.
2) gDNA-Kontamination.
Lösungen:
1) Führen Sie eine Explosionsprüfung der Primer-Spezifität durch und überarbeiten Sie die Konstruktion, falls dies unzureichend ist.
2) Eine Bestätigung durch die NRC-Negativkontrolle (sofern vorhanden) erfordert die erneute Vorbereitung der Vorlage.
3.4 Unordentliche Schmelzkurvenpeaks
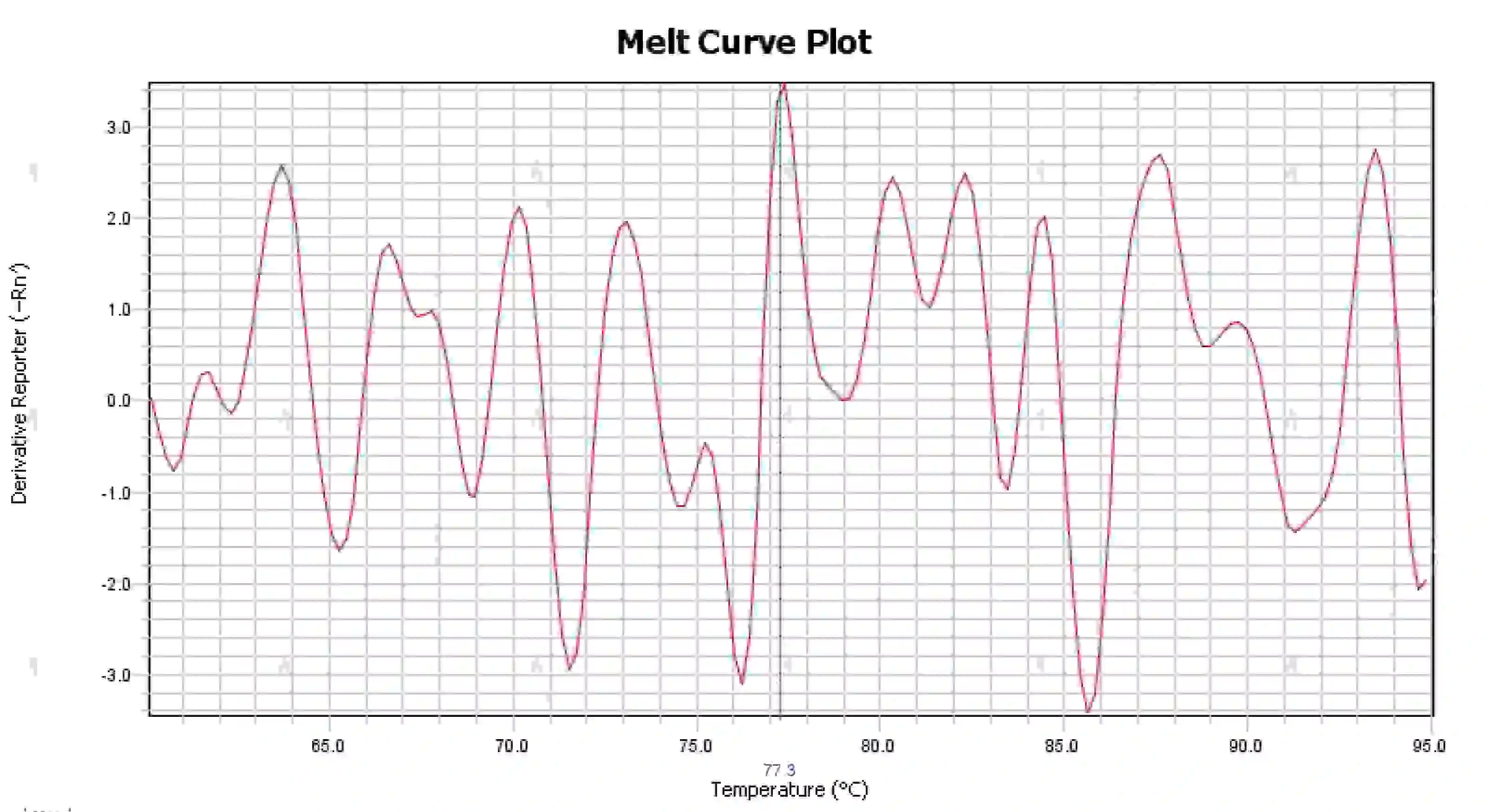
Abbildung 16. Schmelzkurvendiagramm
Mögliche Ursachen:
1) Verunreinigung des Reaktionssystems.
2) Werden Reagenzien hellem Licht oder hohen Temperaturen ausgesetzt, führt dies zum Versagen der Reagenzien.
3) Das Instrument wurde längere Zeit nicht kalibriert.
4) Nichtübereinstimmung zwischen Verbrauchsmaterialien und Instrumenten.
Lösungen:
1) Durch die Kombination der NTC- und NRC-Ergebnisse zur Bestätigung einer Kontamination wird empfohlen, Verunreinigungen aus Wasser, Primern, Enzymen und der Umwelt nacheinander auszuschließen.
2) Es wird empfohlen, mit neuen Reagenzien Vergleichsversuche durchzuführen.
3) Es wird eine regelmäßige Wartung der Instrumentenkalibrierung empfohlen.
4) Bestätigen Sie die Anforderungen des entsprechenden Instruments an Verbrauchsmaterialien.
3.5 Störspitzen am vorderen Ende der Schmelzkurve
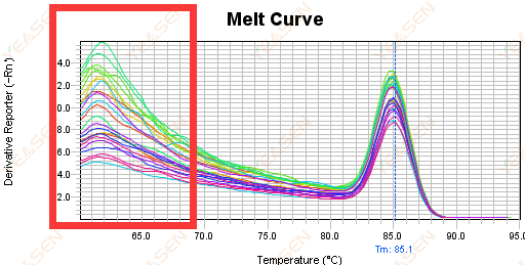
Abbildung 17. Schmelzkurvendiagramm
Mögliche Ursachen:
Die Rox-Konzentration stimmt nicht mit dem Modell überein.
Lösungen:
Es wird empfohlen, die Rox-Korrektur abzubrechen, um zu sehen, ob die Schmelzkurve normal ist.
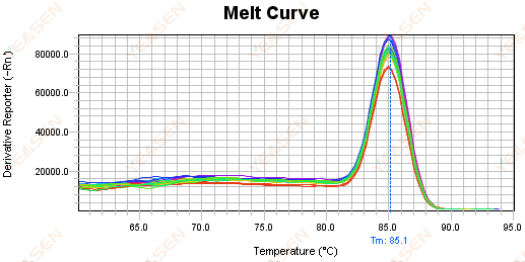
Abbildung 18. Schmelzkurvendiagramm
4. Bestellinformationen
Tabelle 1. Bestellinformationen
| Produktname | Produktcode | Spezifikation |
| Hieff Unicon™ Universal Blue qPCR Master Mix (farbstoffbasiert) | 11184ES03 | 1 ml |
| 11184ES08 | 5×1 ml | |
| 11184ES50 | 50×1 ml | |
| 11184ES60 | 100×1 ml |
5. Verwandte Produkte
Die von
Tabelle 2. Verwandte Produkte
| Produktpositionierung | Produktname | Produktcode |
| Einstufige gDNA-Entfernung und Reverse-Transkription | Hifair™V einstufiger RT-gDNA-Verdau SuperMix für qPCR (Anfrage) | 11142ES |
| Hochempfindlicher Mastermix (mit gDNA-Entfernungsschritt) | Hifair™ III 1. Strang cDNA Synthese SuperMix für qPCR (gDNA Digester plus) | 11141ES |
| Hochempfindliches Kit (mit gDNA-Entfernungsschritt) | Hifair™ III 1. Strang cDNA Synthese Kit (gDNA Digester plus) (Anfrage) | 11139ES |
| Gewöhnlicher quantitativer Mastermix (Sondenmethode) | Hieff™ qPCR TaqMan Sonden-Mastermix (Anfrage) | 11205ES |
6. Zum Thema Lesen
DNase I und ihre Anwendungen in der Biomedizin
Die Richtlinie zur Auswahl der reversen Transkriptase
