Pengurutan berthroughput tinggi, yang sering disebut sebagai teknologi pengurutan generasi berikutnya (NGS), merupakan lompatan maju yang signifikan dari metode pengurutan DNA awal, seperti pengurutan Sanger. NGS memungkinkan pembuatan profil secara bersamaan dari ratusan ribu, bahkan jutaan, urutan molekul asam nukleat. Keunggulannya meliputi throughput yang luar biasa, efektivitas biaya, skalabilitas, dan spektrum aplikasi yang luas, yang menjadikannya sebagai teknologi pengurutan yang dominan di seluruh dunia.
Alur kerja pengurutan NGS mencakup empat fase utama: persiapan sampel, konstruksi pustaka, pengurutan, dan analisis data. Inti dari konstruksi pustaka adalah penempelan sekuens adaptor platform NGS yang terstandarisasi ke kedua ujung DNA genomik yang terfragmentasi. Langkah ini bertujuan untuk menghasilkan pasokan molekul asam nukleat pustaka yang cukup, yang disiapkan untuk pengurutan pada instrumen NGS melalui amplifikasi PCR. Bergantung pada sifat sampel, konstruksi pustaka NGS dapat dikategorikan menjadi konstruksi pustaka DNA dan konstruksi pustaka RNA. Enzim memainkan peran penting dalam eksperimen yang saling berhubungan ini. Jadi, enzim kunci mana yang terlibat dalam proses konstruksi pustaka?
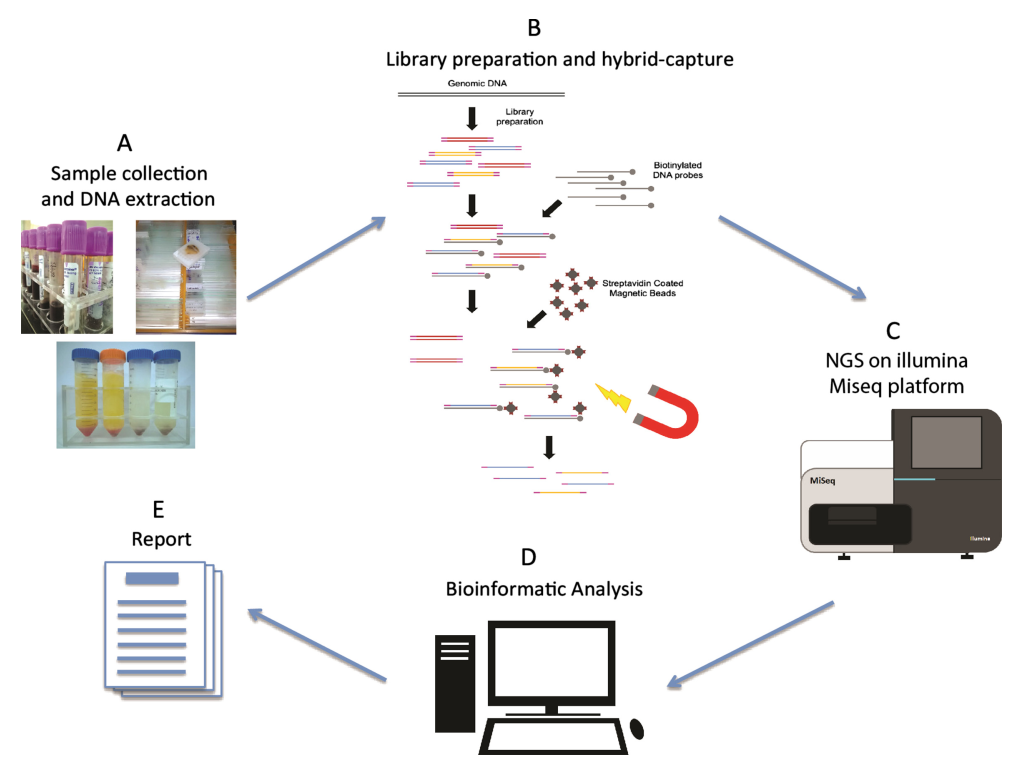
Gambar 1. Alur kerja sekuensing generasi berikutnya[2]
1. Konstruksi perpustakaan DNA dan enzim utamanya
2. Konstruksi perpustakaan RNA dan enzim utamanya
3. Pedoman enzim inti NGS dalam konstruksi perpustakaan DNA & RNA
1. Konstruksi perpustakaan DNA dan enzim utamanya
Dalam proses konstruksi pustaka DNA, konstruksi pustaka adaptor ligasi klon TA merupakan cara teknologi yang paling umum digunakan saat ini. Proses konstruksi pustaka utama adalah sebagai berikut:

Gambar 2. Proses konstruksi pustaka DNA (Illumina)
1.1 Fragmentasi DNA
Sequencer saat ini biasanya memiliki panjang sekuensing dalam kisaran 150-500 pasangan basa (bp). Akibatnya, diperlukan penggunaan metode fragmentasi mekanis atau enzimatik untuk memecah fragmen DNA genomik besar menjadi fragmen yang lebih kecil. Fragmentasi mekanis dapat menyebabkan kehilangan sampel yang relatif tinggi dan melibatkan proses operasional yang lebih rumit. Di sisi lain, pencernaan enzimatik merupakan metode yang umum digunakan untuk memecah DNA genomik. Dibandingkan dengan metode mekanis, pencernaan enzimatik lebih hemat biaya dan mudah, dengan reaksi yang hanya memerlukan periode tertentu setelah penambahan enzim fragmentasi.
Saat ini, terdapat dua jenis fragmen yang digunakan. Satu bergantung pada transposase Tn5, berdasarkan prinsip transposon, sementara yang lain menggunakan campuran endonuklease. Namun, efektivitas fragmen ini dapat dipengaruhi oleh kandungan GC dan preferensi basa DNA. Sebaliknya, fragmen yang dikembangkan oleh
1.2 Perbaikan akhir, dA-Tailing
DNA yang terfragmentasi akan menghasilkan ujung lengket 5'/3' dan DNA ujung tumpul, dan semua ujung lengket perlu diubah menjadi ujung tumpul, termasuk tonjolan 3' yang dihilangkan dan ujung DNA yang menonjol 5' diisi. Saat menggunakan ligasi TA untuk ligasi adaptor, fragmen DNA juga perlu difosforilasi pada ujung 5' dan menambahkan "A" pada ujung 3' agar saling melengkapi dengan adaptor dengan ujung lengket "T".Proses di atas diselesaikan dengan kerja sama DNA polimerase T4, polinukleotida kinase T4, dan Tak DNA polimerase.
DNA polimerase T4 (Cat#12901) memiliki aktivitas DNA polimerase 5'→3', yang dapat mengkatalisis sintesis DNA sepanjang arah 5'→3' dan mengisi ujung 5' yang menonjol. Pada saat yang sama, enzim tersebut juga memiliki aktivitas eksonuklease 3'→5' untuk membelah ujung 3' yang menjorok, sehingga mengubah fragmen DNA yang mengandung ujung lengket menjadi DNA ujung tumpul.
Karena ujung 5' dari primer dan adaptor PCR sintetis biasanya berupa gugus hidroksil, bukan gugus fosfat. Oleh karena itu, kinase polinukleotida T4 (Cat#12902) diperlukan untuk mengkatalisis pemindahan gugus ATP γ -fosfat ke ujung 5' -hidroksil rantai oligonukleotida dengan adanya ATP, sebagai persiapan untuk langkah ligasi adaptor berikutnya.
S-Taq DNA polimerase (Cat#13486) memiliki aktivitas polimerase 5'→3', yang dapat mensintesis DNA dari arah 5'→3'. Sementara itu, ia memiliki aktivitas deoksinukleotidil transferase, yang dapat menambahkan nukleotida "A" ke ujung 3' produk PCR.

Gambar 3. Beberapa enzim terlibat dalam proses perbaikan akhir
Gambar 4. S-taq memiliki efisiensi yang sangat tinggi dalam menambahkan A ke empat basa ATCG di ujung 3' segmen gen yang dideteksi oleh elektroforesis kapiler.
1.3 Ligasi adaptor
Adaptor merupakan komponen penting dari pustaka. Dalam ranah pengurutan Illumina, adaptor tipe-Y yang umum digunakan mencakup urutan P5/P7, Index, dan Rd1/Rd2 SP. Di antara ini, urutan P5/P7 berfungsi untuk dipasangkan dengan urutan yang ada pada chip pengurutan, dengan demikian menambatkan fragmen yang akan dianalisis ke sel aliran untuk menjalankan amplifikasi jembatan. Urutan Index digunakan untuk membedakan antara sampel yang berbeda dalam pustaka campuran yang mengalami pengurutan, sementara Rd1/Rd2 SP menunjukkan daerah untuk mengikat primer pengurutan Read1 dan Read2.
Untuk tugas ligasi adaptor, Ligase DNA T4 (Cat#12996) adalah pilihan standar. Ia menunjukkan kemampuan untuk memperbaiki retakan untai tunggal yang ditemukan pada DNA untai ganda dan menyambung kembali nukleotida yang berdekatan.
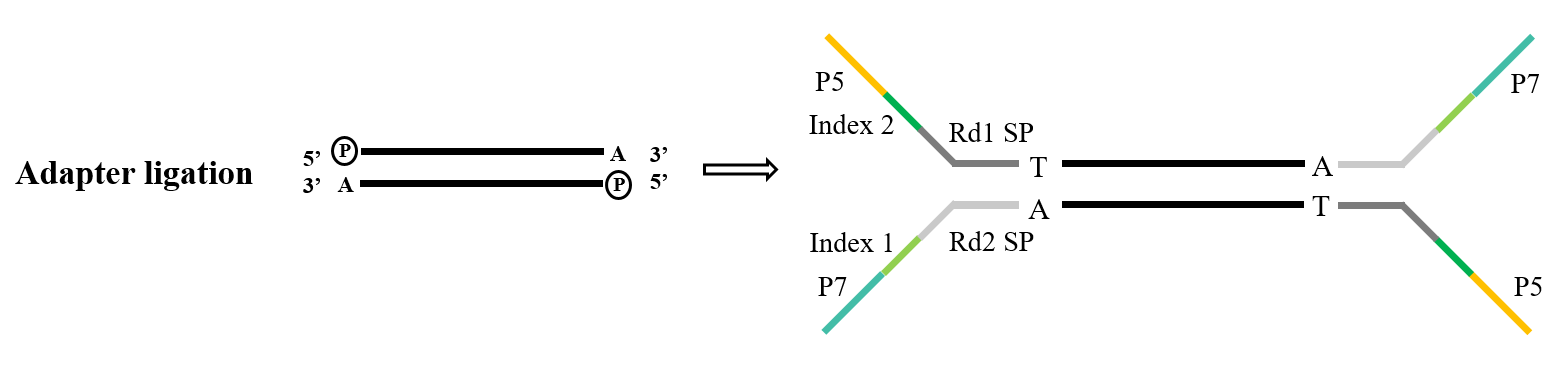
Gambar 5. Proses ligasi adaptor umum (Illumina)

Gambar 6. Verifikasi Mutasi Ligase DNA T4 dengan Mengligasi DNA 170-bp dengan Adaptor 80-bp.
1.4 Amplifikasi PCR
Dapatkan cukup sekuens DNA dengan adaptor melalui reaksi PCR, dan selesaikan pengurutan sekuens asam nukleat sampel pada mesin. Hieff CanaceWaktu Standar Pro High-Fidelity DNA Polymerase (Cat#13476) yang umum digunakan dalam PCR memiliki aktivitas polimerase 5'→3' dan dapat mensintesis DNA dalam arah 5'→3'. Selain itu, enzim ini juga memiliki aktivitas eksonuklease 3'→5', yang dapat mengoreksi kesalahan penggabungan basa selama proses amplifikasi, untuk mengamplifikasi fragmen DNA dengan cepat dan dengan fidelitas tinggi.
2. Konstruksi perpustakaan RNA dan enzim utamanya
Berdasarkan jenis RNA, konstruksi pustaka RNA dapat dibagi menjadi pustaka mRNA, pustaka LncRNA, dan lain-lain. Pustaka RNA konvensional mencakup proses-proses berikut:
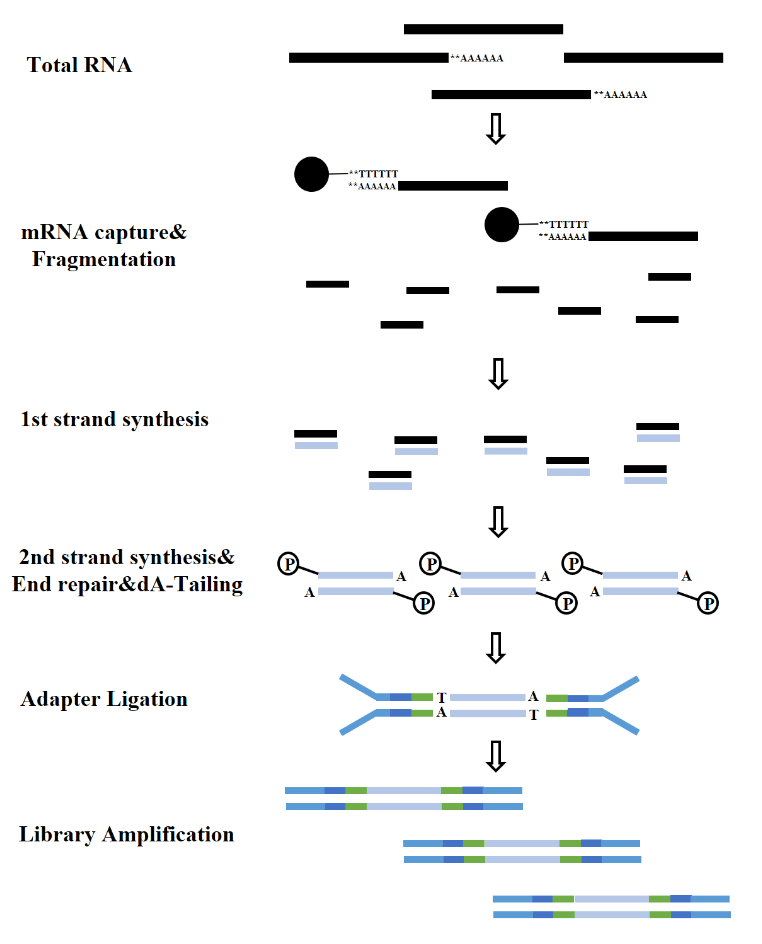
Gambar 7. Proses konstruksi pustaka mRNA (Illumina)
2.1 Pengayaan RNA
Baik dalam menangani eukariota maupun prokariota, RNA ribosom (rRNA) menonjol sebagai RNA yang paling melimpah, yang mencakup hingga 80% dari total kandungan RNA. Saat mengurutkan total RNA dari sampel secara langsung, sebagian besar data pengurutan akan terkait dengan rRNA. Untuk mengurangi gangguan ini, metode pengayaan RNA harus digunakan. Ada dua metode utama untuk ini: pengayaan mRNA berdasarkan oligo-dT dan metode penipisan rRNA.
Pada eukariota, mRNA menunjukkan struktur poli(A) yang berbeda pada ujung 3'. Manik-manik Oligo-dT dapat digunakan untuk menangkap semua mRNA yang ditranskripsi dari sampel, sehingga cocok untuk analisis transkripsi, terutama dengan sampel RNA berkualitas tinggi. Di sisi lain, metode penipisan rRNA memiliki persyaratan yang lebih longgar pada kualitas sampel dan dapat diterapkan pada sampel berkualitas rendah (misalnya, sampel FFPE) dan sampel RNA berkualitas tinggi, serta sampel prokariotik. Pendekatan komersial yang umum digunakan melibatkan penggunaan pencernaan RNase H untuk menghilangkan rRNA, dengan mengikuti langkah-langkah khusus berikut:
- Mensintesis probe oligonukleotida spesifik yang dirancang untuk mengikat rRNA.
- Gunakan RNase H (Cat#12906), yang mampu mendegradasi RNA dalam untai hibrid RNA-DNA, untuk secara selektif menghilangkan rRNA yang terikat pada probe.
- Terakhir, cerna probe DNA dengan DNase I (Kat#10325), yang dapat mendegradasi DNA untai tunggal dan ganda, sehingga secara efektif menghilangkan rRNA. Untuk informasi lebih lanjut tentang DNase I, Anda dapat mengikuti tautan ini.
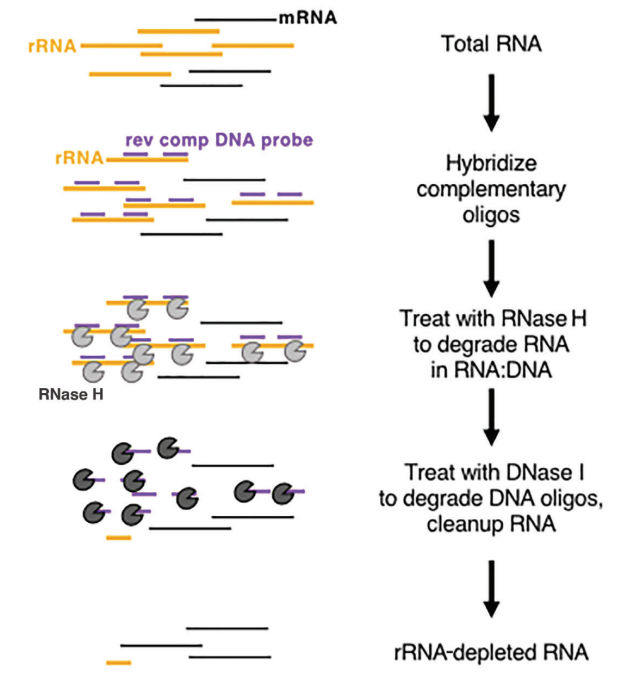
Gambar 8: Diagram skema deplesi rRNA berbasis enzim[5]
2.2 Fragmentasi RNA
Biasanya, di bawah aksi kation logam divalen dan suhu tinggi, fragmen besar RNA dipecah menjadi fragmen kecil.
2.3 Sintesis cDNA untai pertama
Transkripsi balik dari target RNA yang diperoleh ke untai pertama cDNA. Karena RNA mudah terdegradasi oleh RNase yang ada di lingkungan, penggunaan Penghambat RNase (Kat#14672) selama transkripsi balik dapat menghambat aktivitas enzim ini dan melindungi RNA dari degradasi RNase. Pada saat yang sama, transkriptase balik (Cat#11112) digunakan untuk mentranskripsi balik RNA templat menjadi cDNA. Transkriptase balik memiliki aktivitas DNA polimerase yang bergantung pada RNA dan dapat menggunakan RNA sebagai templat untuk mensintesis cDNA dalam arah 5'→3'. Untai tunggal DNA bersifat komplementer terhadap templat RNA.
Selama tanggal 1 sintesis untai cDNA, penggabungan aktinomisin D telah meningkatkan konstruksi pustaka untai spesifik, secara signifikan meningkatkan spesifisitas rantai. Inovasi ini telah menyederhanakan proses eksperimen, menyederhanakannya bagi para peneliti.
Akan tetapi, aktinomisin D memiliki kekurangan: bersifat toksik dan memerlukan perlindungan dari cahaya. Dalam lanskap permintaan yang meningkat saat ini untuk kit penyusun campuran dan pustaka pelat, kebutuhan untuk melindungi dari cahaya menimbulkan keterbatasan pada pengembangan kit pelat.
Untung,

Gambar 9: Rekayasa MMLV untuk mengidentifikasi mutan MMLV yang dapat berkontribusi pada Standed RNA-seq
2.4 Sintesis cDNA untai ke-2
cDNA untai tunggal yang dihasilkan melalui transkripsi balik sangat tidak stabil, sehingga memerlukan sintesis untai kedua cDNA segera di bawah pengaruh DNA polimerase I. Selama sintesis untai kedua ini, RNase H berperan dengan menghilangkan untai RNA dari struktur hibrida RNA-DNA. Ia bekerja sama dengan DNA polimerase I (Cat#12903) untuk memfasilitasi sintesis katalitik untai kedua cDNA yang saling melengkapi. DNA polimerase I memiliki aktivitas DNA polimerase 5'→3' dan, dipandu oleh cetakan dan primer, mensintesis urutan yang melengkapi cDNA untai tunggal dalam arah 5'→3'.
Langkah-langkah selanjutnya dalam proses ini meliputi perbaikan ujung, dA-Tailing, ligasi adaptor, dan amplifikasi PCR, yang semuanya dirinci dalam prosedur konstruksi pustaka DNA dan tidak perlu diulang di sini. Perlu dicatat bahwa setelah transkripsi balik selesai, tidak diperlukan fragmentasi lebih lanjut dari fragmen asam nukleat.
3. Pedoman enzim inti NGS dalam konstruksi perpustakaan DNA & RNA
Tabel 1.Pedoman enzim inti NGS dalam konstruksi perpustakaan DNA & RNA
| Jenis | Posisi produk | Nama Produk | Kucing# |
| Perpustakaan RNA konstruksi | RNA-nya penipisan/sintesis cDNA untai ke-2 | 12906ES | |
| RNA-nya penipisan | 10325ES | ||
| Sintesis cDNA untai pertama | 14672ES | ||
| 11112ES | |||
| Sintesis cDNA untai ke-2 | 12903ES | ||
| Perpustakaan RNA konstruksi & DNA perpustakaan konstruksi | Perbaikan akhir | 12901ES | |
| 12902ES | |||
| dA-Penataan | 13486ES | ||
| Ligasi adaptor | 10301ES | ||
| PCR amplifikasi | 2×Super Canace® II High-Fidelity Mix untuk Amplifikasi Perpustakaan | 12621ES |
Tabel 2.Perpustakaan DNA & RNA Peralatan Persiapan
| Nama | Kucing# | Catatan | |
| DNA | Kit Persiapan Pustaka DNA Hieff NGS | 13577ES | Tumor/Metode Mekanik |
| Kit Persiapan Pustaka DNA Hieff NGS OnePot Pro V2 | 12194ES | Tumor/Metode enzimatik | |
| Hieff NGS Satu Pot Kit Persiapan Pustaka DNA II untuk Illumina | 13490ES | Pathgen/ Enzim/ Waktu reguler (140 menit) | |
| Kit Persiapan Pustaka DNA Flash Hieff NGS OnePot | 12316ES | Pathgen/ Enzimetik/ Sangat Cepat (100 menit) | |
| Kit Persiapan Bersama Perpustakaan DNA & RNA Hieff NGS V2 | 12305ES | Pathgen/ Enzimetik/ DNA & RNA Co-Prep | |
| RNA | Kit Persiapan Pustaka mRNA Mode Ganda Hieff NGS Ultima | 12308ES | Tanpa manik magnetik oligo dT, 11 tabung |
| Kit Persiapan Pustaka mRNA Mode Ganda Hieff NGS Ultima | 12309ES | manik magnet oligo dT plus, 14 tabung | |
| Kit Persiapan Perpustakaan RNA Mode Ganda Hieff NGS® Ultima | 12310ES | Versi campuran, 5 tabung | |
| Kit Persiapan Perpustakaan RNA Hieff NGS ® EvoMax (versi campuran) (aktinomisin D Bebas) | 12340ES | Versi campuran, (Aktinomisin D Bebas) | |
| Kit Deplesi rRNA Hieff NGS® MaxUp (Tanaman) | 12254ES | Tanaman | |
| Kit Deplesi rRNA Manusia Hieff NGS® MaxUp (rRNA & ITS/ETS) | 12257ES | Manusia |
Referensi:
[1] Mardis, Elaine R. Platform Sequencing Generasi Berikutnya[J]. Tinjauan Tahunan Kimia Analitik, 2013, 6(1):287-303.
[2] Gulilat M, Lamb T, Teft WA, dkk. Sequencing generasi berikutnya yang ditargetkan sebagai alat untuk pengobatan resisi[J]. BMC Medical Genomics, 2019, 12(1):81.
[3] Lundberg KS, Dan DS, Adams M, et al. Amplifikasi fidelitas tinggi menggunakan DNA polimerase termostabil yang diisolasi dari Pyrococcus furiosus[J]. Gene, 1991, 108(1):1-6.
[4] Miyazaki K. Fragmentasi DNA acak dengan endonuklease V: aplikasi untuk pengocokan DNA[J]. Penelitian Asam Nukleat, 2002, 30(24):e139.
[5] Baldwin A, Morris AR, Mukherjee N. Metode Mudah, Hemat Biaya, dan Skalabel untuk Menguras RNA Ribosom Manusia untuk RNA-seq[J]. Protokol Saat Ini, 2021, 1(6):e176.

