Arbeiten Sie entlang beider Linien, miRNA-Expressionsanalyse wird einfacher
Die Länge reifer miRNA beträgt nur etwa 20 nt. Normalerweise deckt der Vorwärtsprimer die gesamte Länge ab oder weist sogar einen überschüssigen Teil auf, und der Rückwärtsprimer kann nirgendwo platziert werden. Die aktuelle Marktlösung besteht darin, die Länge des Reverse-Transkripts während der Reverse-Transkription zu erhöhen. Zu den gängigen Methoden gehören die Tailing-Methode und die Stem-Loop-Methode.
1. Einleitung
2. Empfohlene Produkte
3. Verwandte Produktliste
1. Einleitung
MicroRNAs (miRNAs) sind einzelsträngige kleine RNAs mit einer Größe von etwa 21–23 Basen, die nach der Verarbeitung durch das Dicer-Enzym aus einzelsträngigen RNA-Vorläufern mit einer Haarnadelstruktur von etwa 70–90 Basen erzeugt werden. miRNA bindet hauptsächlich spezifisch an die nicht-kodierende Region am 3'-Ende der Zielgen-mRNA und reguliert den Translationsprozess der posttranskriptionellen Zielgen-mRNA, wodurch die mRNA-Translation blockiert und der mRNA-Abbau gefördert wird, was zu einer verringerten Proteinexpression führt. Die Rolle von miRNAs ist an der Entstehung und Entwicklung verschiedener Herz-Kreislauf-Erkrankungen wie Ontogenese, Gewebedifferenzierung, Zellproliferation und Apoptose beteiligt.
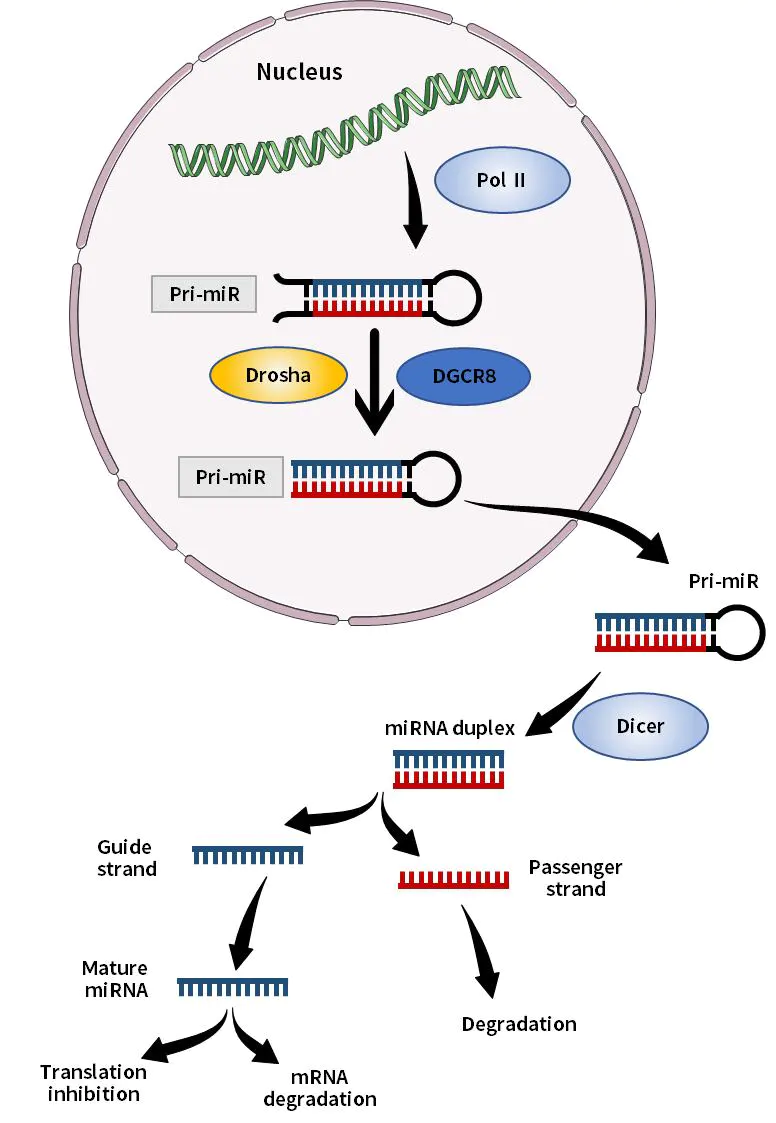
Abbildung 1. Schematische Darstellung der miRNA-Synthese in vivo
Die Länge reifer miRNA beträgt nur etwa 20 nt. Normalerweise deckt der Vorwärtsprimer die gesamte Länge ab oder weist sogar einen überschüssigen Teil auf, und der Rückwärtsprimer kann nirgendwo platziert werden. Die aktuelle Marktlösung besteht darin, die Länge des Reverse-Transkripts während der Reverse-Transkription zu erhöhen. Zu den gängigen Methoden gehören die Tailing-Methode und die Stem-Loop-Methode.
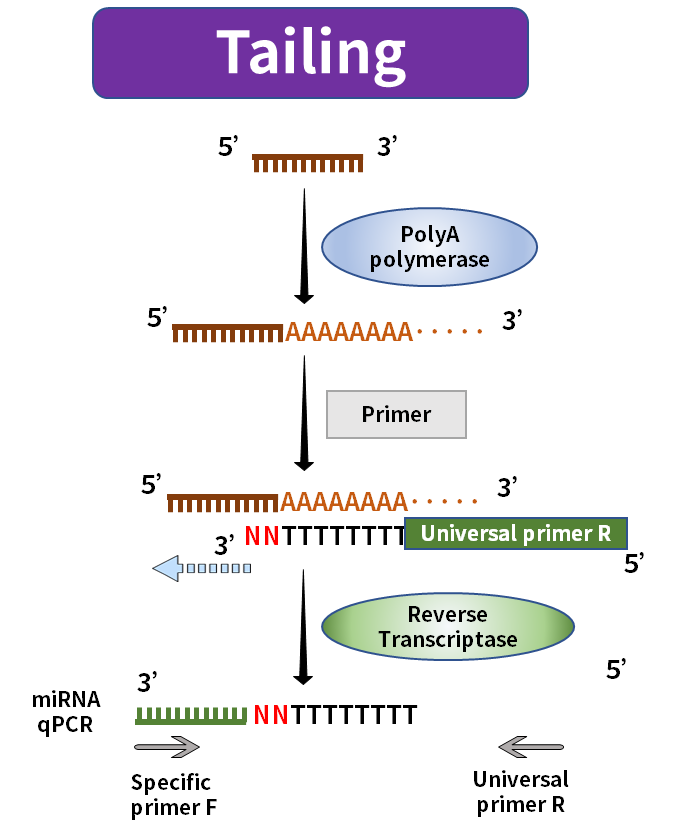
Abbildung 2. miRNA-Synthese durch Tailing-Methode
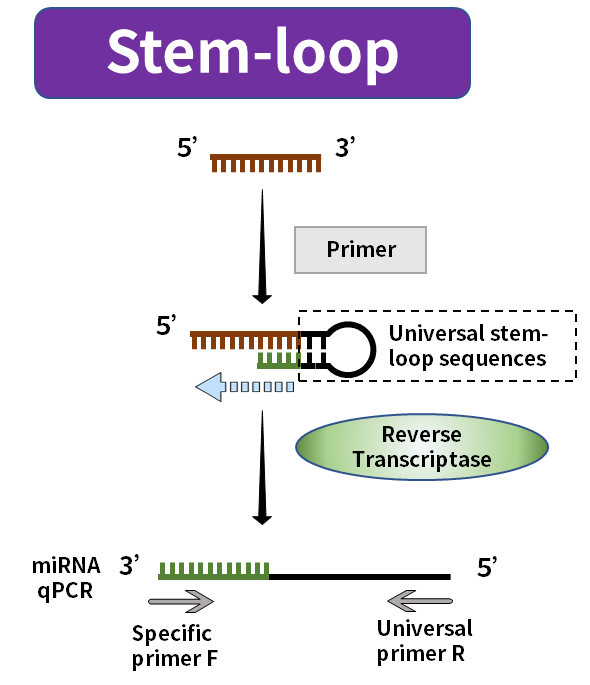
Abbildung 3. miRNA-Synthese mit der Stammschleifenmethode
Bei der Tailing-Methode wird PolyA-Polymerase verwendet, um der reifen miRNA einen Poly(A)-Schwanz hinzuzufügen, und Oligo-dT mit der universellen Sequenz wird als Reverse-Transkriptionsprimer verwendet, um die verlängerte miRNA zu synthetisieren, die dem ersten Strang der cDNA entspricht. Bei der Stem-Loop-Methode wird die in die Stem-Loop-Struktur gefaltete universelle Sequenz als Primer verwendet, um den ersten Strang der cDNA zu synthetisieren, der der verlängerten miRNA entspricht. Die Stem-Loop-Struktur wird während des anschließenden Amplifikationsprozesses bei einer geeigneten Temperatur geöffnet. und mit den entsprechenden Amplifikationsprimern kombiniert.
Tabelle 1.Vergleich von Tailing und Stem-Loop
| Kategorie | Beschattung | Stiel-Schleife |
| Anwendbarkeit | Geeignet für die gleichzeitige Untersuchung mehrerer verschiedener miRNAs | Geeignet für den genauen quantitativen Nachweis einiger miRNAs in einer Probe |
| Vorteil | Eine einzige Reverse Transkription kann mehrere miRNAs mit hohem Durchsatz erkennen | Durch Reverse Transkription kann jeweils nur eine miRNA mit hoher Spezifität nachgewiesen werden |
| System | Zusammen mit der beteiligten miRNA kann die reverse Transkription im selben Reaktionssystem mit hoher Genauigkeit | Intern beteiligte miRNAs werden getrennt und in den beiden Systemen rücktranskribiert, was fehleranfällig ist |
| Spezifität | ★★★★☆ | ★★★★★ |
| Empfindlichkeit | ★★★★★ | ★★★★☆ |
| Zeit | ★★★★☆ | ★★★★★ |
2. Empfohlene Produkte
2.1 Yeasen miRNA Schwanzinversion quantitative Kombination (11148ES&11171ES)
Abbildung 4. 11148ES-Hifair™ miRNA 1. Strang cDNA Synthese Kit
Das Kit verwendet die Tailing-Methode zur Reverse-Transkription des miRNA-Erststrangs cDNA. Der 2×Hifair™ miRNA RT-Puffer im Produkt enthält alle Rohstoffe und Primer für die miRNA-Tailing-Reaktion und die Reverse-Transkriptionsreaktion und wurde sorgfältig optimiert. Er kann sicherstellen, dass die Poly(A)-Modifikation und die Reverse-Transkriptionsprozesse des miRNA-3'-Endes gleichzeitig effizient durchgeführt werden.
Abbildung 5. 11171ES -Hieff™ miRNA Universal qPCR SYBR Master Mix
Eine neue Generation vorgemischter Fluoreszenz-Quantitative-PCR-Nachweisreagenzien, die speziell für den quantitativen miRNA-Nachweis entwickelt wurden und einen speziellen passiven Referenzfarbstoff ROX enthalten. Sie sind für alle qPCR-Instrumente geeignet. Die ROX-Konzentration muss auf verschiedenen Instrumenten nicht angepasst werden. Die Amplifikation kann einfach während der Vorbereitungsreaktion durch Hinzufügen von Primern und Vorlagen zum System durchgeführt werden. Die DNA-Polymerase verwendet eine chemisch modifizierte Hot-Start-Polymerase und arbeitet mit einem speziellen Puffersystem zusammen, wodurch die Reaktionsspezifität und -empfindlichkeit erhöht wird und eine genaue Quantifizierung in einem größeren Bereich möglich ist.
2.2 Produktleistung
2.2.1 Kompatibel mit qPCR-Instrumenten aller Plattformen

Abbildung 6. Kompatible Ergebnisse mit verschiedenen qPCR-Instrumenten
Unter Verwendung der Gesamt-RNA von 293T-Zellen als Vorlage wurde die Reverse Transkription mit dem Hifair™ miRNA 1st Strand cDNA Synthesis Kit (Kat.-Nr. 11148ES) durchgeführt.Die erhaltene cDNA wurde 10-fach verdünnt und dann mit Hieff™ miRNA Universal qPCR SYBR Master Mix (Kat.-Nr. 11171ES) die internen Referenzgene von hsa-miR-let-7b-5p, hsa-miR-let-7c-5p und U6 erkannt.
2.2.2 Hohe Nachweisrate, bis zu 10 pg
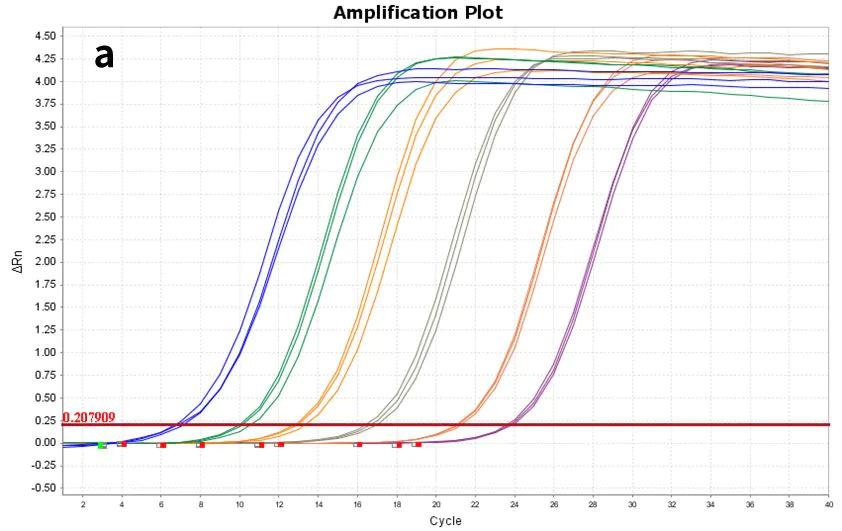
Abbildung 7. Amplifikationsdiagramm
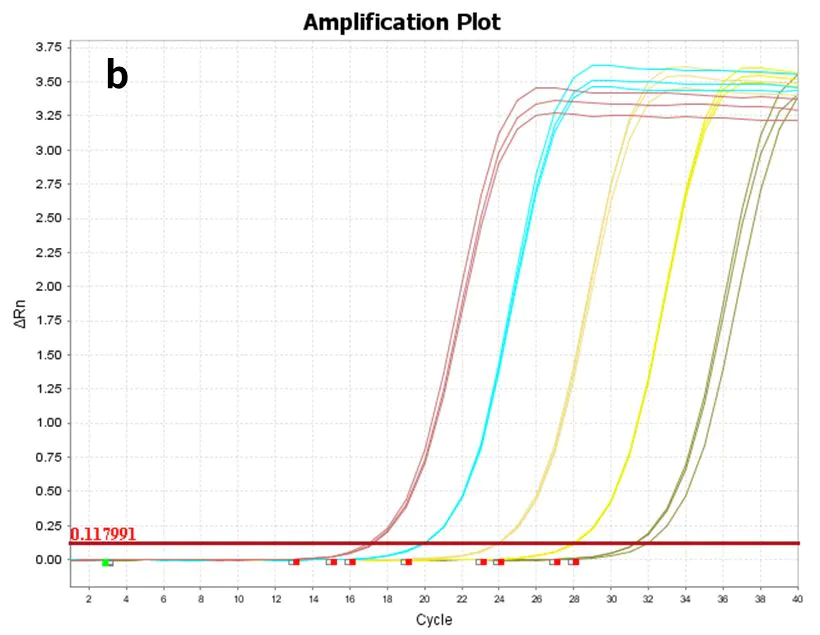
Abbildung 8. Amplifikationsdiagramm
Unter Verwendung von synthetischem hsa-miR-let-7e-5p(a) und 293T-Zell-Gesamt-RNA(b) als Vorlagen, verdünnt auf die folgenden Gradienten: 60 Kopien bis 606 Kopien (6 Gradienten) und 10 pg-100 ng (5 Gradienten), rücktranskribiert mit dem Hifair™ miRNA 1st Strand cDNA Synthesis Kit (Kat.-Nr. 11148ES), und die resultierende cDNA wurde mit dem Hieff™ miRNA Universal qPCR SYBR Master Mix (Kat.-Nr. 11171ES) auf die Genexpression von hsa-miR-let-7e-5p geprüft.
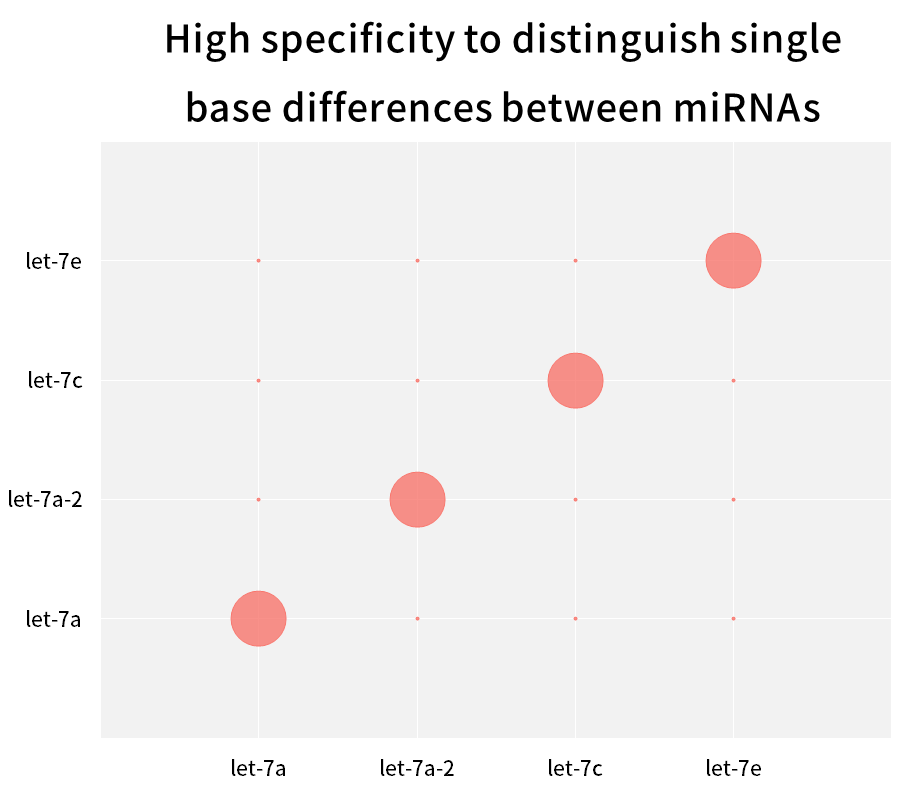
2.2.3 Hohe Spezifität, kann einzelne Basenunterschiede zwischen miRNAs in der gleichen Familie unterscheiden
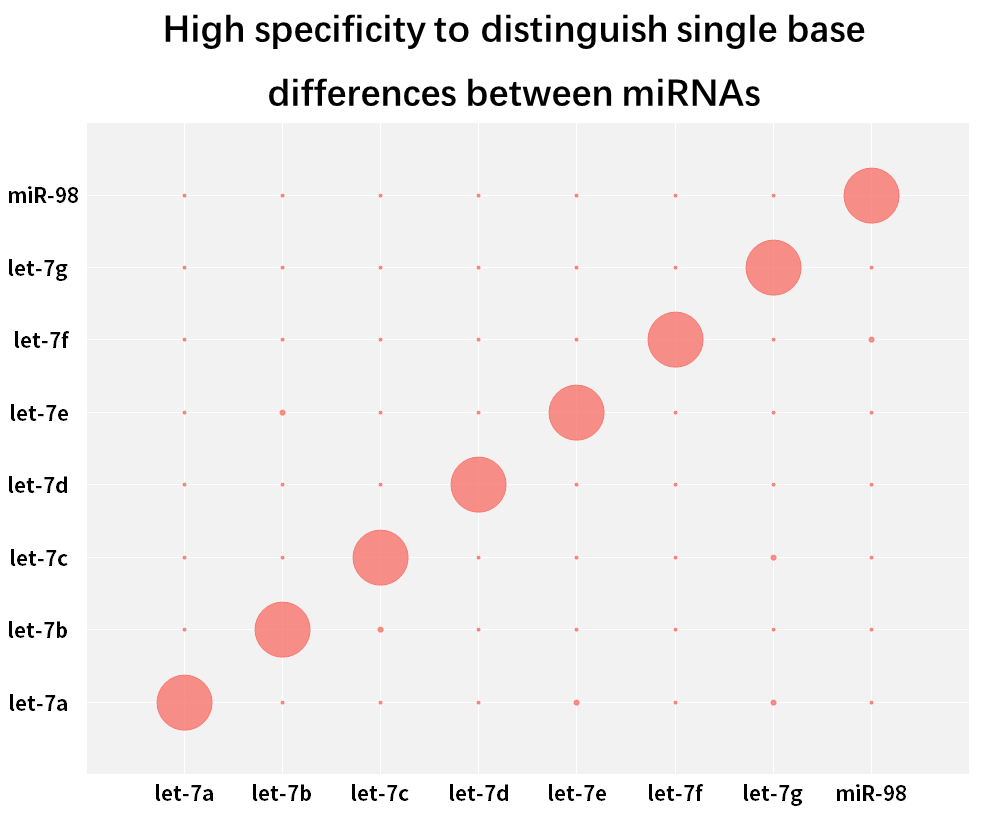
Abbildung 9. Unterscheiden Sie einzelne Basenunterschiede zwischen miRNAs
Die menschliche hsa-let-7-Familie hat mehrere miRNAs, die sich nur in wenigen Basen oder sogar nur in einer einzigen Base voneinander unterscheiden. Synthetische miRNAs für jede Let-7-Isoform (hsa-miR-let-7a~Let-7i, has-miR-98) wurden mit dem Hifair™ miRNA 1st Strand cDNA Synthesis Kit (Kat.-Nr. 11148ES) in cDNA rücktranskribiert, wobei der Hieff™ miRNA Universal qPCR SYBR Master Mix (Kat.-Nr. 11171ES) verwendet wurde, um diese miRNAs separat mit unterschiedlichen Primern zu amplifizieren und die Übereinstimmungsrate mit Gleichung 2-ΔCT x 100 % zu berechnen. Die Ergebnisse zeigen, dass eng verwandte Mitglieder der Subtypfamilie effektiv unterschieden werden können.
2.2.4 Hervorragende Verstärkungseffizienz
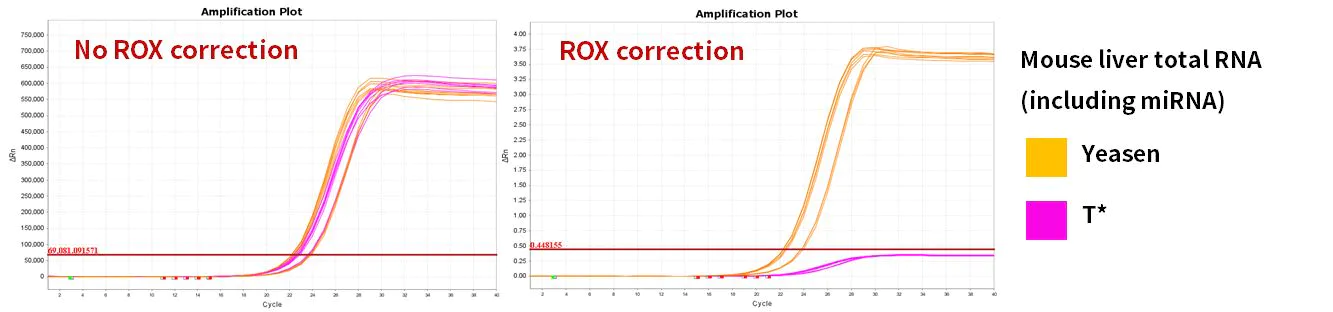
Abbildung 10. Verstärkungseffizienz
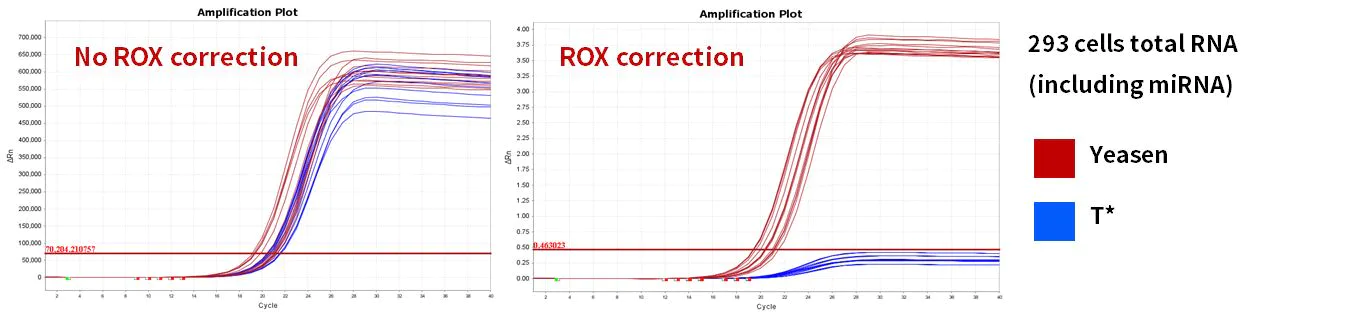
Abbildung 11. Verstärkungseffizienz
Die Gene miR-let-7b-5p, miR-let-7c-5p, miR-let-7e-5p und miR-let-7f-5p wurden unter Verwendung von menschlichen 293T-Zellen und Gesamt-RNA aus der Leber von Mäusen als Vorlagen amplifiziert. Die Ergebnisse zeigten, dass das Hifair™ miRNA 1st Strand cDNA Synthesis Kit (Kat.-Nr. 11148ES) im Vergleich zu ähnlichen Produkten eine hervorragende Leistung mit dem Reaktionssystem des Hieff™ miRNA Universal qPCR SYBR Master Mix (Kat.-Nr. 11171ES) erzielte.
2.2.5 Ausgezeichnete Stabilität

Abbildung 12. Produktstabilitätsanalyse bei 37 °C für 7 Tage

Abbildung 13. Analyse der Ergebnisse nach 50 Frost-Tau-Zyklen
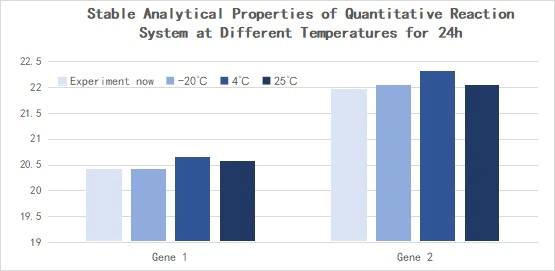
Abbildung 14. Stabile analytische Eigenschaften des quantitativen Reaktionssystems bei verschiedenen Temperaturen für 24 Stunden
Die Reverse Transkription wurde mit dem Hifair™ miRNA 1st Strand cDNA Synthesis Kit (Kat.-Nr. 11148ES) unter Verwendung von 293T-Zell-Gesamt-RNA als Vorlage durchgeführt, und die erhaltene cDNA wurde 10-fach verdünnt und mit dem Hieff™ miRNA Universal qPCR SYBR Master Mix (Kat.-Nr. 11171ES) nachgewiesen. Expression von hsa-miR-let-7b-5p, hsa-miR-let-7c-5p, hsa-miR-let-7e-5p. △Ct<0,5.
2.3 Yeasen miRNA Stamm-Schleifen-Inversion quantitative Kombination (11139ES&11170ES)
Abbildung 15. 11139ES-Hifair™ III 1. Strang cDNA Synthese Kit (gDNA Digester plus)
Das Kit wurde auf Basis der Hifair™ III Reverse Transkriptase entwickelt.Die cDNA-Synthesegeschwindigkeit des Enzyms ist hoch und die thermische Stabilität ist deutlich verbessert. Gleichzeitig erhöht das Enzym die Affinität zur Vorlage und eignet sich für die Reverse Transkription einer kleinen Anzahl von Vorlagen und Genen mit geringer Kopienzahl. Die Fähigkeit der Hifair™ III Reverse Transkriptase, cDNA in voller Länge zu synthetisieren, wurde ebenfalls verbessert, wodurch die Synthese von cDNA bis zu 19,8 kb möglich ist.

Abbildung 16. 11170ES-Hieff™ miRNA Universal qPCR SYBR Master Mix
Dieses Produkt ist ein spezieller Mastermix für die miRNA-Quantifizierung mit der chimären Fluoreszenzmethode SYBR Green I. Da miRNA-Sequenzen kurz sind und miRNA-Sequenzen in derselben Familie häufig sehr ähnlich sind, ist bei der Quantifizierung Spezifität äußerst wichtig. Dieses Produkt basiert auf Hot-Start-DNA-Polymerase mit optimiertem Puffer, wodurch die unspezifische Amplifikation stark reduziert werden kann. Gleichzeitig macht der spezielle ROX-Referenzfarbstoff den Mastermix für alle qPCR-Instrumente geeignet, und es ist nicht erforderlich, ROX auf verschiedenen Instrumenten anzupassen. Die Amplifikation kann durch Hinzufügen von Primern und Vorlagen bei der Vorbereitung des Reaktionssystems durchgeführt werden.
2.3.1 Kompatibel mit qPCR-Instrumenten aller Plattformen
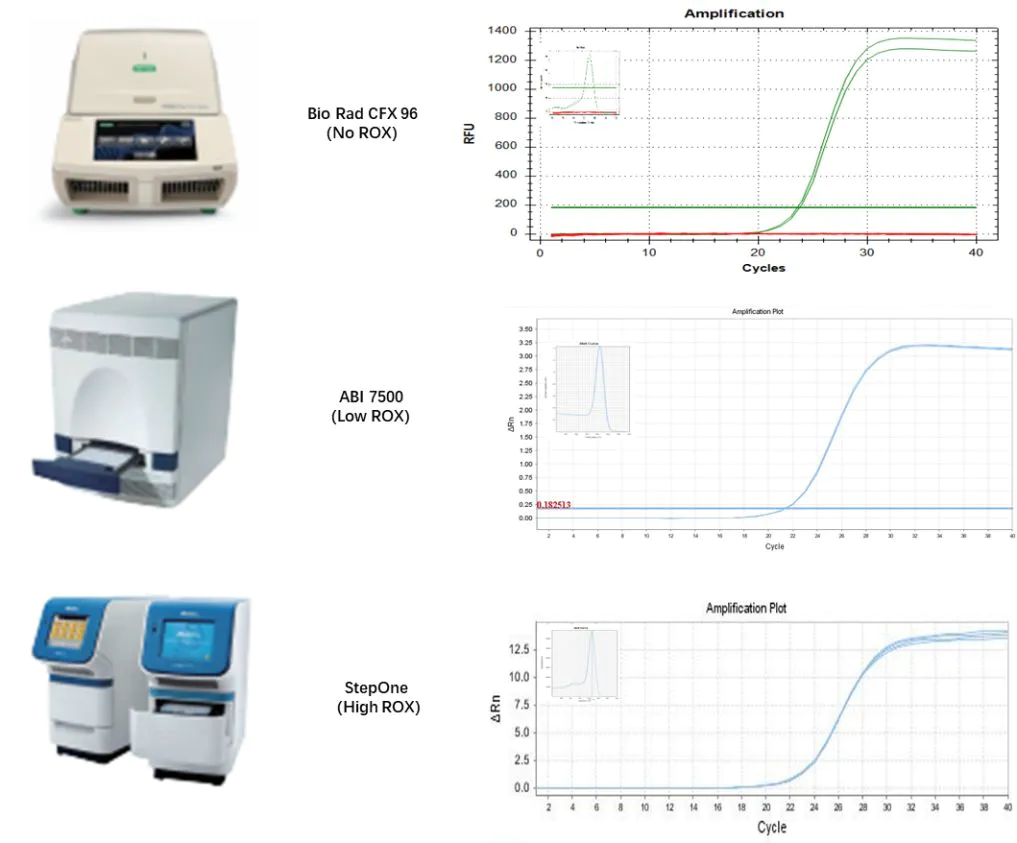
Abbildung 17. Kompatible Ergebnisse mit verschiedenen qPCR-Instrumenten
Unter Verwendung der Gesamt-RNA von Mausleberzellen als Vorlage wurde die Reverse Transkription mit Hifair™ III 1st Strand durchgeführt.
cDNA-Synthesekit (gDNA Digester plus) (Kat.-Nr. 11139ES), und die erhaltene cDNA wurde 40-fach verdünnt und mit dem Hieff™ miRNA Universal qPCR SYBR Master Mix (Kat.-Nr. 11170ES) wurde das menschliche mmu-miR-125a-Gen amplifiziert.
2.3.2 Hohe Empfindlichkeit, bis zu 10 pg

Abbildung 18. Empfindlichkeitserkennung
Unter Verwendung der Gesamt-RNA von Mausleberzellen als Vorlage wurde eine Reverse Transkription mit dem Hifair™ III 1st Strand cDNA Synthesis Kit (gDNA digester plus) (Kat.-Nr. 11139ES) durchgeführt und die erhaltene cDNA wurde seriell 10-fach verdünnt (Bereich 10 pg/µl). -0,1 µg/µl), amplifizieren die Gene mmu-miR-125a, mmu-miR-132 und mmu-miR-351 murinen Ursprungs.
2.3.3 Hohe Spezifität, kann einzelne Basenunterschiede zwischen miRNAs in der gleichen Familie unterscheiden
Abbildung 19. Unterscheiden Sie einzelne Basenunterschiede zwischen miRNAs
Die menschliche hsa-let-7-Familie hat mehrere miRNAs, die sich in wenigen Basen voneinander unterscheiden oder sogar nur in einer einzigen Base. Diese miRNAs wurden separat mit verschiedenen Primern unter Verwendung des Hieff™ miRNA Universal qPCR SYBR Master Mix (Kat.-Nr. 11170ES) amplifiziert und die Übereinstimmungsrate wurde mit der Formel 2^-Δct x 100 % berechnet.
2.3.4 Gute Wiederholbarkeit

Abbildung 20. Wiederholbarkeitserkennung
Unter Verwendung der Gesamt-RNA menschlicher 293T-Zellen als Vorlage für die Reverse Transkription wurde die Reverse Transkription mit dem Hifair™ III 1st Strand cDNA Synthesis Kit (gDNA digester plus) (Kat.-Nr. 11139ES) durchgeführt, die erhaltene cDNA 50-fach verdünnt und mit dem Hieff™ miRNA Universal qPCR SYBR Master Mix (Kat.-Nr. 11170ES) das menschliche hsa-let-7a-Gen in einem Replikationsexperiment mit 90 Vertiefungen amplifiziert.
2.3.5 Gute Stabilität

Abbildung 21.Stabilitätsanalyse von 11170ES bei verschiedenen Temperaturen für 14 Tage
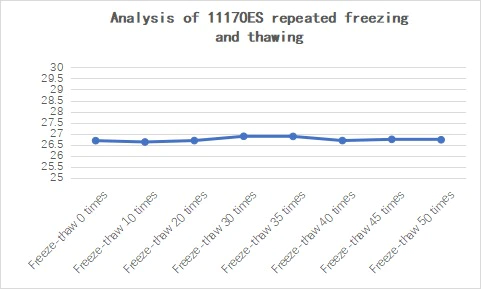
Abbildung 22. Analyse von 11170ES nach wiederholtem Einfrieren und Auftauen

Abbildung 23. Stabile Analyseeigenschaften des quantitativen Reaktionssystems bei verschiedenen Temperaturen für 24 Stunden
Die Gesamt-RNA von Mausleberzellen wurde als Vorlage für die Reverse Transkription mit dem Hifair™ III 1st Strand cDNA Synthesis Kit (gDNA digester plus) (Kat.-Nr. 11139ES) verwendet. Die erhaltene cDNA wurde 100-fach verdünnt, um das murine mmu- miR-132-Gen zu amplifizieren.
3. Verwandte Produktliste
Tabelle 2. Liste der zugehörigen Produkte