توالی یابی با توان عملیاتی بالا، که اغلب به عنوان فناوری توالی یابی نسل بعدی (NGS) نامیده می شود، نشان دهنده یک جهش به جلو نسبت به روش های اولیه توالی یابی DNA، مانند توالی یابی سانگر است. NGS امکان بررسی همزمان صدها هزار، اگر نگوییم میلیون ها، توالی مولکول اسید نوکلئیک را فراهم می کند. مزایای آن شامل توان عملیاتی استثنایی، مقرون به صرفه بودن، مقیاس پذیری و طیف وسیعی از کاربردها است که آن را به عنوان تکنولوژی توالی غالب در سراسر جهان معرفی می کند.
گردش کار توالی یابی NGS شامل چهار مرحله اولیه است: آماده سازی نمونه، ساخت کتابخانه، توالی یابی و تجزیه و تحلیل داده ها. مرکز ساخت کتابخانه، اتصال توالی های آداپتور پلت فرم استاندارد NGS به هر دو انتهای DNA ژنومی تکه تکه شده است. هدف این مرحله تولید یک منبع کافی از مولکول های اسید نوکلئیک کتابخانه ای است که برای توالی یابی بر روی ابزار NGS از طریق تقویت PCR آماده شده است. بسته به ماهیت نمونه، ساخت کتابخانه NGS را می توان به ساخت کتابخانه DNA و ساخت کتابخانه RNA دسته بندی کرد. آنزیم ها نقشی محوری در این آزمایشات به هم پیوسته دارند. بنابراین، کدام آنزیم های کلیدی در فرآیند ساخت کتابخانه نقش دارند؟
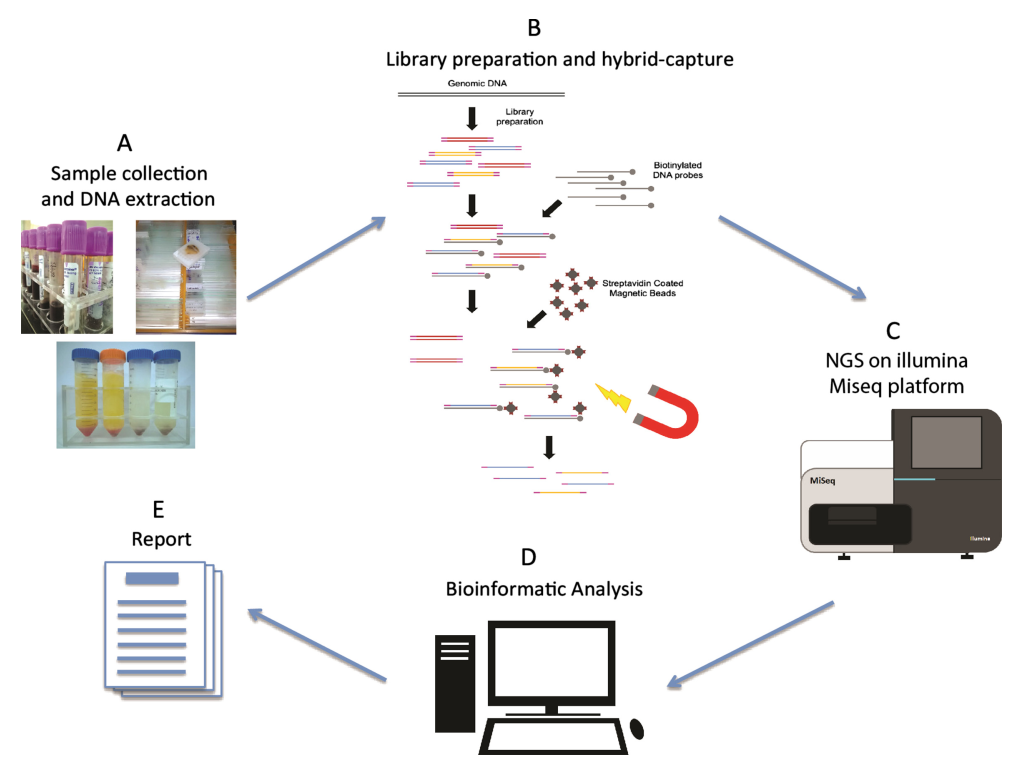
شکل 1. جریان کار توالی یابی نسل بعدی[2]
1. ساخت کتابخانه DNA و آنزیم های کلیدی آن
2. ساخت کتابخانه RNA و آنزیم های کلیدی آن
3. راهنمای آنزیم های هسته NGS در ساخت کتابخانه DNA و RNA
1. ساخت کتابخانه DNA و آنزیم های کلیدی آن
در فرآیند ساخت کتابخانه DNA، ساخت کتابخانه آداپتور بستن کلون TA رایج ترین وسیله تکنولوژی مورد استفاده در حال حاضر است. فرآیند اصلی ساخت کتابخانه به شرح زیر است:

شکل 2. فرآیند ساخت کتابخانه DNA (Illumina)
1.1 تکه تکه شدن DNA
ترتیبدهندههای فعلی معمولاً دارای طول توالی در محدوده 150-500 جفت باز (bp) هستند. در نتیجه، استفاده از روش های تکه تکه شدن مکانیکی یا آنزیمی برای شکستن قطعات بزرگ DNA ژنومی به قطعات کوچکتر ضروری می شود. تکه تکه شدن مکانیکی می تواند منجر به از دست دادن نمونه نسبتاً زیاد شود و شامل فرآیند عملیاتی پیچیده تری است. از سوی دیگر، هضم آنزیمی یک روش متداول برای قطعه قطعه کردن DNA ژنومی است. در مقایسه با روشهای مکانیکی، هضم آنزیمی مقرونبهصرفهتر و سادهتر است، به طوری که واکنش تنها به یک دوره مشخص پس از افزودن آنزیم قطعهسازی نیاز دارد.
در حال حاضر، در درجه اول دو نوع قطعه در حال استفاده وجود دارد. یکی بر اساس ترانسپوزون Tn5 بر اساس اصول ترانسپوزون تکیه می کند، در حالی که دیگری از مخلوطی از اندونوکلئازها استفاده می کند. با این حال، اثربخشی این قطعات را می توان تحت تأثیر محتوای GC و ترجیحات پایه DNA قرار داد. در مقابل، قطعات توسعهیافته توسط Yeasen (Cat#12917) یک اثر هضم پایدار ارائه میدهند و اولویت مکان قابلتوجهی پایینتری نسبت به Tn5 transposase نشان میدهند. آنها به طور مداوم نتایج توالی یابی عالی را برای انواع مختلف نمونه های DNA، از جمله نمونه های FFPE ارائه می دهند.
1.2 پایان تعمیر، dA-Tailing
DNA تکه تکه شده، انتهای چسبنده 5'/3' و DNA انتهایی را تولید می کند، و تمام انتهای چسبنده باید به انتهای انتها تبدیل شوند، از جمله برآمدگی های 3' برداشته شده و انتهای DNA برآمده 5' پر می شود. هنگام استفاده از بستن TA برای بستن آداپتور، قطعه DNA نیز باید در انتهای 5 فسفریله شود و A به انتها "5" اضافه شود. آداپتور با انتهای چسبنده "T".فرآیند فوق با همکاری DNA پلیمراز T4، پلی نوکلئوتید کیناز T4 و طاق DNA پلیمراز
DNA پلیمراز T4 (Cat#12901) دارای فعالیت DNA پلیمراز 5'→3' است که می تواند سنتز DNA را در جهت 5'→3' کاتالیز کند و انتهای بیرون زده 5' را پر کند. در همان زمان، آنزیم همچنین دارای فعالیت اگزونوکلئاز 3'→5' برای بریدن انتهای آویزان 3' است، در نتیجه قطعات DNA حاوی انتهای چسبنده را به DNA انتهایی تبدیل می کند.
از آنجایی که انتهای 5' پرایمرها و آداپتورهای PCR مصنوعی معمولاً به جای گروه های فسفات، گروه های هیدروکسیل هستند. بنابراین، پلی نوکلئوتید کیناز T4 (Cat#12902) برای کاتالیز کردن انتقال گروههای γ-فسفات ATP به انتهای هیدروکسیل 5' زنجیره الیگونوکلئوتیدی در حضور ATP، برای آمادهسازی برای مرحله بعدی بستن آداپتور، مورد نیاز است.
S-Taq DNA پلیمراز (Cat#13486) دارای فعالیت پلیمراز 5'→3' است که می تواند DNA را از جهت 5'→3' سنتز کند. در همین حال، دارای فعالیت دئوکسی نوکلئوتیدیل ترانسفراز است که می تواند یک نوکلئوتید "A" را به انتهای 3' محصول PCR اضافه کند.

شکل 3. چندین آنزیم در فرآیند ترمیم نهایی نقش دارند
شکل 4. S-taq دارای کارایی بسیار بالایی در افزودن A به چهار پایه ATCG انتهای 3' بخش های ژنی است که توسط الکتروفورز مویرگی شناسایی شده است.
1.3 بستن آداپتور
آداپتورها یک جزء حیاتی از کتابخانه را تشکیل می دهند. در قلمرو توالی یابی Illumina، آداپتورهای نوع Y که معمولاً مورد استفاده قرار می گیرند، دنباله های P5/P7، Index و Rd1/Rd2 SP را در بر می گیرند. در این میان، توالی P5/P7 به هدف جفت شدن با توالی موجود در تراشه توالییابی عمل میکند، در نتیجه قطعاتی را که قرار است بر روی سلول جریان تجزیه و تحلیل شوند برای اجرای تقویت پل، لنگر میاندازند. توالی Index برای تمایز بین نمونههای مختلف در کتابخانه مختلط که در معرض توالییابی قرار میگیرند استفاده میشود، در حالی که Rd1/Rd2 SP مناطقی را برای اتصال پرایمرهای توالییابی Read1 و Read2 نشان میدهد.
برای کار بستن آداپتور، T4 DNA لیگاز (Cat#12996) انتخاب استاندارد است این قابلیت را نشان می دهد که بریدگی های تک رشته ای موجود در DNA دو رشته ای را ترمیم کند و نوکلئوتیدهای مجاور را دوباره به هم متصل کند.
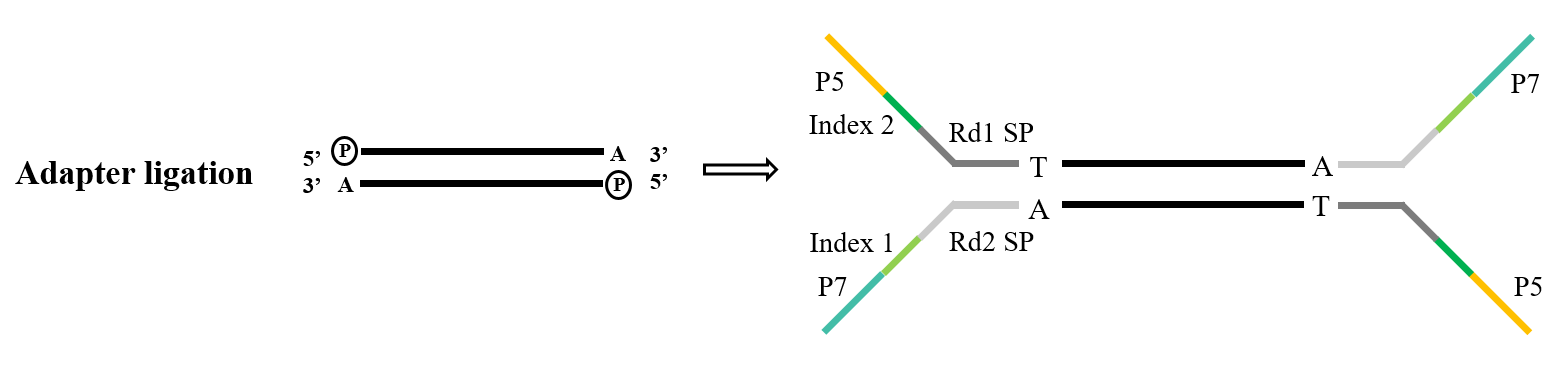
شکل 5. فرآیند بستن آداپتور عمومی (Illumina)

شکل 6. تایید جهش T4 DNA لیگاز با بستن DNA 170 جفت باز با آداپتورهای 80 جفت باز.
1.4 تقویت PCR
توالی های DNA کافی را با آداپتورها از طریق واکنش PCR بدست آورید و توالی یابی نمونه توالی اسید نوکلئیک را روی دستگاه کامل کنید. هیف کناسTM Pro High-Fidelity DNA Polymerase (Cat#13476) که معمولاً در PCR استفاده می شود دارای فعالیت پلیمراز 5'→3' است و می تواند DNA را در جهت 5'→3' سنتز کند. علاوه بر این، فعالیت اگزونوکلئاز 3'→5' را نیز دارد که می تواند ادغام اشتباه بازها را در طول فرآیند تقویت تصحیح کند تا قطعات DNA را به سرعت و با وفاداری بالا تقویت کند.
2. ساخت کتابخانه RNA و آنزیم های کلیدی آن
با توجه به انواع RNA، ساخت یک کتابخانه RNA را می توان به کتابخانه mRNA، کتابخانه LncRNA و غیره تقسیم کرد. کتابخانه مرسوم RNA شامل فرآیندهای زیر است:
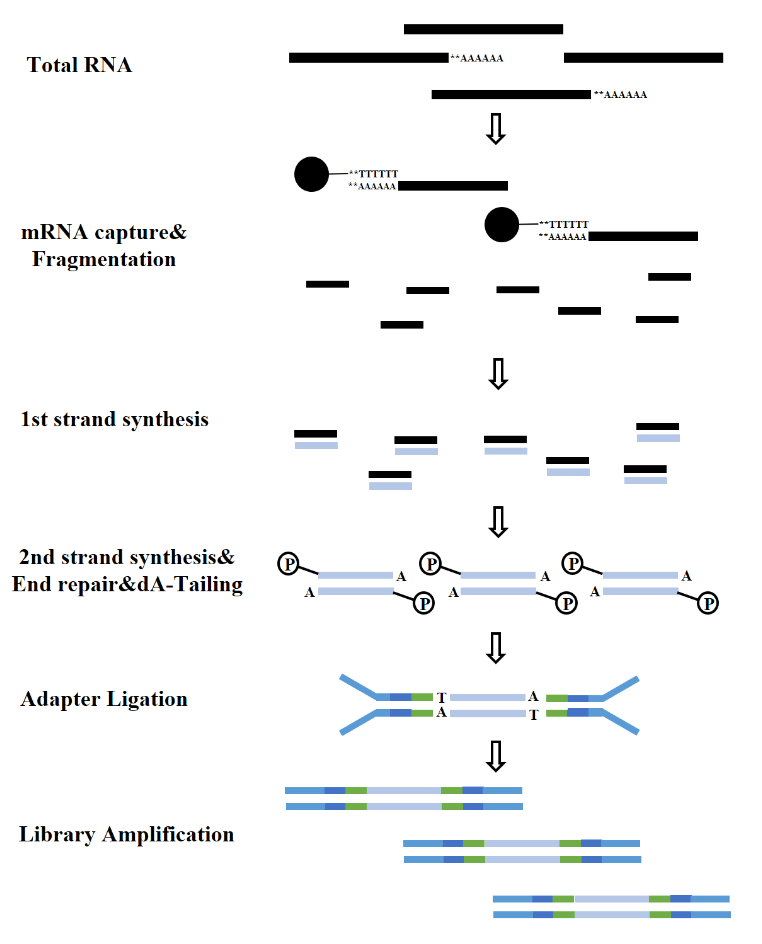
شکل 7. فرآیند ساخت کتابخانه mRNA (Illumina)
2.1 غنی سازی RNA
چه با یوکاریوت ها و چه پروکاریوت ها، RNA ریبوزومی (rRNA) به عنوان فراوان ترین RNA برجسته است و تا 80٪ از کل محتوای RNA را تشکیل می دهد. هنگام تعیین توالی RNA کل یک نمونه به طور مستقیم، بخش قابل توجهی از داده های توالی یابی به rRNA مربوط می شود. برای کاهش این تداخل، باید از روش غنی سازی RNA استفاده شود. دو روش اصلی برای این کار وجود دارد: غنیسازی mRNA بر اساس روشهای تخلیه oligo-dT و rRNA.
در یوکاریوت ها، mRNA ساختار پلی (A) مشخصی را در انتهای 3' نشان می دهد. دانه های Oligo-dT را می توان برای گرفتن تمام mRNA های رونویسی شده از نمونه استفاده کرد و آن را برای تجزیه و تحلیل رونویسی، به ویژه با نمونه های RNA با کیفیت بالا، مناسب می کند. از سوی دیگر، روشهای تخلیه rRNA الزامات ملایمتری در کیفیت نمونه دارند و میتوانند هم برای نمونههای با کیفیت پایین (مانند نمونههای FFPE) و هم نمونههای RNA با کیفیت بالا و هم برای نمونههای پروکاریوتی اعمال شوند. رویکرد تجاری متداول استفاده از هضم RNase H برای حذف rRNA، به دنبال این مراحل خاص است:
- پروب های الیگونوکلئوتیدی خاصی را که برای اتصال به rRNA طراحی شده اند، سنتز کنید.
- از RNase H (Cat#12906)، که قادر به تجزیه RNA در رشته هیبریدی RNA-DNA است، برای حذف انتخابی rRNA متصل به پروب ها استفاده کنید.
- در نهایت، پروب های DNA را با هضم کنید DNase I (Cat#10325)، که می تواند DNA تک رشته ای و دو رشته ای را تخریب کند و به طور موثر rRNA را از بین ببرد. برای اطلاعات بیشتر در مورد DNase I می توانید این لینک را دنبال کنید.
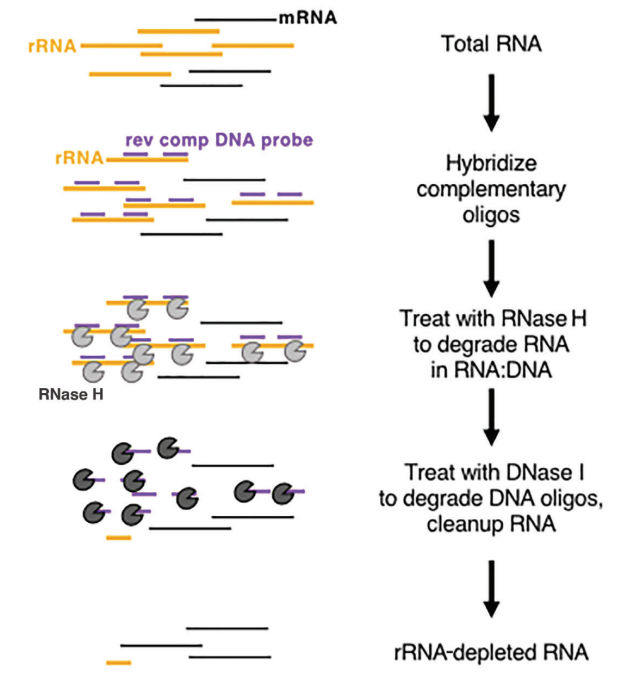
شکل 8: نمودار شماتیک کاهش rRNA مبتنی بر آنزیم[5]
2.2 تکه تکه شدن RNA
معمولاً تحت تأثیر کاتیون های فلزی دو ظرفیتی و دمای بالا، قطعات بزرگ RNA به قطعات کوچک شکسته می شوند.
2.3 سنتز cDNA رشته اول
رونویسی معکوس RNA هدف به دست آمده به اولین رشته cDNA. از آنجایی که RNA به راحتی توسط RNaseهای موجود در محیط تجزیه می شود، استفاده از مهارکننده RNase (Cat#14672) در طول رونویسی معکوس می تواند فعالیت این آنزیم ها را مهار کند و RNA را از تخریب RNase محافظت کند. در عین حال، ترانس کریپتاز معکوس (Cat#11112) برای رونویسی معکوس RNA الگو به cDNA استفاده شد. رونوشت معکوس دارای فعالیت DNA پلیمراز وابسته به RNA است و می تواند از RNA به عنوان الگویی برای سنتز cDNA در جهت 5'→3' استفاده کند. تک رشته DNA مکمل الگوی RNA است.
در طول 1 سنتز cDNA رشتهای، ادغام اکتینومایسین D به طور غیرقابل انکاری ساخت کتابخانههای خاص رشته را بهبود بخشیده است و ویژگی زنجیره را به طور قابلتوجهی افزایش میدهد. این نوآوری فرآیند آزمایشی را ساده کرده و آن را برای محققان ساده کرده است.
با این حال، اکتینومایسین D معایبی دارد: سمیت دارد و نیاز به محافظت در برابر نور دارد. در چشمانداز امروزی که تقاضای فزاینده برای کیتهای ساخت کتابخانه پیشآمیخته و صفحهای دارد، ضرورت محافظت در برابر نور، محدودیتهایی در پیشرفت کیت صفحه ایجاد میکند.
خوشبختانه پلتفرم Yeasen ZymeEditor یک جهش یافته آنزیم MMLV (Inquiry) را معرفی کرده است که جایگزین عملکرد اکتینومایسین D می شود. کیت (Cat: 12340ES) با بی بو، غیر سمی و n ساخته شده است.o نیاز به اجتناب نور این ویژگی زنجیره ای برتر را ارائه می دهد و نگرانی های مربوط به سلامت و حساسیت به نور را از بین می برد.

شکل 9: مهندسی MMLV برای شناسایی جهشیافتههای MMLV که میتوانند در Standed RNA-seq کمک کنند
2.4 سنتز cDNA رشته دوم
cDNA تک رشته ای تولید شده از طریق رونویسی معکوس بسیار ناپایدار است و سنتز فوری رشته دوم cDNA را تحت تأثیر DNA پلیمراز I ضروری می کند. در طول این سنتز رشته دوم، RNase H با حذف رشته RNA از ساختار هیبریدی RNA-DNA وارد بازی می شود. با هماهنگی کار می کند DNA پلیمراز I (Cat#12903) برای تسهیل سنتز کاتالیزوری رشته دوم مکمل cDNA. DNA پلیمراز I دارای فعالیت DNA پلیمراز 5'→3' است و با هدایت یک الگو و آغازگر، دنباله ای را سنتز می کند که cDNA تک رشته ای را در جهت 5'→3' تکمیل می کند.
مراحل بعدی در فرآیند شامل ترمیم انتهایی، dA-Tailing، بستن آداپتور و تقویت PCR است که همه آنها در روش ساخت کتابخانه DNA به تفصیل آمده و نیازی به تکرار در اینجا نیست. شایان ذکر است که پس از تکمیل رونویسی معکوس، دیگر نیازی به تکه تکه شدن قطعه اسید نوکلئیک نیست.
3. راهنمای آنزیم های هسته NGS در ساخت کتابخانه DNA و RNA
Yeasen یک شرکت بیوتکنولوژی است که در تحقیق، توسعه، تولید و فروش سه معرف اصلی بیولوژیکی فعالیت می کند: مولکول ها، پروتئین ها و سلول ها. شرکت Yeasen Biotech انواع آنزیم های مربوط به ساخت کتابخانه NGS را تولید می کند. از نمودار زیر می توانید مناسب ترین محصول ساخت کتابخانه را انتخاب کنید.
جدول 1.راهنمای آنزیم های هسته NGS در ساخت کتابخانه DNA و RNA
| تایپ کنید | موقعیت یابی محصول | نام محصول | گربه# |
| کتابخانه RNA ساخت و ساز | rRNA تخلیه / سنتز cDNA رشته 2 | 12906ES | |
| rRNA تهی شدن | 10325ES | ||
| سنتز cDNA رشته 1 | 14672ES | ||
| 11112ES | |||
| سنتز cDNA رشته 2 | 12903ES | ||
| کتابخانه RNA ساخت و ساز & DNA کتابخانه ساخت و ساز | پایان تعمیر | 12901ES | |
| 12902ES | |||
| dA-Tailing | 13486 ES | ||
| بستن آداپتور | 10301ES | ||
| PCR تقویت | 12621ES |
جدول 2.کتابخانه DNA و RNA کیت آماده سازی
| نام | گربه# | یادداشت ها | |
| DNA | کیت آماده سازی کتابخانه DNA Hieff NGS | 13577ES | تومور / روش مکانیکی |
| کیت آماده سازی کتابخانه DNA Hieff NGS OnePot Pro V2 | 12194ES | روش توموری/آنزیمی | |
| Hieff NGS OnePot کیت آماده سازی کتابخانه DNA II برای ایلومینا | 13490ES | پاتژن / آنزیمی / زمان منظم (140 دقیقه) | |
| Hieff NGS OnePot Flash DNA Library Prep Kit | 12316ES | پاتژن / آنزیمی / فوق سریع (100 دقیقه) | |
| Hieff NGS DNA&RNA Library Co-Prep Kit V2 | 12305ES | پاتژن / آنزیم / DNA و RNA Co-Prep | |
| RNA | کیت آماده سازی کتابخانه mRNA دو حالته Hieff NGS Ultima | 12308ES | بدون مهره های مغناطیسی الیگو dT، 11 لوله |
| کیت آماده سازی کتابخانه mRNA با حالت دوگانه Hieff NGS Ultima | 12309ES | دانه های مغناطیسی الیگو dT پلاس، 14 لوله | |
| کیت آماده سازی کتابخانه RNA دو حالته Hieff NGS® Ultima | 12310ES | نسخه پیش میکس، 5 تیوب | |
| کیت آماده سازی کتابخانه Hieff NGS ® EvoMax RNA (نسخه پیش میکس) (اکتینومایسین D رایگان) | 12340ES | نسخه پیش میکس، (اکتینومایسین D رایگان) | |
| Hieff NGS® MaxUp rRNA Depletion Kit (گیاه) | 12254ES | گیاه | |
| Hieff NGS® MaxUp Human rRNA Depletion Kit (rRNA & ITS/ETS) | 12257ES | انسان |
مراجع:
[1] Mardis، Elaine R. پلتفرم های توالی یابی نسل بعدی [J]. بررسی سالانه شیمی تجزیه، 2013، 6 (1): 287-303.
[2] Gulilat M، Lamb T، Teft WA، و همکاران. توالی یابی نسل بعدی را به عنوان ابزاری برای پزشکی اصلاحی هدف قرار داد [J]. BMC Medical Genomics، 2019، 12 (1): 81.
[3] Lundberg KS، Dan DS، Adams M، و همکاران. تقویت با وفاداری بالا با استفاده از یک DNA پلیمراز مقاوم در برابر حرارت جدا شده از Pyrococcus furiosus [J]. ژن، 1991، 108 (1): 1-6.
[4] میازاکی K. تکه تکه شدن تصادفی DNA با اندونوکلئاز V: کاربرد در هم زدن DNA [J]. تحقیقات اسیدهای نوکلئیک، 2002، 30(24): e139.
[5] Baldwin A, Morris AR, Mukherjee N. روشی آسان، مقرون به صرفه و مقیاس پذیر برای تخلیه RNA ریبوزومی انسان برای RNA-seq[J]. پروتکل های جاری، 2021، 1(6):e176.

