High-throughput sequencing, vaak aangeduid als next-generation sequencing (NGS) technologie, vertegenwoordigt een significante sprong voorwaarts ten opzichte van de eerste DNA-sequencing methoden, zoals Sanger sequencing. NGS maakt het mogelijk om gelijktijdig honderdduizenden, zo niet miljoenen, nucleïnezuurmolecuulsequenties te profileren. De voordelen ervan omvatten uitzonderlijke throughput, kosteneffectiviteit, schaalbaarheid en een breed spectrum aan toepassingen, waardoor het de dominante sequencing technologie wereldwijd is geworden.
De NGS-sequencingworkflow omvat vier primaire fasen: monstervoorbereiding, bibliotheekconstructie, sequentiebepaling en data-analyse. Centraal in bibliotheekconstructie staat de bevestiging van gestandaardiseerde NGS-platformadaptersequenties aan beide uiteinden van gefragmenteerd genomisch DNA. Deze stap is gericht op het genereren van een ruime voorraad bibliotheeknucleïnezuurmoleculen, voorbereid voor sequentiebepaling op het NGS-instrument door middel van PCR-amplificatie. Afhankelijk van de aard van het monster kan NGS-bibliotheekconstructie worden gecategoriseerd in DNA-bibliotheekconstructie en RNA-bibliotheekconstructie. Enzymen spelen een cruciale rol in deze onderling verbonden experimenten. Dus, welke sleutelenzymen zijn betrokken bij het proces van bibliotheekconstructie?
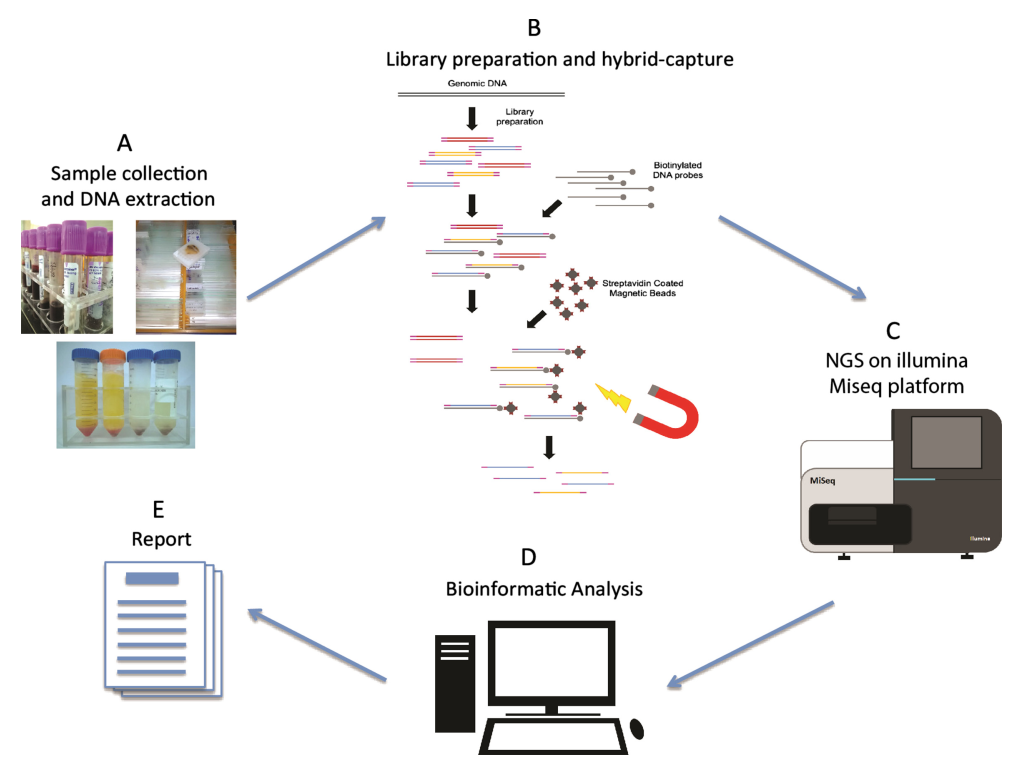
Figuur 1. Next-generation sequencing-workflow[2]
1. De constructie van een DNA-bibliotheek en de belangrijkste enzymen
2. Constructie van RNA-bibliotheek en zijn sleutelenzymen
3. Richtlijn voor NGS-kernenzymen bij de constructie van DNA- en RNA-bibliotheken
1. De constructie van een DNA-bibliotheek en de belangrijkste enzymen
In het proces van DNA-bibliotheekconstructie is TA-kloonligatieadapterbibliotheekconstructie momenteel de meest gebruikte technologie. Het belangrijkste bibliotheekconstructieproces is als volgt:

Figuur 2. DNA-bibliotheekconstructieproces (Illumina)
1.1 DNA-fragmentatie
De huidige sequencers hebben doorgaans een sequentielengte in het bereik van 150-500 baseparen (bp). Als gevolg hiervan is het noodzakelijk om mechanische of enzymatische fragmentatiemethoden te gebruiken om grote genomische DNA-fragmenten op te splitsen in kleinere. Mechanische fragmentatie kan leiden tot relatief veel monsterverlies en omvat een ingewikkelder operationeel proces. Aan de andere kant is enzymatische digestie een veelgebruikte methode voor het fragmenteren van genomisch DNA. In vergelijking met mechanische methoden is enzymatische digestie kosteneffectiever en eenvoudiger, waarbij de reactie slechts een bepaalde periode vereist na de toevoeging van het fragmentatie-enzym.
Momenteel zijn er voornamelijk twee typen fragmenten in gebruik. De ene is gebaseerd op de Tn5-transposase, gebaseerd op transposonprincipes, terwijl de andere een mengsel van endonucleasen gebruikt. De effectiviteit van deze fragmenten kan echter worden beïnvloed door de GC-inhoud en basevoorkeuren van het DNA. Daarentegen zijn de fragmenten die zijn ontwikkeld door
1.2 Eindreparatie, dA-Tailing
Het gefragmenteerde DNA genereert 5'/3' sticky ends en blunt-end DNA, en alle sticky ends moeten worden omgezet in blunt ends, inclusief 3' overhangen verwijderd en 5'-uitstekende DNA uiteinden opgevuld. Bij gebruik van TA ligatie voor adapter ligatie, moet het DNA fragment ook worden gefosforyleerd aan het 5' uiteinde en "A" worden toegevoegd aan het 3' uiteinde om complementair te zijn aan de adapter met het "T" sticky end.Het bovenstaande proces wordt voltooid door de samenwerking van T4 DNA-polymerase, T4-polynucleotidekinase en Tak DNA-polymerase.
T4 DNA-polymerase (Cat#12901) heeft 5'→3' DNA polymerase activiteit, wat de synthese van DNA langs de 5'→3' richting kan katalyseren en het 5' uitstekende uiteinde kan opvullen. Tegelijkertijd heeft het enzym ook 3'→5' exonuclease activiteit om 3' overhangende uiteinden te splitsen, waardoor DNA fragmenten met kleverige uiteinden worden getransformeerd in stomp-eind DNA.
Omdat de 5'-uiteinden van synthetische PCR-primers en adapters meestal hydroxylgroepen zijn in plaats van fosfaatgroepen, is T4-polynucleotidekinase (Cat#12902) vereist om de overdracht van ATP γ-fosfaatgroepen naar het 5'-hydroxyluiteinde van de oligonucleotideketen te katalyseren in aanwezigheid van ATP, ter voorbereiding op de volgende stap van adapterligatie.
S-Taq DNA-polymerase (Cat#13486) heeft 5'→3' polymerase-activiteit, die DNA kan synthetiseren vanuit de 5'→3' richting. Ondertussen heeft het deoxynucleotidyltransferase-activiteit, die een nucleotide "A" kan toevoegen aan het 3' uiteinde van het PCR-product.

Figuur 3. Meerdere enzymen zijn betrokken bij het uiteindelijke reparatieproces
Figuur 4. S-taq heeft een zeer hoge efficiëntie bij het toevoegen van A aan de vier basen van ATCG van het 3'-uiteinde van de gensegmenten die door capillaire elektroforese worden gedetecteerd.
1.3 Adapterligatie
Adapters vormen een cruciaal onderdeel van de bibliotheek. Binnen het domein van Illumina-sequencing omvatten de algemeen gebruikte Y-type adapters P5/P7-, Index- en Rd1/Rd2 SP-sequenties. Van deze dient de P5/P7-sequentie om te paren met de sequentie die aanwezig is op de sequentiechip, waardoor de te analyseren fragmenten worden verankerd op de flowcel om brugamplificatie uit te voeren. De Index-sequentie wordt gebruikt om onderscheid te maken tussen verschillende monsters binnen de gemengde bibliotheek die aan sequencing wordt onderworpen, terwijl Rd1/Rd2 SP de regio's aanduiden voor het binden van de Read1- en Read2-sequencingprimers.
Voor de taak van adapterligatie, T4 DNA-ligase (Cat#12996) is de standaardkeuze. Het vertoont de mogelijkheid om enkelstrengs nicks te repareren die in dubbelstrengs DNA worden aangetroffen en aangrenzende nucleotiden opnieuw te verbinden.
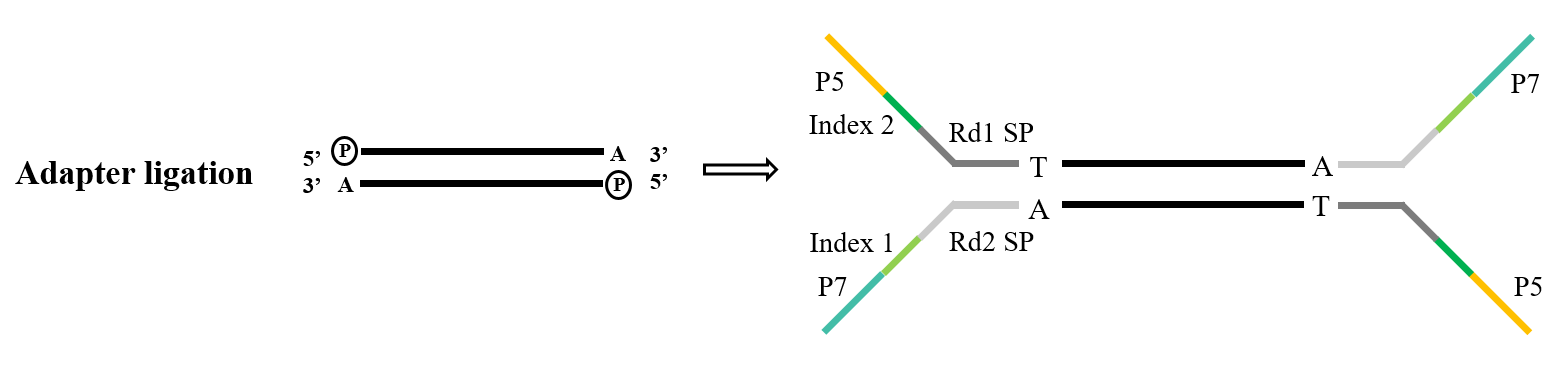
Figuur 5. Algemeen adapterligatieproces (Illumina)

Figuur 6. Verificatie van T4 DNA-ligasemutanten door het ligeren van 170-bp DNA met 80-bp adapters.
1.4 PCR-amplificatie
Verkrijg voldoende DNA-sequenties met adapters via PCR-reactie en voltooi de sequentiebepaling van de monsternucleïnezuursequentie op de machine. Hieff CanaceTM Pro High-Fidelity DNA Polymerase (Cat#13476) dat vaak wordt gebruikt in PCR heeft 5'→3' polymerase-activiteit en kan DNA synthetiseren in de 5'→3' richting. Daarnaast heeft het ook de activiteit van 3'→5' exonuclease, dat de verkeerde incorporatie van basen tijdens het amplificatieproces kan corrigeren, om DNA-fragmenten snel en met hoge betrouwbaarheid te amplificeren.
2. Constructie van RNA-bibliotheek en zijn sleutelenzymen
Afhankelijk van de soorten RNA kan de constructie van een RNA-bibliotheek worden onderverdeeld in een mRNA-bibliotheek, een LncRNA-bibliotheek, enz. Een conventionele RNA-bibliotheek omvat de volgende processen:
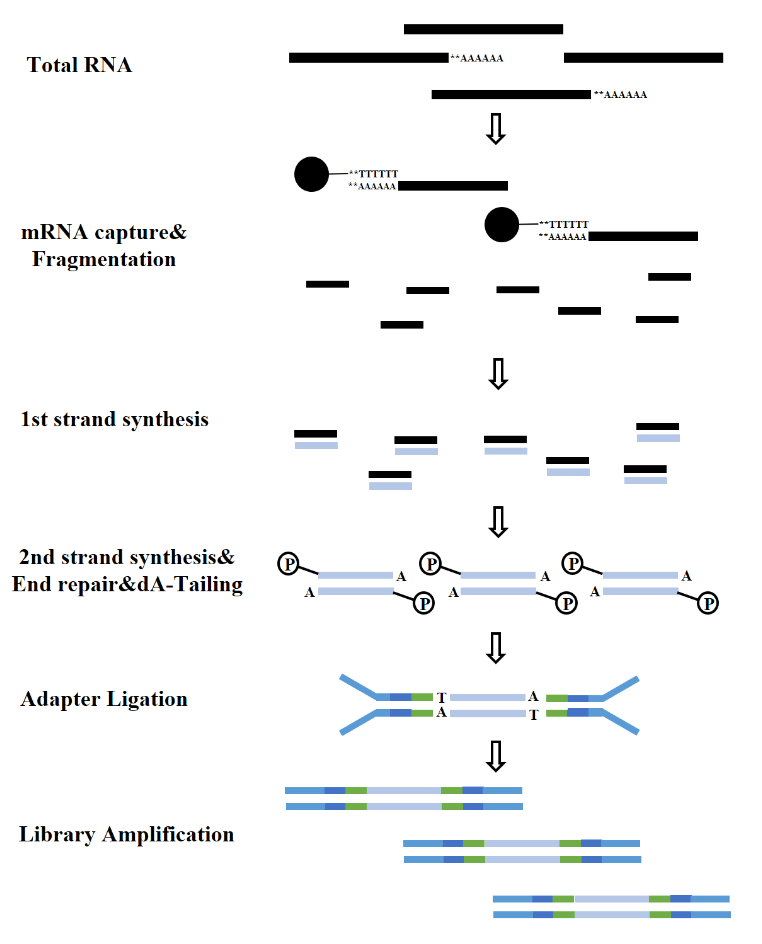
Figuur 7. Proces van mRNA-bibliotheekconstructie (Illumina)
2.1 RNA-verrijking
Of het nu om eukaryoten of prokaryoten gaat, ribosomaal RNA (rRNA) springt eruit als het meest voorkomende RNA, dat tot 80% van de totale RNA-inhoud uitmaakt. Bij het direct sequencen van het totale RNA van een monster, zal een substantieel deel van de sequentiegegevens gerelateerd zijn aan rRNA. Om deze interferentie te verminderen, moet de methode van RNA-verrijking worden gebruikt. Hiervoor zijn twee primaire methoden: mRNA-verrijking op basis van oligo-dT en rRNA-depletiemethoden.
In eukaryoten vertoont mRNA een duidelijke poly(A)-structuur aan het 3'-uiteinde. Oligo-dT-beads kunnen worden gebruikt om alle mRNA dat uit het monster is getranscribeerd, vast te leggen, waardoor het geschikt is voor transcriptionele analyse, met name bij hoogwaardige RNA-monsters. Aan de andere kant hebben rRNA-uitputtingsmethoden soepelere eisen aan de monsterkwaliteit en kunnen ze worden toegepast op zowel monsters van lage kwaliteit (bijv. FFPE-monsters) als RNA-monsters van hoge kwaliteit, evenals prokaryotische monsters. De algemeen gebruikte commerciële aanpak omvat het gebruik van RNase H-digestie om rRNA te verwijderen, waarbij deze specifieke stappen worden gevolgd:
- Synthetiseren van specifieke oligonucleotide-probes die zijn ontworpen om te binden aan rRNA.
- Maak gebruik van RNase H (Cat#12906), dat RNA in de RNA-DNA hybride streng kan afbreken, om selectief het rRNA dat aan de probes gebonden is te verwijderen.
- Verwerk ten slotte de DNA-sondes met DNase I (Cat#10325), die zowel enkel- als dubbelstrengs DNA kan afbreken, en rRNA effectief kan elimineren. Voor meer informatie over DNase I kunt u deze link volgen.
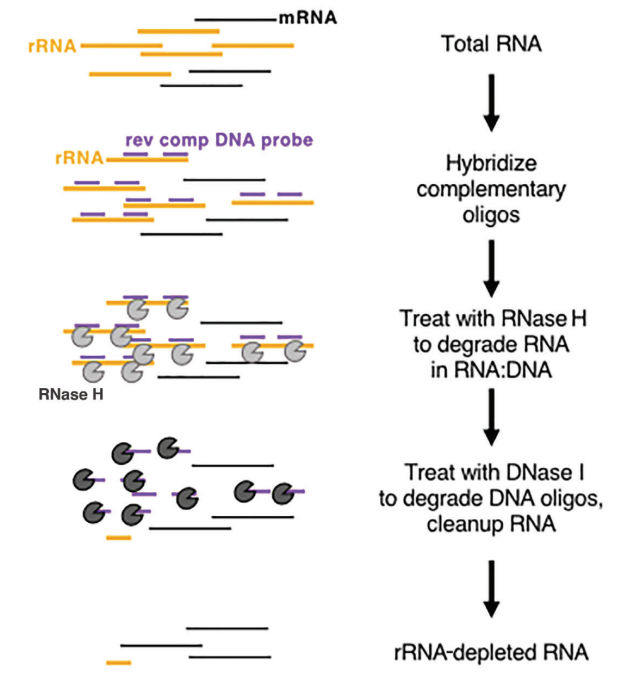
Figuur 8: Schematisch diagram van enzymgebaseerde rRNA-uitputting[5]
2.2 RNA-fragmentatie
Normaal gesproken worden grote RNA-fragmenten onder invloed van tweewaardige metaalkationen en hoge temperaturen in kleine fragmenten gebroken.
2.3 Synthese van 1e streng cDNA
Omgekeerde transcriptie van het verkregen doel-RNA in de eerste streng van cDNA. Omdat RNA gemakkelijk wordt afgebroken door RNases die in de omgeving aanwezig zijn, is het gebruik van RNase-remmer (Cat.nr. 14672) tijdens reverse transcriptie de activiteit van deze enzymen kan remmen en RNA kan beschermen tegen RNase-afbraak. Tegelijkertijd, omgekeerde transcriptase (Cat#11112) werd gebruikt om de template-RNA te reverse-transcriberen naar cDNA. De reverse transcriptase heeft RNA-afhankelijke DNA-polymeraseactiviteit en kan RNA gebruiken als template om een cDNA te synthetiseren in de 5'→3'-richting. De enkelstrengs DNA is complementair aan de RNA-template.
Tijdens de 1e streng cDNA-synthese, de incorporatie van actinomycine D heeft onmiskenbaar de constructie van strengspecifieke bibliotheken verbeterd, wat de specificiteit van de keten aanzienlijk heeft verbeterd. Deze innovatie heeft het experimentele proces gestroomlijnd en vereenvoudigd voor onderzoekers.
Actinomycine D heeft echter ook nadelen: het vertoont toxiciteit en vereist bescherming tegen licht. In het huidige landschap van toenemende vraag naar premixed en plate library constructing kits, vormt de noodzaak om te beschermen tegen licht beperkingen voor de vooruitgang van plate kits.
Gelukkig,

Figuur 9: Engineering van MMLV om MMLV-mutanten te identificeren die kunnen bijdragen aan Standed RNA-seq
2.4 Synthese van 2e streng cDNA
De enkelstrengs cDNA die geproduceerd wordt door reverse transcriptie is zeer onstabiel, wat de onmiddellijke synthese van de tweede streng cDNA noodzakelijk maakt onder invloed van DNA-polymerase I. Tijdens deze tweede strengsynthese komt RNase H in het spel door de RNA-streng te verwijderen uit de RNA-DNA-hybridestructuur. Het werkt samen met DNA-polymerase I (Cat#12903) om de katalytische synthese van de complementaire tweede streng van cDNA te vergemakkelijken. DNA-polymerase I bezit 5'→3' DNA-polymerase-activiteit en synthetiseert, geleid door een template en primer, een sequentie die de enkelstrengs cDNA in de 5'→3'-richting complementeert.
De volgende stappen in het proces omvatten end repair, dA-Tailing, adapter ligation en PCR-amplificatie, die allemaal gedetailleerd worden beschreven in de DNA-bibliotheekconstructieprocedure en hier niet herhaald hoeven te worden. Het is de moeite waard om op te merken dat zodra reverse transcriptie is voltooid, er geen verdere fragmentatie van het nucleïnezuurfragment nodig is.
3. Richtlijn voor NGS-kernenzymen bij de constructie van DNA- en RNA-bibliotheken
Tabel 1.Richtlijn voor NGS-kernenzymen bij de constructie van DNA- en RNA-bibliotheken
| Type | Productpositionering | Productnaam | Kat# |
| RNA-bibliotheek bouw | rRNA uitputting/2e streng cDNA-synthese | 12906ES | |
| rRNA uitputting | 10325ES | ||
| Synthese van 1e streng cDNA | 14672ES | ||
| 11112ES | |||
| Synthese van 2e streng cDNA | 12903ES | ||
| RNA-bibliotheek bouw & DNA bibliotheek bouw | Einde reparatie | 12901ES | |
| 12902ES | |||
| dA-staart | 13486ES | ||
| Adapterligatie | 10301ES | ||
| PCR versterking | 2×Super Canace® II High-Fidelity Mix voor bibliotheekversterking | 12621ES |
Tabel2.DNA & RNA Bibliotheek Voorbereidingskit
| Naam | Kat# | Notities | |
| DNA | Hieff NGS DNA Bibliotheek Voorbereidingskit | 13577ES | Tumor/Mechanische methode |
| Hieff NGS OnePot Pro DNA Bibliotheek Prep Kit V2 | 12194ES | Tumor/ Enzymatische methode | |
| Hoog NGS OnePot II DNA-bibliotheekvoorbereidingskit voor Illumina | 13490ES | Pathgeen/ Enzymatisch/ normale tijd (140min) | |
| Hieff NGS OnePot Flash DNA Bibliotheek Prep Kit | 12316ES | Pathgeen/ Enzymatisch/ Ultrasnel (100 minuten) | |
| Hieff NGS DNA&RNA-bibliotheek Co-Prep Kit V2 | 12305ES | Pathgeen/ Enzymatisch/ DNA & RNA Co-Prep | |
| RNA | Hieff NGS Ultima Dual-mode mRNA Bibliotheek Prep Kit | 12308ES | Zonder oligo dT magnetische kralen, 11 buisjes |
| Hieff NGS Ultima Dual-mode mRNA Bibliotheek Prep Kit | 12309ES | oligo dT magnetische kralen plus, 14 buisjes | |
| Hieff NGS® Ultima Dual-mode RNA Bibliotheekvoorbereidingskit | 12310ES | Voorgemengde versie, 5 tubes | |
| Hieff NGS ® EvoMax RNA Library Prep Kit (voorgemengde versie) (actinomycine D Vrij) | 12340ES | Voorgemixte versie, (Actinomycine D Vrij) | |
| Hieff NGS® MaxUp rRNA-uitputtingskit (plant) | 12254ES | Plant | |
| Hieff NGS® MaxUp Human rRNA Depletion Kit (rRNA & ITS/ETS) | 12257ES | Menselijk |
Referenties:
[1] Mardis, Elaine R. Next-Generation Sequencing Platforms[J]. Jaarlijks overzicht van analytische chemie, 2013, 6(1):287-303.
[2] Gulilat M, Lamb T, Teft WA, et al. Gerichte next generation sequencing als hulpmiddel voor recision medicine[J]. BMC Medical Genomics, 2019, 12(1):81.
[3] Lundberg KS, Dan DS, Adams M, et al. High-fidelity amplificatie met behulp van een thermostabiele DNA-polymerase geïsoleerd uit Pyrococcus furiosus[J]. Gene, 1991, 108(1):1-6.
[4] Miyazaki K. Willekeurige DNA-fragmentatie met endonuclease V: toepassing op DNA-shuffling[J]. Nucleic Acids Research, 2002, 30(24):e139.
[5] Baldwin A, Morris AR, Mukherjee N. Een eenvoudige, kosteneffectieve en schaalbare methode om menselijk ribosomaal RNA uit te putten voor RNA-seq[J]. Current Protocols, 2021, 1(6):e176.

