Высокопроизводительное секвенирование, часто называемое технологией секвенирования следующего поколения (NGS), представляет собой значительный шаг вперед по сравнению с первоначальными методами секвенирования ДНК, такими как секвенирование по Сэнгеру. NGS позволяет одновременно профилировать сотни тысяч, если не миллионы, последовательностей молекул нуклеиновых кислот. К его достоинствам относятся исключительная производительность, экономическая эффективность, масштабируемость и широкий спектр приложений, что делает его преобладающей технологией секвенирования во всем мире.
Рабочий процесс секвенирования NGS включает четыре основных этапа: подготовка образца, создание библиотеки, секвенирование и анализ данных. Центральным моментом в создании библиотеки является присоединение стандартизированных последовательностей адаптеров платформы NGS к обоим концам фрагментированной геномной ДНК. Этот шаг направлен на создание достаточного запаса молекул нуклеиновых кислот библиотеки, подготовленных для секвенирования на инструменте NGS посредством ПЦР-амплификации. В зависимости от природы образца создание библиотеки NGS можно разделить на создание библиотеки ДНК и создание библиотеки РНК. Ферменты играют ключевую роль в этих взаимосвязанных экспериментах. Итак, какие ключевые ферменты участвуют в процессе создания библиотеки?
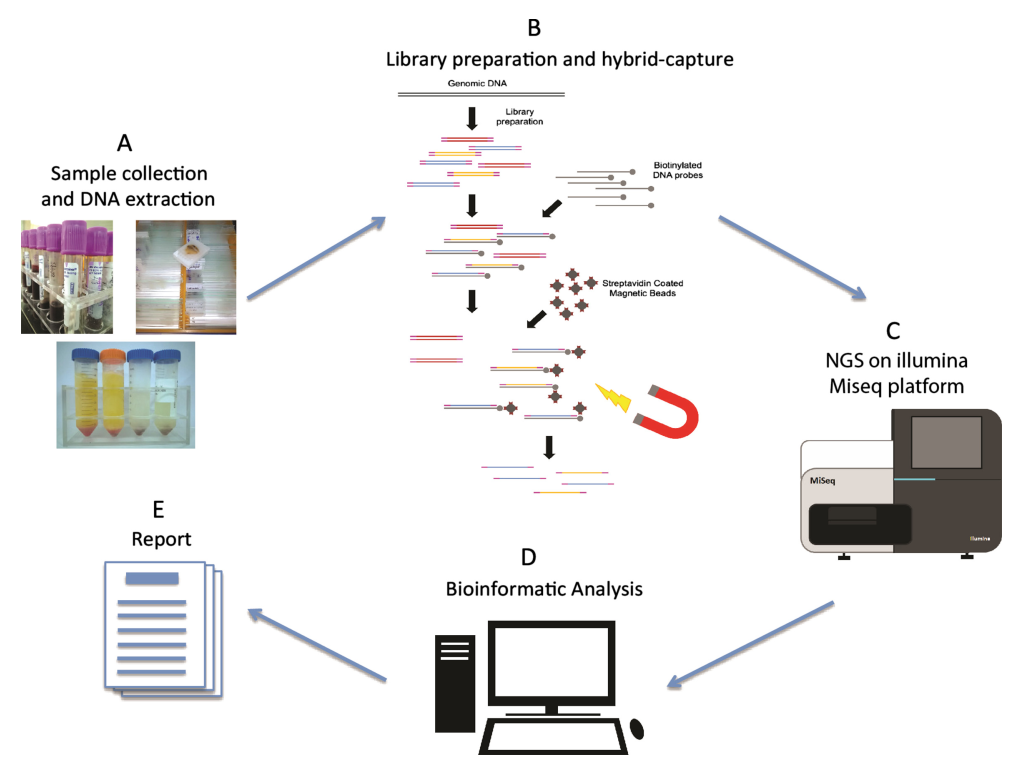
Рисунок 1. Рабочий процесс секвенирования нового поколения[2]
1. Создание библиотеки ДНК и ее ключевые ферменты
2. Создание библиотеки РНК и ее ключевые ферменты
3. Руководство по основным ферментам NGS при создании библиотек ДНК и РНК
1. Создание библиотеки ДНК и ее ключевые ферменты
В процессе построения библиотеки ДНК, построение библиотеки адаптеров лигирования TA-клона является наиболее часто используемым технологическим средством в настоящее время. Основной процесс построения библиотеки выглядит следующим образом:

Рисунок 2. Процесс создания библиотеки ДНК (Illumina)
1.1 Фрагментация ДНК
Современные секвенаторы обычно имеют длину секвенирования в диапазоне 150-500 пар оснований (пн). В результате становится необходимым использовать механические или ферментативные методы фрагментации для разделения больших фрагментов геномной ДНК на более мелкие. Механическая фрагментация может привести к относительно высокой потере образцов и подразумевает более сложный операционный процесс. С другой стороны, ферментативное расщепление является широко используемым методом фрагментации геномной ДНК. По сравнению с механическими методами ферментативное расщепление более экономично и просто, при этом реакция требует только установленного периода после добавления фермента фрагментации.
В настоящее время в основном используются два типа фрагментов. Один основан на транспозазе Tn5, основанной на принципах транспозона, в то время как другой использует смесь эндонуклеаз. Однако эффективность этих фрагментов может зависеть от содержания GC и предпочтений оснований ДНК. Напротив, фрагменты, разработанные Йесеном (Cat#12917), обеспечивают стабильный эффект переваривания и демонстрируют значительно более низкое предпочтение сайта по сравнению с транспозазой Tn5. Они неизменно дают отличные результаты секвенирования для различных типов образцов ДНК, включая образцы из FFPE.
1.2 Ремонт концов, dA-Tailing
Фрагментированная ДНК будет генерировать липкие концы 5'/3' и ДНК с тупыми концами, и все липкие концы необходимо преобразовать в тупые концы, включая удаление 3'-выступов и заполнение 5'-выступающих концов ДНК. При использовании лигирования TA для лигирования адаптера фрагмент ДНК также необходимо фосфорилировать на 5'-конце и добавить «A» на 3'-конце, чтобы он был комплементарным адаптеру с липким концом «T».Вышеуказанный процесс завершается взаимодействием ДНК-полимеразы Т4, полинуклеотидкиназы Т4 и Так ДНК-полимераза.
ДНК-полимераза Т4 (кат. № 12901) имеет 5'→3' ДНК-полимеразную активность, которая может катализировать синтез ДНК вдоль направления 5'→3' и заполнять выступающий 5' конец. В то же время фермент также имеет 3'→5' экзонуклеазную активность для расщепления 3' выступающих концов, тем самым преобразуя фрагменты ДНК, содержащие липкие концы, в ДНК с тупыми концами.
Поскольку 5'-концы синтетических ПЦР-праймеров и адаптеров обычно представляют собой гидроксильные группы вместо фосфатных групп. Поэтому полинуклеотидкиназа T4 (Cat#12902) необходима для катализа переноса АТФ γ-фосфатных групп на 5'-гидроксильный конец олигонуклеотидной цепи в присутствии АТФ в рамках подготовки к следующему этапу лигирования адаптера.
S-Так ДНК-полимераза (Cat#13486) имеет 5'→3' полимеразную активность, которая может синтезировать ДНК в направлении 5'→3'. Между тем, он имеет дезоксинуклеотидилтрансферазную активность, которая может добавлять нуклеотид «A» к 3' концу продукта ПЦР.

Рисунок 3. В процессе конечного восстановления задействовано множество ферментов.
Рисунок 4. S-taq обладает очень высокой эффективностью добавления А к четырем основаниям ATCG 3'-конца сегментов гена, обнаруженных с помощью капиллярного электрофореза.
1.3 Лигирование адаптера
Адаптеры являются важнейшим компонентом библиотеки. В области секвенирования Illumina обычно используемые адаптеры Y-типа охватывают последовательности P5/P7, Index и Rd1/Rd2 SP. Среди них последовательность P5/P7 служит для сопряжения с последовательностью, присутствующей на чипе секвенирования, тем самым закрепляя фрагменты, которые должны быть проанализированы, на проточной ячейке для выполнения мостовой амплификации. Последовательность Index используется для различения различных образцов в смешанной библиотеке, подвергаемой секвенированию, в то время как Rd1/Rd2 SP обозначают области для связывания праймеров секвенирования Read1 и Read2.
Для задачи лигирования адаптера, ДНК-лигаза T4 (кат. № 12996) является стандартным выбором. Он демонстрирует способность восстанавливать одноцепочечные разрывы, обнаруженные в двухцепочечной ДНК, и повторно соединять соседние нуклеотиды.
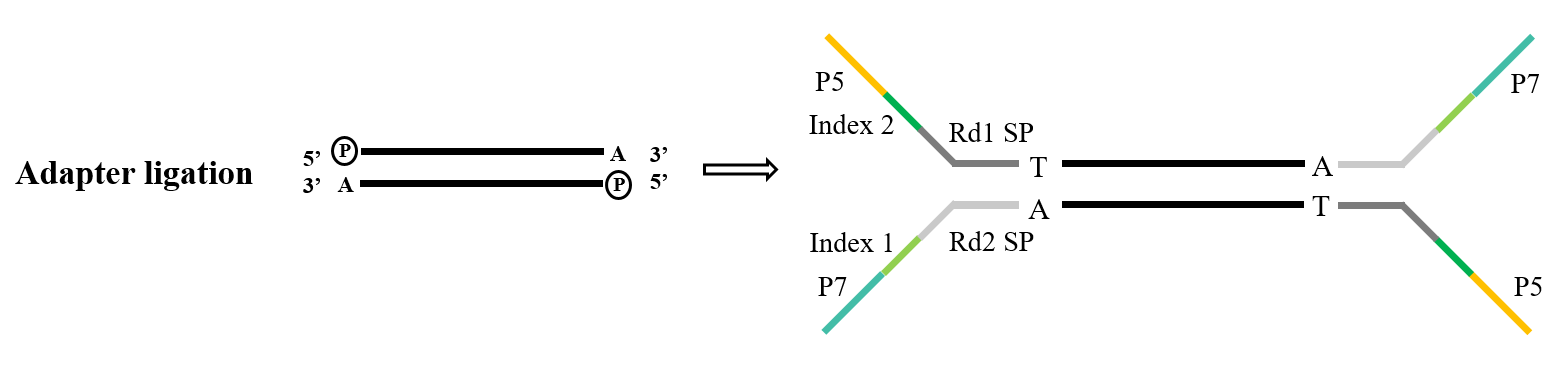
Рисунок 5. Общий процесс лигирования адаптера (Illumina)

Рисунок 6. Проверка мутантов ДНК-лигазы Т4 путем лигирования 170-пн ДНК с 80-пн адаптерами.
1.4 ПЦР-амплификация
Получите достаточное количество последовательностей ДНК с адаптерами с помощью реакции ПЦР и завершите секвенирование последовательности нуклеиновой кислоты образца на машине. Hieff CanaceТМ Pro High-Fidelity DNA Polymerase (Cat#13476), обычно используемая в ПЦР, имеет 5'→3' полимеразную активность и может синтезировать ДНК в направлении 5'→3'. Кроме того, она также имеет активность 3'→5' экзонуклеазы, которая может исправить неправильное включение оснований в процессе амплификации, чтобы амплифицировать фрагменты ДНК быстро и с высокой точностью.
2. Построение библиотеки РНК и ее ключевые ферменты
В зависимости от типов РНК построение библиотеки РНК можно разделить на библиотеку мРНК, библиотеку LncRNA и т. д. Традиционная библиотека РНК включает в себя следующие процессы:
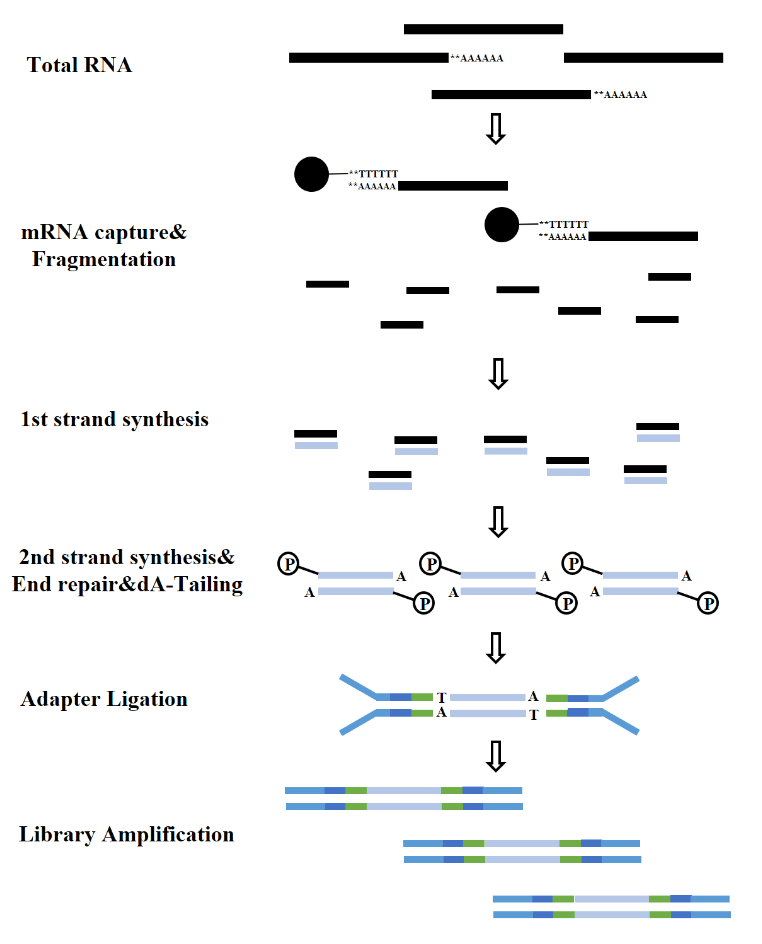
Рисунок 7. Процесс создания библиотеки мРНК (Illumina)
2.1 Обогащение РНК
Независимо от того, имеем ли мы дело с эукариотами или прокариотами, рибосомальная РНК (рРНК) выделяется как наиболее распространенная РНК, составляя до 80% от общего содержания РНК. При прямом секвенировании общей РНК образца значительная часть данных секвенирования будет связана с рРНК. Чтобы смягчить эту интерференцию, необходимо использовать метод обогащения РНК. Для этого существует два основных метода: обогащение мРНК на основе олиго-dT и методы истощения рРНК.
У эукариот мРНК демонстрирует отчетливую структуру поли(А) на 3'-конце. Бусины Oligo-dT можно использовать для захвата всей мРНК, транскрибированной из образца, что делает его пригодным для транскрипционного анализа, особенно с высококачественными образцами РНК. С другой стороны, методы истощения рРНК предъявляют более мягкие требования к качеству образцов и могут применяться как к образцам низкого качества (например, образцам FFPE), так и к высококачественным образцам РНК, а также к прокариотическим образцам. Обычно используемый коммерческий подход включает использование переваривания РНКазой H для удаления рРНК, следуя этим конкретным шагам:
- Синтезировать специфические олигонуклеотидные зонды, предназначенные для связывания с рРНК.
- Используйте РНКазу H (кат. № 12906), которая способна расщеплять РНК в гибридной цепи РНК-ДНК, чтобы выборочно удалить рРНК, связанную с зондами.
- Наконец, расщепите ДНК-зонды с помощью ДНКаза I (кат. № 10325), который может разрушать как одноцепочечную, так и двухцепочечную ДНК, эффективно устраняя рРНК. Для получения дополнительной информации о ДНКазе I вы можете перейти по этой ссылке.
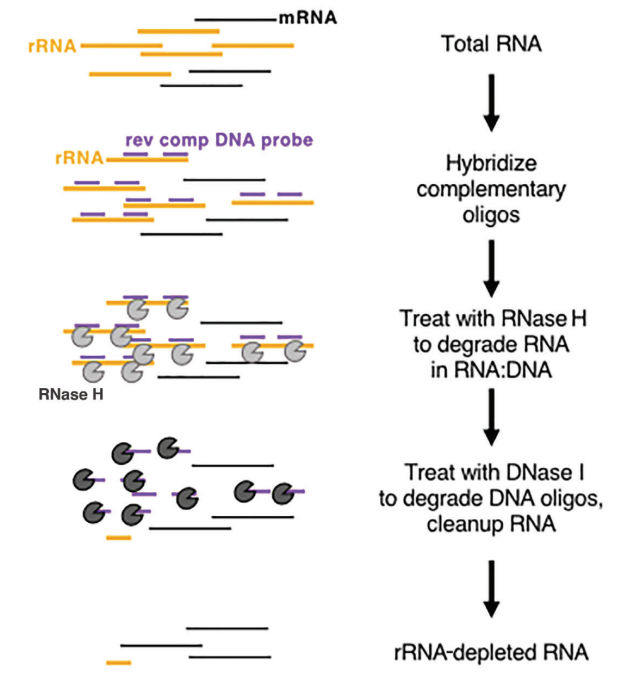
Рисунок 8: Схематическая диаграмма истощения рРНК на основе ферментов[5]
2.2 Фрагментация РНК
Обычно под действием катионов двухвалентных металлов и высокой температуры крупные фрагменты РНК распадаются на мелкие фрагменты.
2.3 Синтез первой цепи ДНК
Обратная транскрипция полученной целевой РНК в первую цепь кДНК. Поскольку РНК легко разрушается РНКазами, присутствующими в окружающей среде, использование Ингибитор РНКазы (Кат. № 14672) во время обратной транскрипции может подавлять активность этих ферментов и защищать РНК от деградации РНКазой. В то же время, обратная транскриптаза (Кат.№11112) была использована для обратной транскрипции шаблонной РНК в кДНК. Обратная транскриптаза имеет РНК-зависимую ДНК-полимеразную активность и может использовать РНК в качестве матрицы для синтеза кДНК в направлении 5'→3'. Одиночная цепь ДНК комплементарна шаблону РНК.
В течение 1-го синтез цепи ДНК, включение актиномицина D, несомненно, улучшило построение библиотек, специфичных для цепи, значительно повысив специфичность цепи. Это нововведение упростило экспериментальный процесс, упростив его для исследователей.
Однако у актиномицина D есть свои недостатки: он токсичен и требует защиты от света. В сегодняшнем ландшафте растущего спроса на предварительно смешанные и пластинчатые наборы для создания библиотек необходимость защиты от света накладывает ограничения на развитие пластинчатых наборов.
К счастью, платформа Yeasen ZymeEditor представила новаторский мутант фермента MMLV (Inquiry), который заменяет функцию актиномицина D. Новый Комплект (кат.: 12340ES) разработан с использованием нетоксичных, не имеющих запаха и нетоксичныхo нужно избегать свет. Он обеспечивает превосходную специфичность цепи, устраняя проблемы, связанные со здоровьем и чувствительностью к свету.

2.4 Синтез второй цепи ДНК
Одноцепочечная ДНК, полученная путем обратной транскрипции, крайне нестабильна, что требует немедленного синтеза второй цепи ДНК под воздействием ДНК-полимеразы I. Во время этого синтеза второй цепи в игру вступает РНКаза H, удаляя цепь РНК из гибридной структуры РНК-ДНК. Она работает совместно с ДНК-полимераза I (Кат.№12903) для облегчения каталитического синтеза комплементарной второй цепи кДНК. ДНК-полимераза I обладает 5'→3' ДНК-полимеразной активностью и, руководствуясь матрицей и праймером, синтезирует последовательность, которая дополняет одноцепочечную кДНК в направлении 5'→3'.
Последующие этапы процесса включают в себя репарацию концов, dA-Tailing, лигирование адаптеров и амплификацию ПЦР, все из которых подробно описаны в процедуре построения библиотеки ДНК и не нуждаются в повторении здесь. Стоит отметить, что после завершения обратной транскрипции нет необходимости в дальнейшей фрагментации фрагмента нуклеиновой кислоты.
3. Руководство по основным ферментам NGS при создании библиотек ДНК и РНК
Yeasen — биотехнологическая компания, занимающаяся исследованиями, разработкой, производством и продажей трех основных биологических реагентов: молекул, белков и клеток. Компания Yeasen Biotech производит различные ферменты, связанные с построением библиотек NGS. Вы можете выбрать наиболее подходящий продукт для построения библиотек из таблицы ниже.
Таблица 1.Руководство по основным ферментам NGS при создании библиотек ДНК и РНК
| Тип | Позиционирование продукта | Название продукта | Кот# |
| Библиотека РНК строительство | рРНК истощение/синтез 2-й цепи ДНК | 12906ES | |
| рРНК истощение | 10325ES | ||
| Синтез первой цепи ДНК | 14672ES | ||
| 11112ES | |||
| Синтез 2-й цепи ДНК | 12903ES | ||
| Библиотека РНК строительство и ДНК библиотека строительство | Завершить ремонт | 12901ES | |
| 12902ES | |||
| dA-хвост | 13486ES | ||
| Адаптерная лигирование | 10301ES | ||
| ПЦР усиление | 2×Super Canace® II High-Fidelity Mix для усиления библиотеки | 12621ES |
Таблица2.Библиотека ДНК и РНК Подготовительный комплект
| Имя | Кот# | Примечания | |
| ДНК | Набор для подготовки библиотеки ДНК Hieff NGS | 13577ES | Опухоль/ Механический метод |
| Комплект для подготовки библиотеки ДНК Hieff NGS OnePot Pro V2 | 12194ES | Опухоль/Ферментативный метод | |
| Hieff NGS OnePot II Набор для подготовки библиотеки ДНК для Illumina | 13490ES | Патогенный/ферментативный/обычное время (140 мин) | |
| Набор для подготовки библиотеки ДНК Hieff NGS OnePot Flash | 12316ES | Патогенный/ Ферментативный/ Сверхбыстрый (100мин) | |
| Набор для совместной подготовки библиотеки ДНК и РНК Hieff NGS V2 | 12305ES | Pathgen/Enzymetic/ДНК и РНК Co-Prep | |
| РНК | Набор для подготовки библиотеки мРНК Hieff NGS Ultima Dual-mode | 12308ES | Без магнитных шариков олиго дТ, 11 пробирок |
| Набор для подготовки библиотеки мРНК Hieff NGS Ultima Dual-mode | 12309ES | олиго дТ магнитные шарики плюс, 14 пробирок | |
| Набор для подготовки библиотеки РНК Hieff NGS® Ultima Dual-mode | 12310ES | Готовая версия, 5 тюбиков | |
| Набор для подготовки библиотеки РНК Hieff NGS ® EvoMax (готовая версия) (актиномицин D) Бесплатно) | 12340ES | Готовая версия, (Актиномицин D Бесплатно) | |
| Набор для истощения рРНК Hieff NGS® MaxUp (растительный) | 12254ES | Растение | |
| Набор для истощения человеческой рРНК Hieff NGS® MaxUp (рРНК и ITS/ETS) | 12257ES | Человек |
Ссылки:
[1] Мардис, Элейн Р. Платформы секвенирования следующего поколения [J]. Ежегодный обзор аналитической химии, 2013, 6(1):287-303.
[2] Гулилат М., Лэмб Т., Тефт ВА и др. Целевое секвенирование следующего поколения как инструмент для прецизионной медицины [J]. BMC Medical Genomics, 2019, 12(1):81.
[3] Лундберг К.С., Дэн Д.С., Адамс М. и др. Высокоточная амплификация с использованием термостабильной ДНК-полимеразы, выделенной из Pyrococcus furiosus[J]. Gene, 1991, 108(1):1-6.
[4] Миядзаки К. Случайная фрагментация ДНК с помощью эндонуклеазы V: применение к перетасовке ДНК [J]. Nucleic Acids Research, 2002, 30(24):e139.
[5] Болдуин А., Моррис А. Р., Мукерджи Н. Простой, экономически эффективный и масштабируемый метод истощения человеческой рибосомальной РНК для РНК-секвенирования [J]. Current Protocols, 2021, 1(6):e176.

