High-throughput-sekvensering, ofta kallad nästa generations sekvenseringsteknik (NGS), representerar ett betydande steg framåt från de ursprungliga DNA-sekvenseringsmetoderna, såsom Sanger-sekvensering. NGS möjliggör samtidig profilering av hundratusentals, om inte miljoner, nukleinsyramolekylsekvenser. Dess fördelar inkluderar exceptionell genomströmning, kostnadseffektivitet, skalbarhet och ett brett spektrum av applikationer, vilket etablerar den som den dominerande sekvenseringstekniken över hela världen.
NGS-sekvenseringsarbetsflödet omfattar fyra primära faser: provberedning, bibliotekskonstruktion, sekvensering och dataanalys. Centralt för bibliotekskonstruktion är fästningen av standardiserade NGS-plattformsadaptersekvenser till båda ändarna av fragmenterat genomiskt DNA. Detta steg syftar till att generera ett rikligt utbud av biblioteksnukleinsyramolekyler, förberedda för sekvensering på NGS-instrumentet genom PCR-amplifiering. Beroende på provets natur kan NGS-bibliotekskonstruktion kategoriseras i DNA-bibliotekskonstruktion och RNA-bibliotekskonstruktion. Enzymer spelar en central roll i dessa sammanlänkade experiment. Så, vilka nyckelenzymer är involverade i processen för bibliotekskonstruktion?
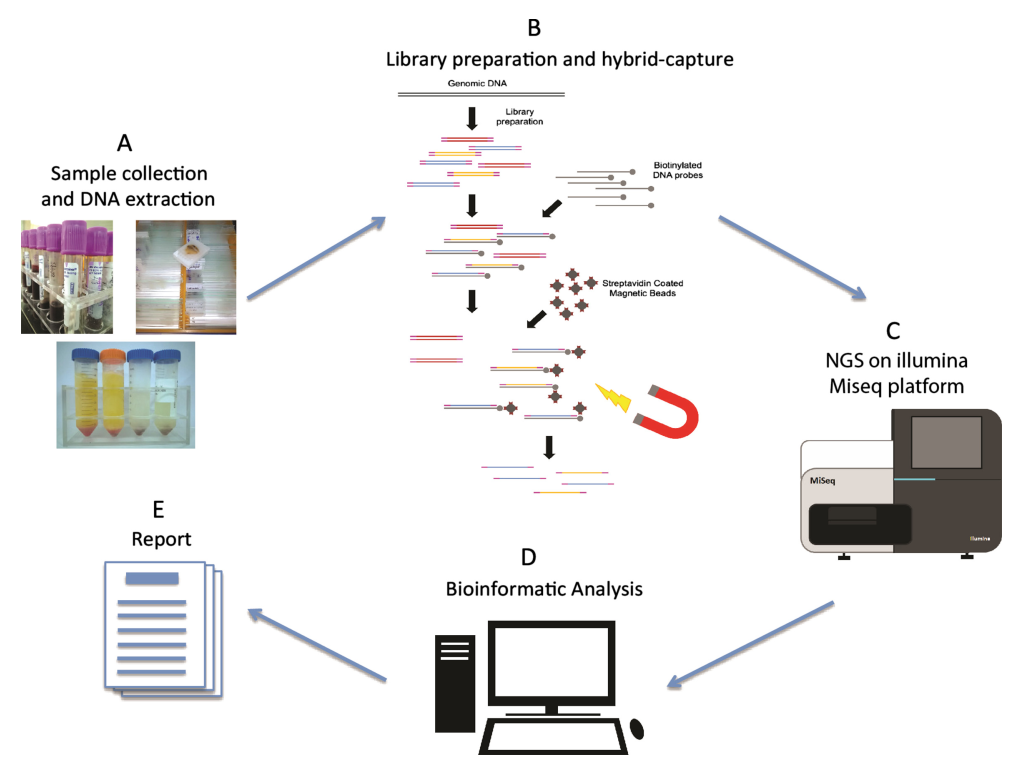
Figur 1. Nästa generations arbetsflöde för sekvensering[2]
1. DNA-bibliotekskonstruktion och dess nyckelenzymer
2. RNA-bibliotekskonstruktion och dess nyckelenzymer
3. Riktlinjer för NGS kärnenzymer i DNA- och RNA-bibliotekskonstruktion
1. DNA-bibliotekskonstruktion och dess nyckelenzymer
I processen för DNA-bibliotekskonstruktion är konstruktionen av TA-klonligeringsadapterbibliotek det mest använda teknikmedlet för närvarande. Den huvudsakliga konstruktionsprocessen för biblioteket är som följer:

Figur 2. Byggprocessen för DNA-biblioteket (Illumina)
1.1 DNA-fragmentering
De nuvarande sekvenserarna har typiskt en sekvenseringslängd i intervallet 150-500 baspar (bp). Som ett resultat blir det nödvändigt att använda mekaniska eller enzymatiska fragmenteringsmetoder för att bryta ner stora genomiska DNA-fragment till mindre. Mekanisk fragmentering kan leda till relativt hög provförlust och innebär en mer komplicerad operativ process. Å andra sidan är enzymatisk nedbrytning en vanlig metod för att fragmentera genomiskt DNA. Jämfört med mekaniska metoder är enzymatisk nedbrytning mer kostnadseffektiv och okomplicerad, där reaktionen endast kräver en bestämd period efter tillsatsen av fragmenteringsenzymet.
För närvarande finns det i första hand två typer av fragment som används. Den ena förlitar sig på Tn5-transposaset, baserat på transposonprinciper, medan den andra använder en blandning av endonukleaser. Effektiviteten hos dessa fragment kan emellertid påverkas av DNA:ts GC-innehåll och baspreferenser. Däremot erbjuder fragmenten utvecklade av Yeasen (Cat #12917) en stabil digestionseffekt och uppvisar signifikant lägre platspreferens jämfört med Tn5-transposas. De ger konsekvent utmärkta sekvenseringsresultat för olika typer av DNA-prover, inklusive de från FFPE-prover.
1.2 Slutreparation, dA-Tailing
Det fragmenterade DNA:t kommer att generera 5'/3'-klibbiga ändar och trubbiga ändar, och alla klibbiga ändar måste omvandlas till trubbiga ändar, inklusive 3'-överhäng borttagna och 5'-utskjutande DNA-ändar ifyllda. När man använder TA-ligering för adapterligering, måste DNA-fragmentet också fosforyleras vid 3'-änden för att addera "A5"-änden och lägga till adaptern i adaptern. den "T" klibbiga änden.Ovanstående process fullbordas genom samarbete mellan T4 DNA-polymeras, T4-polynukleotidkinas och Taq DNA-polymeras.
T4 DNA-polymeras (kat#12901) har 5'→3' DNA-polymerasaktivitet, som kan katalysera syntesen av DNA längs 5'→3'-riktningen och fylla i den 5'-utskjutande änden. Samtidigt har enzymet också 3'→5'-exonukleasaktivitet för att klyva 3'-överhängande ändar, och därigenom transformera DNA-fragment som innehåller klibbiga ändar till trubbiga ändar.
Eftersom 5'-ändarna av syntetiska PCR-primrar och adaptrar vanligtvis är hydroxylgrupper istället för fosfatgrupper. Därför krävs T4-polynukleotidkinas (Cat#12902) för att katalysera överföringen av ATP-y-fosfatgrupper till 5'-hydroxyländen av oligonukleotidkedjan i närvaro av ATP, som förberedelse för nästa steg av adapterligering.
S-Taq DNA-polymeras (Katt#13486) har 5'→3'-polymerasaktivitet, som kan syntetisera DNA från 5'→3'-riktningen. Under tiden har den deoxinukleotidyltransferasaktivitet, som kan lägga till en nukleotid "A" till 3'-änden av PCR-produkten.

Figur 3. Flera enzymer är involverade i slutreparationsprocessen
Figur 4. S-taq har en mycket hög effektivitet att addera A till de fyra baserna av ATCG i 3'-änden av gensegmenten som detekteras genom kapillärelektrofores.
1.3 Adapterligering
Adaptrar utgör en avgörande komponent i biblioteket. Inom området för Illumina-sekvensering omfattar de vanligen använda adaptrarna av Y-typ P5/P7, Index och Rd1/Rd2 SP-sekvenser. Bland dessa tjänar P5/P7-sekvensen till att paras ihop med sekvensen som finns på sekvenseringschippet, och därigenom förankra fragmenten som ska analyseras på flödescellen för att utföra bryggamplifiering. Indexsekvensen används för att skilja mellan olika prover inom det blandade biblioteket som utsätts för sekvensering, medan Rd1/Rd2 SP betecknar regionerna för bindning av Read1- och Read2-sekvenseringsprimrarna.
För uppgiften att adapterligering, T4 DNA-ligas (Katt#12996) är standardvalet. Den uppvisar förmågan att reparera enkelsträngade hack som finns i dubbelsträngat DNA och återkoppla intilliggande nukleotider.
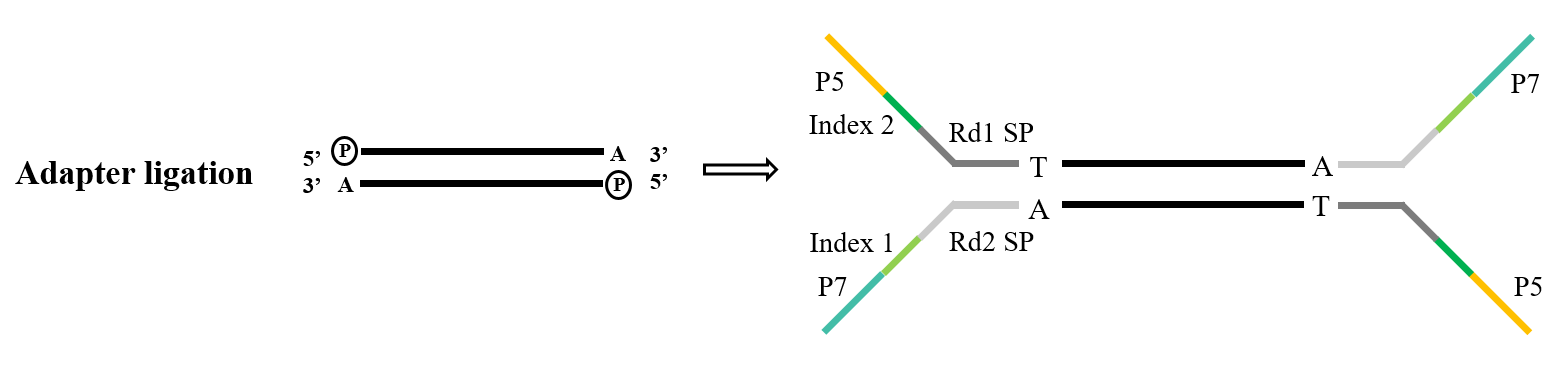
Figur 5. Allmän adapterligeringsprocess (Illumina)

Figur 6. Verifiering av T4 DNA-ligasmutanter genom att ligera 170-bp DNA med 80-bp adaptrar.
1.4 PCR-amplifiering
Skaffa tillräckligt med DNA-sekvenser med adaptrar genom PCR-reaktion och slutför sekvenseringen av provnukleinsyrasekvensen på maskinen. Hieff CanaceTM Pro High-Fidelity DNA-polymeras (Cat#13476) som vanligtvis används i PCR har 5'→3'-polymerasaktivitet och kan syntetisera DNA i 5'→3'-riktningen. Dessutom har den också aktiviteten av 3'→5'-exonukleas, vilket kan korrigera felaktig inkorporering av baser under amplifieringsprocessen, för att amplifiera DNA-fragment snabbt och med hög tillförlitlighet.
2. RNA-bibliotekskonstruktion och dess nyckelenzymer
Beroende på typerna av RNA kan konstruktionen av ett RNA-bibliotek delas in i mRNA-bibliotek, LncRNA-bibliotek, etc. Konventionellt RNA-bibliotek inkluderar följande processer:
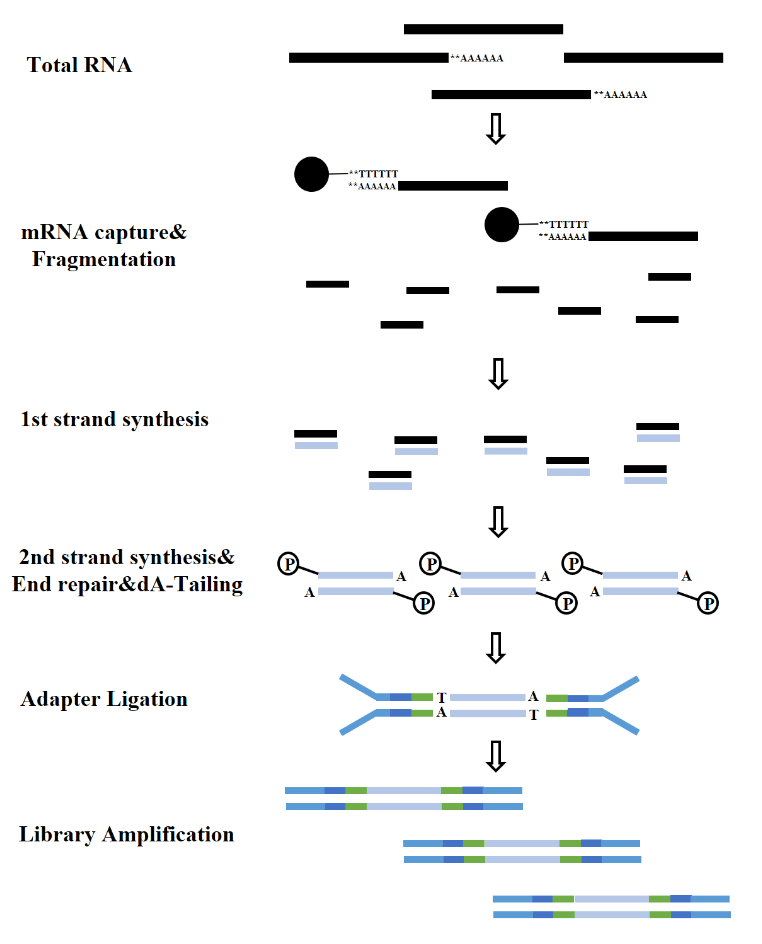
Figur 7. mRNA-bibliotekskonstruktionsprocess (Illumina)
2.1 RNA-anrikning
Oavsett om det handlar om eukaryoter eller prokaryoter, sticker ribosomalt RNA (rRNA) ut som det vanligaste RNA:t, som utgör upp till 80 % av det totala RNA-innehållet. Vid sekvensering av det totala RNA:t i ett prov direkt, kommer en betydande del av sekvenseringsdata att relateras till rRNA. För att mildra denna interferens måste metoden för RNA-berikning användas. Det finns två primära metoder för detta: mRNA-anrikning baserad på oligo-dT- och rRNA-utarmningsmetoder.
I eukaryoter uppvisar mRNA en distinkt poly(A)-struktur vid 3'-änden. Oligo-dT-pärlor kan användas för att fånga upp allt mRNA som transkriberats från provet, vilket gör det lämpligt för transkriptionsanalys, särskilt med RNA-prover av hög kvalitet. Å andra sidan har rRNA-utarmningsmetoder mildare krav på provkvalitet och kan tillämpas på både lågkvalitativa prover (t.ex. FFPE-prover) och högkvalitativa RNA-prover, såväl som prokaryota prover. Det vanliga kommersiella tillvägagångssättet involverar användningen av RNase H-digestion för att avlägsna rRNA, genom att följa dessa specifika steg:
- Syntetisera specifika oligonukleotidsonder utformade för att binda till rRNA.
- Använd RNase H (Cat#12906), som kan bryta ned RNA i RNA-DNA-hybridsträngen, för att selektivt avlägsna rRNA bundet till proberna.
- Slutligen smälta DNA-sonderna med DNas I (Katt#10325), som kan bryta ned både enkel- och dubbelsträngat DNA, vilket effektivt eliminerar rRNA. För mer information om DNase I kan du följa denna länk.
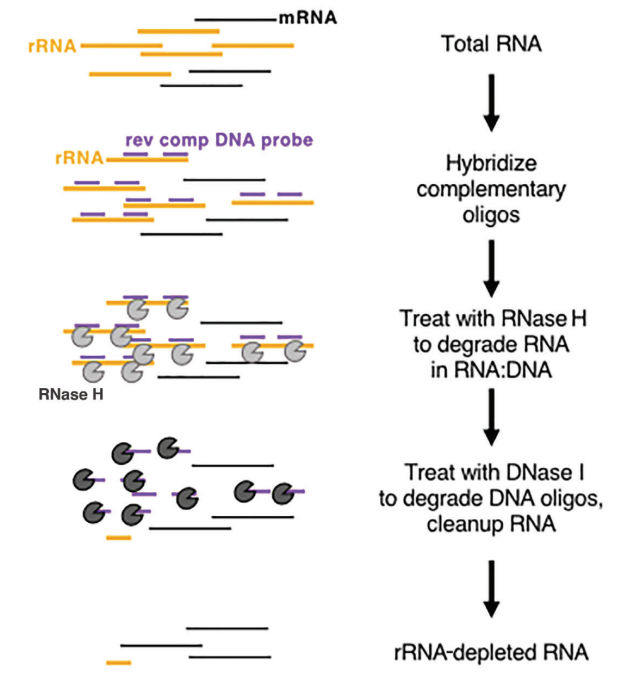
Figur 8: Schematiskt diagram av enzymbaserad rRNA-utarmning[5]
2.2 RNA-fragmentering
Vanligtvis, under inverkan av tvåvärda metallkatjoner och hög temperatur, bryts stora fragment av RNA i små fragment.
2.3 1:a strängens cDNA-syntes
Omvänd transkription av det erhållna mål-RNA:t till den första strängen av cDNA. Eftersom RNA lätt bryts ned av RNaser som finns i miljön kan användningen av RNas-hämmare (Katt#14672) under omvänd transkription kan hämma aktiviteten av dessa enzymer och skydda RNA från RNas-nedbrytning. Samtidigt, omvänt transkriptas (Katt#11112) användes för att omvända transkribera mall-RNA:t till cDNA. Det omvända transkriptaset har RNA-beroende DNA-polymerasaktivitet och kan använda RNA som mall för att syntetisera ett cDNA i 5'→3'-riktningen. Den enkla DNA-strängen är komplementär till RNA-mallen.
Under den 1:a sträng cDNA-syntes, har införlivandet av aktinomycin D onekligen förbättrat konstruktionen av strängspecifika bibliotek, vilket avsevärt förbättrat kedjespecificiteten. Denna innovation har effektiviserat den experimentella processen och förenklat den för forskare.
Emellertid har aktinomycin D sina nackdelar: det uppvisar toxicitet och kräver skydd mot ljus. I dagens landskap med ökande efterfrågan på färdigblandade och plåtbibliotekskonstruktionssatser, innebär nödvändigheten att skydda mot ljus begränsningar för plåtsatsens framsteg.
Lyckligtvis har Yeasen ZymeEditor-plattformen introducerat en banbrytande MMLV-enzymmutant (Inquiry) som ersätter funktionen av actinomycin D. En ny Kit (Cat: 12340ES) har utvecklats med luktfri, giftfri och no behöver undvika ljus. Den erbjuder överlägsen kedjespecificitet, vilket eliminerar farhågor relaterade till hälsa och ljuskänslighet.

Bild 9: Engineering av MMLV för att identifiera MMLV-mutanter som skulle kunna bidra till Standed RNA-seq
2.4 2:a strängens cDNA-syntes
Det enkelsträngade cDNA som produceras genom omvänd transkription är mycket instabilt, vilket kräver omedelbar syntes av den andra strängen av cDNA under påverkan av DNA-polymeras I. Under denna andra strängsyntes kommer RNas H in i bilden genom att ta bort RNA-strängen från RNA-DNA-hybridstrukturen. Det fungerar tillsammans med DNA-polymeras I (kat#12903) för att underlätta den katalytiska syntesen av den komplementära andra strängen av cDNA. DNA-polymeras I besitter 5'→3'-DNA-polymerasaktivitet och, styrt av en mall och primer, syntetiserar en sekvens som kompletterar det enkelsträngade cDNA:t i 5'→3'-riktningen.
De efterföljande stegen i processen inkluderar slutreparation, dA-Tailing, adapterligering och PCR-amplifiering, som alla är detaljerade i DNA-bibliotekskonstruktionsproceduren och behöver inte upprepas här. Det är värt att notera att när omvänd transkription väl är klar finns det inget behov av ytterligare fragmentering av nukleinsyrafragmentet.
3. Riktlinjer för NGS kärnenzymer i DNA- och RNA-bibliotekskonstruktion
Yeasen är ett bioteknikföretag som är engagerat i forskning, utveckling, produktion och försäljning av tre viktiga biologiska reagenser: molekyler, proteiner och celler. Yeasen Biotech-företag producerar en mängd olika enzymer relaterade till NGS-bibliotekskonstruktion. Du kan välja den mest lämpliga biblioteksbyggprodukten från tabellen nedan.
Tabell 1.Riktlinje för NGS kärnenzymer i DNA- och RNA-bibliotekskonstruktion
| Typ | Produktpositionering | Produktnamn | Katt# |
| RNA-bibliotek konstruktion | rRNA utarmning/syntes av cDNA för andra strängen | 12906ES | |
| rRNA tömning | 10325ES | ||
| 1:a strängens cDNA-syntes | 14672ES | ||
| 11112ES | |||
| 2:a strängens cDNA-syntes | 12903ES | ||
| RNA-bibliotek konstruktion & DNA bibliotek konstruktion | Avsluta reparationen | 12901ES | |
| 12902ES | |||
| dA-Tailing | 13486ES | ||
| Adapterligering | 10301ES | ||
| PCR förstärkning | 2×Super Canace® II High-Fidelity-mix för biblioteksförstärkning | 12621ES |
Tabell 2.DNA & RNA bibliotek Prep Kit
| Namn | Katt# | Anteckningar | |
| DNA | Hieff NGS DNA Library Prep Kit | 13577ES | Tumör/mekanisk metod |
| Hieff NGS OnePot Pro DNA Library Prep Kit V2 | 12194ES | Tumör/enzymetisk metod | |
| Hieff NGS OnePot II DNA Library Prep Kit för Illumina | 13490ES | Patogen/enzymetisk/vanlig tid (140min) | |
| Hieff NGS OnePot Flash DNA Library Prep Kit | 12316ES | Patogen/Enzymetisk/ Ultrasnabb (100 min) | |
| Hieff NGS DNA&RNA Library Co-Prep Kit V2 | 12305ES | Pathgen/ Enzymetic/ DNA & RNA Co-Prep | |
| RNA | Hieff NGS Ultima Dual-mode mRNA Library Prep Kit | 12308ES | Utan oligo dT magnetiska pärlor, 11 rör |
| Hieff NGS Ultima Dual-mode mRNA Library Prep Kit | 12309ES | oligo dT magnetiska pärlor plus, 14 rör | |
| Hieff NGS® Ultima Dual-mode RNA Library Prep Kit | 12310ES | Färdigblandad version, 5 rör | |
| Hieff NGS ® EvoMax RNA Library Prep Kit (Förblandad version) (aktinomycin D Gratis) | 12340ES | Förblandad version, (Actinomycin D Gratis) | |
| Hieff NGS® MaxUp rRNA-utarmningskit (växt) | 12254ES | Plantera | |
| Hieff NGS® MaxUp Human rRNA Depletion Kit (rRNA & ITS/ETS) | 12257ES | Mänsklig |
Referenser:
[1] Mardis, Elaine R. Nästa generations sekvenseringsplattformar[J]. Annual Review of Analytical Chemistry, 2013, 6(1):287-303.
[2] Gulilat M, Lamb T, Teft WA, et al. Riktad nästa generations sekvensering som ett verktyg för recisionsmedicin[J]. BMC Medical Genomics, 2019, 12(1):81.
[3] Lundberg KS, Dan DS, Adams M, et al. High-fidelity amplifiering med användning av ett termostabilt DNA-polymeras isolerat från Pyrococcus furiosus[J]. Gene, 1991, 108(1):1-6.
[4] Miyazaki K. Slumpmässig DNA-fragmentering med endonukleas V: tillämpning på DNA-shuffling[J]. Nucleic Acids Research, 2002, 30(24):e139.
[5] Baldwin A, Morris AR, Mukherjee N. En enkel, kostnadseffektiv och skalbar metod för att utarma mänskligt ribosomalt RNA för RNA-seq[J]. Current Protocols, 2021, 1(6):e176.

