Học sâu các nguyên liệu thô của enzyme cốt lõi được sử dụng trong Phát hiện PCR của SARS-CoV-2
Từ tháng 3 năm 2022, chủng đột biến Omicron xảo quyệt một lần nữa phá vỡ cuộc sống yên bình của người dân, dịch bệnh do virus corona mới bùng phát trên khắp cả nước và ảnh hưởng đến 30 tỉnh (khu tự trị và thành phố). Là một biện pháp hiệu quả để phòng ngừa và kiểm soát chính xác dịch bệnh SARS-CoV-2, phát hiện axit nucleic đã trở thành một cách sống bình thường. "Hôm nay bạn đã làm xét nghiệm axit nucleic chưa?" Nó cũng đã trở thành lời chào hàng ngày của mọi người. Nói đến đó, bạn có biết những nguyên liệu thô cốt lõi nào là cần thiết trong quá trình phát hiện axit nucleic không? Bài viết này sẽ giới thiệu về nguyên liệu thô cốt lõi quan trọng trong enzyme phát hiện axit nucleic.
1. Quy trình phát hiện axit nucleic cho SARS-CoV-2
2. Enzym cốt lõi trong chiết xuất axit nucleic
3. Các enzyme cốt lõi trong RT-qPCR
4. Các Enzym cốt lõi của Phát hiện Axit Nucleic SARS-CoV-2 từ
1. Quy trình phát hiện axit nucleic đối với SARS-CoV-2
Enzym là một lớp chất xúc tác sinh học cực kỳ quan trọng với hiệu suất xúc tác và tính đặc hiệu phản ứng cao. Hầu hết các phản ứng sinh hóa đều cần sự tham gia của enzim. Trong quá trình phát hiện axit nucleic của 2019-nCoV (thể hiện trong Hình 1), các loại enzim phân tử khác nhau đóng vai trò quan trọng trong các giai đoạn thử nghiệm khác nhau như chiết xuất axit nucleic và RT-qPCR. Tiếp theo, theo các liên kết thử nghiệm khác nhau trong quá trình phát hiện axit nucleic, chúng tôi sẽ phân loại các nguyên liệu thô của enzim cốt lõi được sử dụng trong quá trình phát hiện axit nucleic.
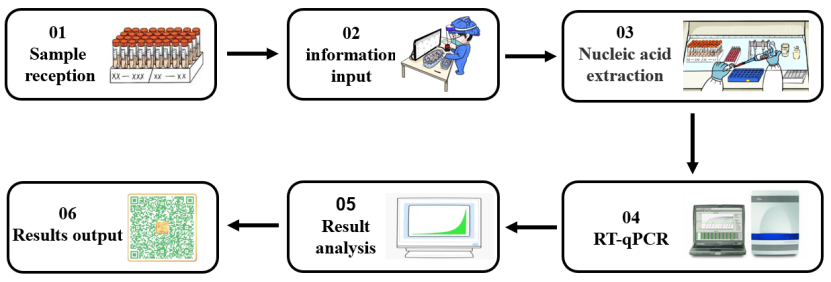
Hình 1. Quy trình phát hiện axit nucleic đối với SARS-CoV-2
2. Các enzyme cốt lõi trong chiết xuất axit nucleic
Quá trình chiết xuất axit nucleic của virus corona mới chủ yếu bao gồm hai bước: ly giải và tinh chế. Ly giải là quá trình phá hủy cấu trúc tế bào của mẫu để axit nucleic trong mẫu được tự do trong hệ thống ly giải; Tinh chế là quá trình tách hoàn toàn axit nucleic khỏi các thành phần khác trong hệ thống ly giải, chẳng hạn như protein, muối và các tạp chất khác, và quá trình phản ứng đòi hỏi sự tham gia của proteinase K, deoxyribonuclease I và chất ức chế RNase.
2.1 Protease K
Proteinase K là một protease serine có hoạt tính cắt rộng, các vị trí cắt là các liên kết peptide đầu carboxyl của các axit amin aliphatic và thơm (Hình 2). Trong quá trình chiết xuất axit nucleic, proteinase K có thể phân hủy các histon liên kết chặt chẽ với axit nucleic, thúc đẩy quá trình tách axit nucleic và giúp axit nucleic mẫu dễ chiết xuất hơn. Ngoài ra, proteinase K có thể phân hủy hoạt động của RNA hydrolase (RNase) và ức chế quá trình thủy phân RNA khuôn mẫu của RNase.
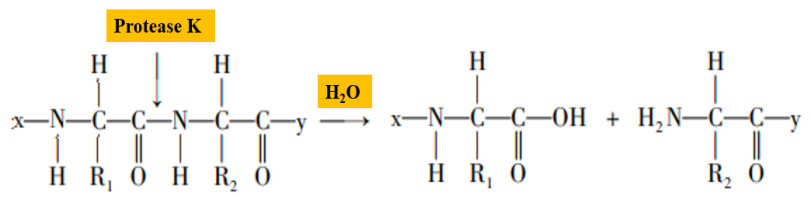
Hình 2. Sơ đồ của proteinase K thủy phân liên kết peptide
2.2 Deoxyribonuclease I
Deoxyribonuclease I (DNase I) có thể xúc tác nhiều dạng DNA khác nhau, nhắm tới mục tiêu cắt các liên kết phosphodiester liền kề với pyrimidine và tạo ra polynucleotide có nhóm phosphate ở đầu 5' và nhóm hydroxyl ở đầu 3', sản phẩm tiêu hóa trung bình là polytetranucleotide nhỏ nhất.Trong quá trình chiết xuất axit nucleic SARS-CoV-2, DNase I chủ yếu được sử dụng để loại bỏ tạp chất bộ gen trong mẫu RNA, tránh dư lượng DNA trong khuôn mẫu RNA và cải thiện độ tinh khiết của khuôn mẫu.
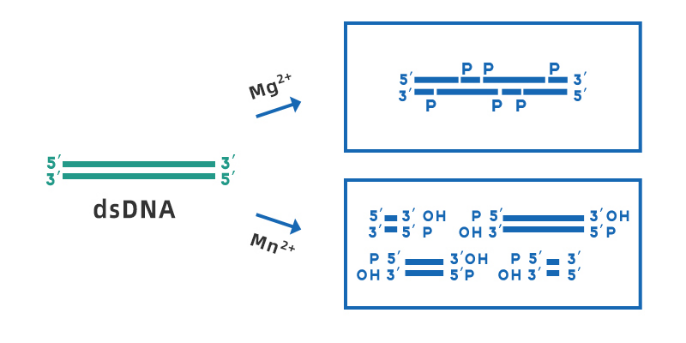
Hình 3. Sơ đồ phân cắt dsDNA của DNase I khi có mặt Mg2+ và Mn2+
2.3 Chất ức chế RNase
Trong quá trình phát hiện axit nucleic SARS-CoV-2, việc chiết xuất và tinh chế axit nucleic mẫu hoặc chuẩn bị hệ thống phản ứng thử nghiệm có thể dẫn đến nhiễm bẩn ribonuclease (RNase), dẫn đến sự phân hủy khuôn mẫu RNA. Để tránh nhiễm bẩn RNase, cần có Chất ức chế RNase.
Chất ức chế RNase là chất ức chế RNase đặc hiệu trong nhau thai người, có khả năng liên kết đặc hiệu với RNase để tạo thành phức hợp có liên kết không cộng hóa trị và vô hiệu hóa RNase.
3. Các enzyme cốt lõi trong RT-qPCR
Sau khi hoàn tất quá trình chiết xuất axit nucleic của mẫu SARS-CoV-2, việc phát hiện axit nucleic có thể được hoàn tất bằng RT-qPCR. Trong quá trình thực hiện các thí nghiệm này, DNA polymerase, reverse transcriptase và uracil DNA glycosylase đều là nguyên liệu cốt lõi thiết yếu của enzyme.
3.1 Phiên mã ngược
Sau khi chiết xuất và tinh chế, RNA SARS-CoV-2 cần phiên mã ngược để xúc tác quá trình trùng hợp dNTP để tạo ra trình tự cDNA bổ sung cho RNA khuôn mẫu (Hình 4). Đối với phản ứng RT-qPCR, nên chọn phiên mã ngược chịu nhiệt độ cao. Hiện nay, phiên mã ngược MMLV được sử dụng rộng rãi nhất, do không có hoạt tính endonuclease DNA và hoạt tính RNase H thấp, nên có nhiều ưu điểm hơn trong ứng dụng nhân bản cDNA.
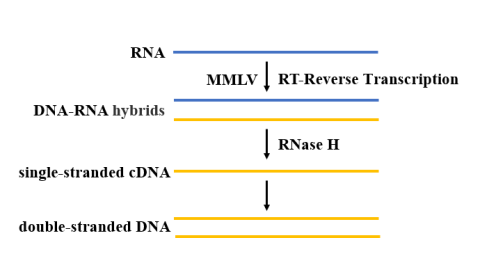
Hình 4. Sơ đồ quá trình phiên mã ngược
3.2 ADN polymerase
Sau khi quá trình phiên mã ngược khuôn mẫu hoàn tất để tạo ra cDNA mạch kép, DNA polymerase "linh hồn" trong phản ứng PCR cần phải xuất hiện, bằng cách trùng hợp các deoxyribonucleotide tự do để kéo dài chuỗi DNA và một lượng lớn DNA khuôn mẫu được khuếch đại trong ống nghiệm để đạt được mục đích phát hiện axit nucleic của vi-rút.
DNA polymerase thường được sử dụng trong phản ứng RT-qPCR là hot-start Taq DNA polymerase. Loại enzyme này không hoạt động ở nhiệt độ phòng.Nó chỉ có hoạt động trùng hợp sau khi khởi động nóng, có thể giảm thiểu việc tạo ra tín hiệu nền. Nó giải quyết các vấn đề khuếch đại không đặc hiệu do tạo ra primer-dimer hoặc không khớp trong các phản ứng PCR thông thường. Hiện nay, các phương pháp sửa đổi khởi động nóng DNA polymerase thường được sử dụng chủ yếu bao gồm sửa đổi hóa học, sửa đổi phối tử và sửa đổi kháng thể. Các nguyên tắc của các phương pháp sửa đổi khởi động nóng khác nhau được thể hiện trong Hình 5.
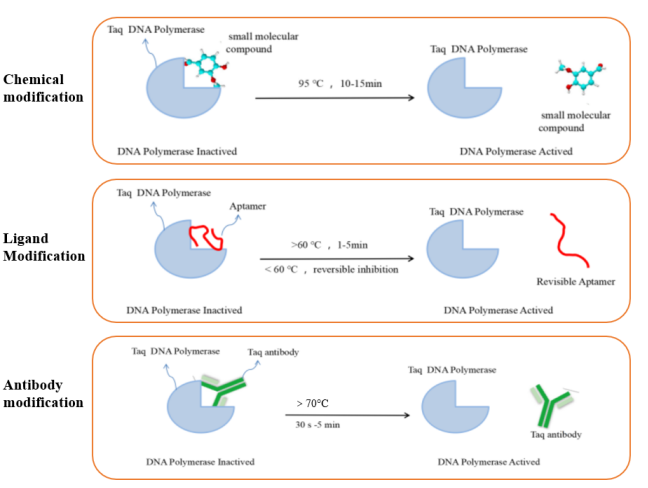
Hình 5. Sơ đồ các loại enzyme khởi động nóng được biến đổi khác nhau
3.3 Uracil ADN glycosylase
Trong quá trình phát hiện axit nucleic của virus corona mới, ô nhiễm khí dung trong môi trường hoạt động là yếu tố phổ biến nhất gây ra kết quả PCR dương tính giả. Việc thêm enzyme UDG (Uracil DNA Glycosylase, uracil DNA glycosylase) vào hệ thống khuếch đại có thể loại bỏ hiệu quả các chất ô nhiễm còn sót lại của quá trình khuếch đại (chủ yếu ở dạng khí dung) trộn lẫn trong hệ thống PCR để đảm bảo độ chính xác của kết quả khuếch đại. Nguyên lý chống ô nhiễm của enzyme UDG được thể hiện trong Hình 6.
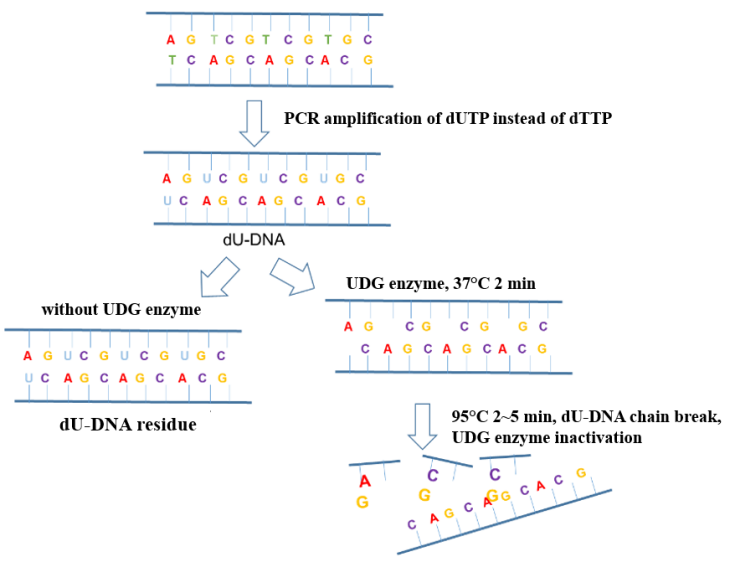
Hình 6. Sơ đồ nguyên lý chống ô nhiễm của enzyme UDG
4.Các Enzym cốt lõi của Phát hiện Axit Nucleic SARS-CoV-2 từ Yeasen
| Quá trình | Sự miêu tả | Tên sản phẩm | Mã sản phẩm |
| Xử lý mẫu | Tiêu hóa protein | 10401ES | |
| Chiết xuất RNA | tái tổ hợp DNase I (RNase-free) (Yêu cầu) | 10325ES | |
| ức chế RNase | Chất ức chế RNase ở chuột (40U/μL) | 10603ES | |
| Phiên mã ngược | Phù hợp với RT-qPCR | Hifair™ V Phiên mã ngược (200U/μL) | 11300ES |
| HifairTM V Reverse Transcriptase (600U/μL) không chứa GLyceroL (Yêu cầu) | 11301ES | ||
| khuếch đại PCR | DNA polymerase khởi động nóng | 10726ES | |
| Nhiệt UDG | Uracil DNA Glycosylase (UDG/UNG), không bền với nhiệt, 1 U/μL | 10303ES |
Về việc đọc:
Lựa chọn phiên mã ngược
Chất ức chế RNase ở chuột - Loại bỏ thành công sự nhiễm bẩn RNase và bảo quản RNA
