High-throughput-sekventering, ofte omtalt som næste generations sekventeringsteknologi (NGS), repræsenterer et betydeligt spring fremad fra de oprindelige DNA-sekventeringsmetoder, såsom Sanger-sekventering. NGS giver mulighed for samtidig profilering af hundredtusinder, hvis ikke millioner, af nukleinsyremolekylesekvenser. Dens fordele inkluderer enestående gennemstrømning, omkostningseffektivitet, skalerbarhed og et bredt spektrum af applikationer, hvilket etablerer det som den fremherskende sekventeringsteknologi på verdensplan.
NGS-sekventeringsworkflowet omfatter fire primære faser: prøveforberedelse, bibliotekskonstruktion, sekventering og dataanalyse. Centralt for bibliotekskonstruktion er vedhæftningen af standardiserede NGS-platformadaptersekvenser til begge ender af fragmenteret genomisk DNA. Dette trin sigter mod at generere en rigelig forsyning af biblioteksnukleinsyremolekyler, forberedt til sekventering på NGS-instrumentet gennem PCR-amplifikation. Afhængigt af arten af prøven kan NGS-bibliotekskonstruktion kategoriseres i DNA-bibliotekskonstruktion og RNA-bibliotekskonstruktion. Enzymer spiller en central rolle i disse indbyrdes forbundne eksperimenter. Så hvilke nøgleenzymer er involveret i processen med biblioteksopbygning?
Figur 1. Næste generations arbejdsgang for sekventering[2]
1. DNA-bibliotekskonstruktion og dets nøgleenzymer
2. RNA-bibliotekskonstruktion og dets nøgleenzymer
3. Retningslinje for NGS-kerneenzymer i DNA- og RNA-bibliotekskonstruktion
1. DNA-bibliotekskonstruktion og dets nøgleenzymer
I processen med DNA-bibliotekskonstruktion er TA-klonligeringsadapterbibliotekskonstruktion det mest almindeligt anvendte teknologiske middel på nuværende tidspunkt. Hovedbibliotekets byggeproces er som følger:

Figur 2. DNA-biblioteks konstruktionsproces (Illumina)
1.1 DNA-fragmentering
De nuværende sequencere har typisk en sekventeringslængde i området 150-500 basepar (bp). Som et resultat bliver det nødvendigt at anvende mekaniske eller enzymatiske fragmenteringsmetoder til at nedbryde store genomiske DNA-fragmenter til mindre. Mekanisk fragmentering kan føre til relativt højt prøvetab og involverer en mere indviklet operationel proces. På den anden side er enzymatisk fordøjelse en almindeligt anvendt metode til fragmentering af genomisk DNA. Sammenlignet med mekaniske metoder er enzymatisk fordøjelse mere omkostningseffektiv og ligetil, idet reaktionen kun kræver en bestemt periode efter tilsætning af fragmenteringsenzymet.
I øjeblikket er der primært to typer fragmenter i brug. Den ene er afhængig af Tn5-transposasen, baseret på transposonprincipper, mens den anden anvender en blanding af endonukleaser. Effektiviteten af disse fragmenter kan imidlertid påvirkes af DNA'ets GC-indhold og basepræferencer. I modsætning hertil tilbyder fragmenterne udviklet af Yeasen (Cat#12917) en stabil fordøjelseseffekt og udviser signifikant lavere stedpræference sammenlignet med Tn5-transposase. De giver konsekvent fremragende sekventeringsresultater for forskellige typer DNA-prøver, herunder dem fra FFPE-prøver.
1.2 Slutreparation, dA-Tailing
Det fragmenterede DNA vil generere 5'/3' klæbrige ender og stumpende DNA, og alle klæbrige ender skal omdannes til stumpe ender, inklusive 3'-udhæng fjernet og 5'-udskudte DNA-ender udfyldt. Når du bruger TA-ligering til adapterligering, skal DNA-fragmentet også phosphoryleres i 3'-enden for at blive 3'-enden og tilføje adapteren til 3'-enden. den "T" klæbrige ende.Ovenstående proces fuldendes ved samarbejde mellem T4 DNA-polymerase, T4-polynukleotidkinase og Taq DNA polymerase.
T4 DNA-polymerase (kat #12901) har 5'→3' DNA-polymeraseaktivitet, som kan katalysere syntesen af DNA langs 5'→3'-retningen og udfylde den 5'-fremspringende ende. Samtidig har enzymet også 3'→5' exonukleaseaktivitet til at spalte 3'-overhængende ender, hvorved DNA-fragmenter, der indeholder klæbrige ender, transformeres til stumpende DNA.
Da 5'-enderne af syntetiske PCR-primere og adaptere sædvanligvis er hydroxylgrupper i stedet for phosphatgrupper. Derfor kræves T4-polynukleotidkinase (Cat#12902) for at katalysere overførslen af ATP-y-phosphatgrupper til 5'-hydroxyl-enden af oligonukleotidkæden i nærvær af ATP, som forberedelse til det næste trin af adapterligering.
S-Taq DNA polymerase (Kat #13486) har 5'→3' polymeraseaktivitet, som kan syntetisere DNA fra 5'→3' retning. I mellemtiden har den deoxynukleotidyltransferaseaktivitet, som kan tilføje et nukleotid "A" til 3'-enden af PCR-produktet.

Figur 3. Flere enzymer er involveret i slutreparationsprocessen
Figur 4. S-taq har en meget høj effektivitet til at tilføje A til de fire baser af ATCG i 3'-enden af gensegmenterne påvist ved kapillærelektroforese.
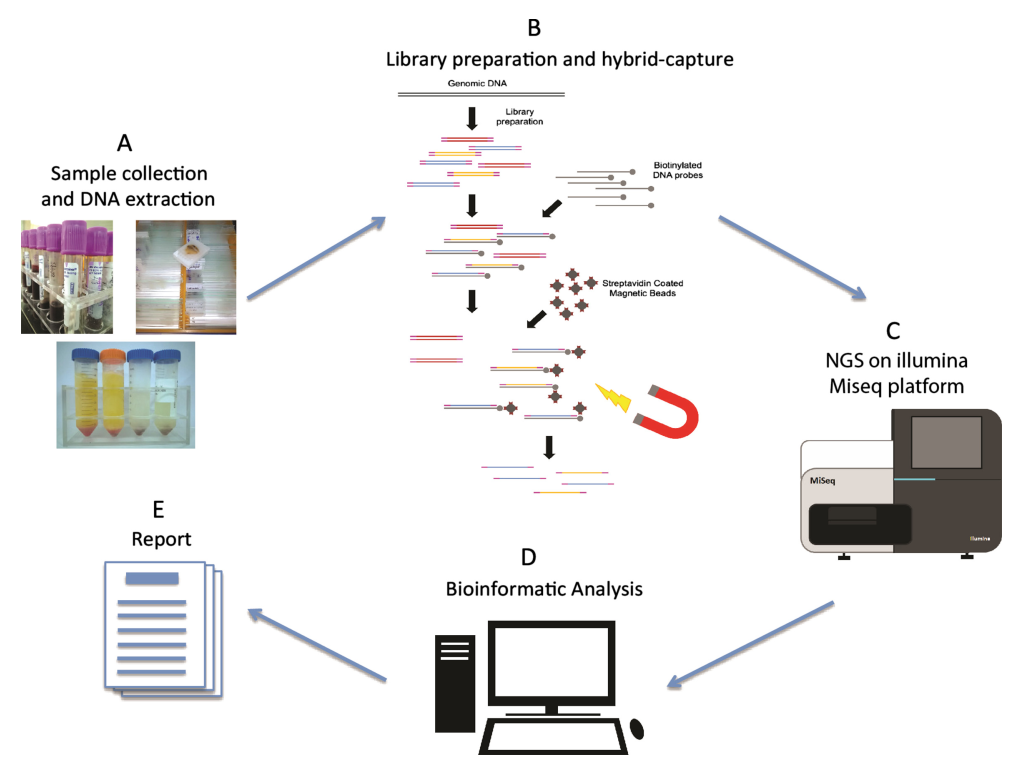
1.3 Adapterligering
Adaptere udgør en afgørende komponent i biblioteket. Inden for Illumina-sekventering omfatter de almindeligt anvendte Y-type adaptere P5/P7, Index og Rd1/Rd2 SP-sekvenser. Blandt disse tjener P5/P7-sekvensen det formål at parre med sekvensen til stede på sekventeringschippen, hvorved de fragmenter, der skal analyseres, forankres på flowcellen for at udføre broamplifikation. Indekssekvensen bruges til at skelne mellem forskellige prøver i det blandede bibliotek, der er udsat for sekventering, mens Rd1/Rd2 SP angiver regionerne til binding af Read1- og Read2-sekventeringsprimerne.
Til opgaven med adapter ligering, T4 DNA-ligase (kat#12996) er standardvalget. Det udviser evnen til at reparere enkeltstrengede hak fundet i dobbeltstrenget DNA og genforbinde tilstødende nukleotider.
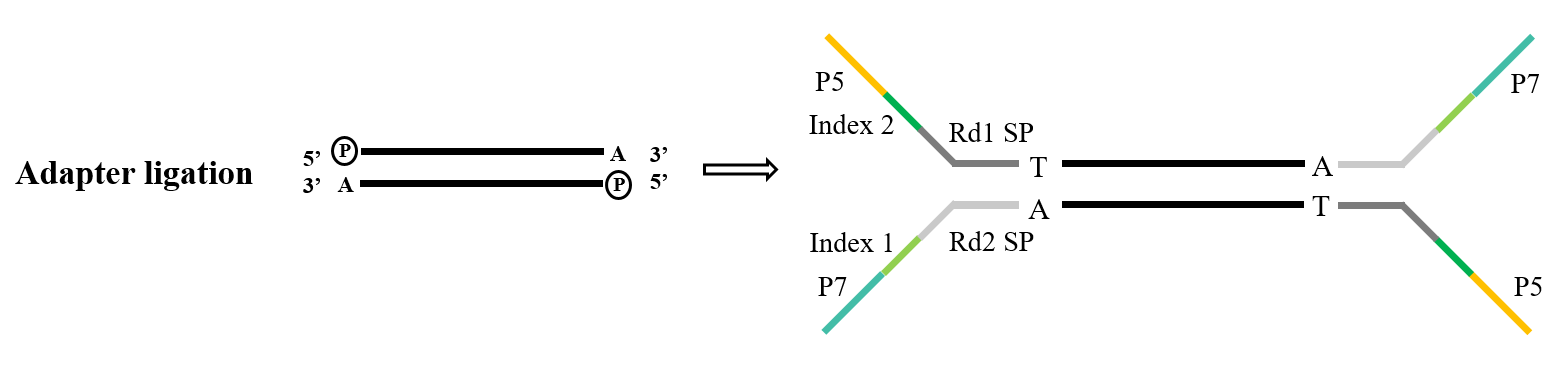
Figur 5. Generel adapterligeringsproces (Illumina)

Figur 6. Verifikation af T4 DNA-ligasemutanter ved ligering af 170-bp-DNA med 80-bp-adaptere.
1.4 PCR-amplifikation
Få nok DNA-sekvenser med adaptere gennem PCR-reaktion, og fuldfør sekventeringen af prøvenukleinsyresekvensen på maskinen. Hieff CanaceTM Pro High-Fidelity DNA-polymerase (kat#13476), der almindeligvis anvendes i PCR, har 5'→3'-polymeraseaktivitet og kan syntetisere DNA i 5'→3'-retningen. Derudover har det også aktiviteten af 3'→5' exonuklease, som kan korrigere den forkerte inkorporering af baser under amplifikationsprocessen, for at amplificere DNA-fragmenter hurtigt og med høj troværdighed.
2. RNA-bibliotekskonstruktion og dets nøgleenzymer
Ifølge RNA-typerne kan konstruktionen af et RNA-bibliotek opdeles i mRNA-bibliotek, LncRNA-bibliotek osv. Konventionelt RNA-bibliotek omfatter følgende processer:
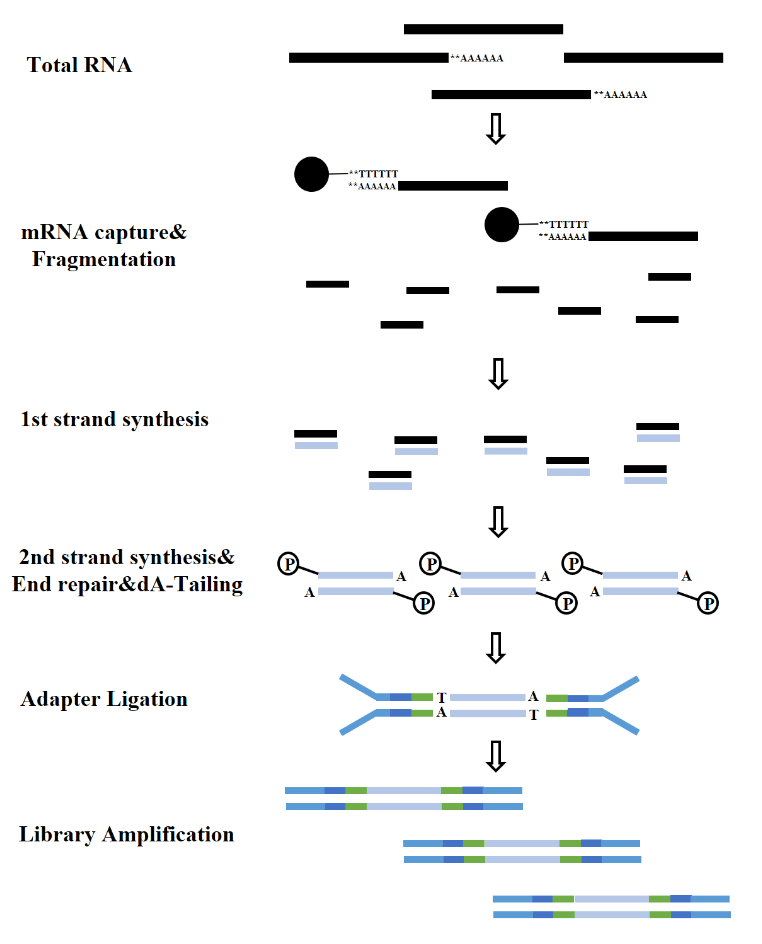
Figur 7. mRNA-biblioteks konstruktionsproces (Illumina)
2.1 RNA berigelse
Uanset om det drejer sig om eukaryoter eller prokaryoter, skiller ribosomalt RNA (rRNA) sig ud som det mest udbredte RNA, der udgør op til 80% af det samlede RNA-indhold. Når man sekventerer det totale RNA i en prøve direkte, vil en væsentlig del af sekventeringsdataene være relateret til rRNA. For at afbøde denne interferens skal metoden til RNA-berigelse anvendes. Der er to primære metoder til dette: mRNA-berigelse baseret på oligo-dT- og rRNA-udtømningsmetoder.
I eukaryoter udviser mRNA en distinkt poly(A) struktur i 3'-enden. Oligo-dT-perler kan anvendes til at fange alt mRNA transskriberet fra prøven, hvilket gør det velegnet til transkriptionel analyse, især med RNA-prøver af høj kvalitet. På den anden side har rRNA-udtømningsmetoder mere lempelige krav til prøvekvalitet og kan anvendes på både lavkvalitetsprøver (f.eks. FFPE-prøver) og RNA-prøver af høj kvalitet såvel som prokaryote prøver. Den almindeligt anvendte kommercielle tilgang involverer brugen af RNase H-fordøjelse til at fjerne rRNA ved at følge disse specifikke trin:
- Syntetisere specifikke oligonukleotidprober designet til at binde til rRNA.
- Anvend RNase H (Cat#12906), som er i stand til at nedbryde RNA i RNA-DNA-hybridstrengen, til selektivt at fjerne rRNA'et bundet til proberne.
- Til sidst fordøj DNA-proberne med DNase I (kat#10325), som kan nedbryde både enkelt- og dobbeltstrenget DNA, hvilket effektivt eliminerer rRNA. For mere information om DNase I, kan du følge dette link.
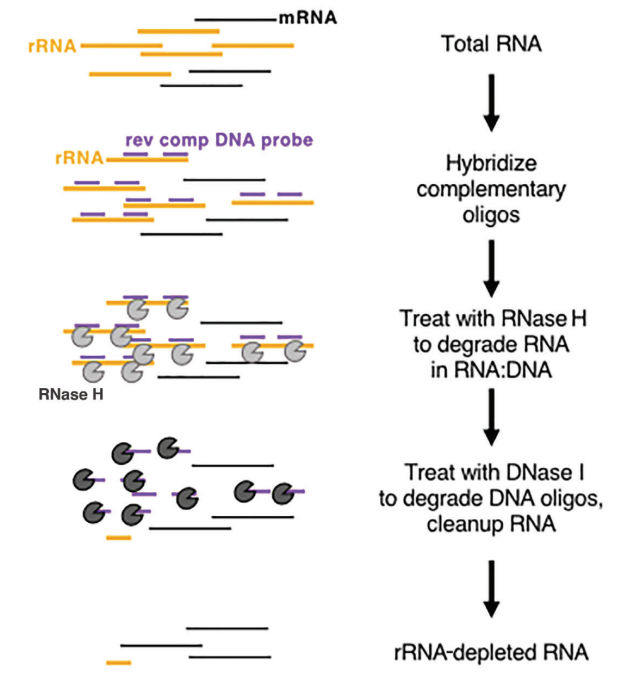
Figur 8: Skematisk diagram af enzymbaseret rRNA-udtømning[5]
2.2 RNA-fragmentering
Normalt, under påvirkning af divalente metalkationer og høj temperatur, bliver store fragmenter af RNA brudt i små fragmenter.
2.3 1. streng cDNA syntese
Omvendt transkription af det opnåede mål-RNA til den første streng af cDNA. Fordi RNA let nedbrydes af RNaser til stede i miljøet, er brugen af RNase-hæmmer (kat#14672) under omvendt transkription kan hæmme aktiviteten af disse enzymer og beskytte RNA mod RNase-nedbrydning. På samme tid, omvendt transkriptase (kat#11112) blev brugt til at reversere transskribering af skabelon-RNA'et til cDNA. Den omvendte transkriptase har RNA-afhængig DNA-polymeraseaktivitet og kan bruge RNA som skabelon til at syntetisere et cDNA i 5'→3'-retningen. Den enkelte DNA-streng er komplementær til RNA-skabelonen.
I løbet af den 1 streng cDNA syntese, har inkorporeringen af actinomycin D unægtelig forbedret konstruktionen af strengspecifikke biblioteker, hvilket væsentligt forbedrer kædespecificiteten. Denne innovation har strømlinet den eksperimentelle proces og forenklet den for forskere.
Actinomycin D har dog sine ulemper: det udviser toksicitet og kræver beskyttelse mod lys. I nutidens landskab med stigende efterspørgsel efter færdigblandede og pladebibliotekskonstruktionssæt, udgør nødvendigheden af at afskærme mod lys begrænsninger for pladesættets fremskridt.
Heldigvis har Yeasen ZymeEditor-platformen introduceret en banebrydende MMLV-enzymmutant (Inquiry), der erstatter funktionen af actinomycin D. En ny Kit (kat: 12340ES) er udviklet med lugtfri, ikke-giftig og no nødt til at undgå lys. Det tilbyder overlegen kædespecificitet, hvilket eliminerer bekymringer relateret til sundhed og lysfølsomhed.

Figur 9: Engineering af MMLV for at identificere MMLV-mutanter, som kunne bidrage til Standed RNA-seq
2.4 2. streng cDNA syntese
Det enkeltstrengede cDNA, der produceres gennem revers transkription, er meget ustabilt, hvilket nødvendiggør den umiddelbare syntese af den anden streng af cDNA under påvirkning af DNA-polymerase I. Under denne anden strengsyntese kommer RNase H i spil ved at fjerne RNA-strengen fra RNA-DNA-hybridstrukturen. Det fungerer sammen med DNA-polymerase I (kat #12903) for at lette den katalytiske syntese af den komplementære anden streng af cDNA. DNA-polymerase I besidder 5'→3'-DNA-polymeraseaktivitet og, styret af en skabelon og primer, syntetiserer en sekvens, der komplementerer enkeltstrenget cDNA i 5'→3'-retningen.
De efterfølgende trin i processen inkluderer endereparation, dA-Tailing, adapterligering og PCR-amplifikation, som alle er detaljeret beskrevet i DNA-bibliotekskonstruktionsproceduren og behøver ikke at blive gentaget her. Det er værd at bemærke, at når revers transkription er afsluttet, er der ikke behov for yderligere fragmentering af nukleinsyrefragmentet.
3. Retningslinje for NGS-kerneenzymer i DNA- og RNA-bibliotekskonstruktion
Yeasen er en bioteknologisk virksomhed, der beskæftiger sig med forskning, udvikling, produktion og salg af tre store biologiske reagenser: molekyler, proteiner og celler. Yeasen Biotech-virksomhed producerer en række enzymer relateret til NGS-bibliotekskonstruktion. Du kan vælge det bedst egnede biblioteksbyggeprodukt fra skemaet nedenfor.
Tabel 1.Vejledning for NGS-kerneenzymer i DNA- og RNA-bibliotekskonstruktion
| Type | Produktpositionering | Produktnavn | Kat# |
| RNA bibliotek konstruktion | rRNA udtømning/2. streng cDNA syntese | 12906ES | |
| rRNA udtømning | 10325ES | ||
| 1. streng cDNA syntese | 14672ES | ||
| 11112ES | |||
| 2. streng cDNA syntese | 12903ES | ||
| RNA bibliotek konstruktion & DNA bibliotek konstruktion | Afslut reparation | 12901ES | |
| 12902ES | |||
| dA-Tailing | 13486ES | ||
| Adapter ligering | 10301ES | ||
| PCR forstærkning | 2×Super Canace® II High-Fidelity-mix til biblioteksforstærkning | 12621ES |
Tabel 2.DNA & RNA bibliotek Forberedelseskit
| Navn | Kat# | Noter | |
| DNA | Hieff NGS DNA Library Prep Kit | 13577ES | Tumor/mekanisk metode |
| Hieff NGS OnePot Pro DNA Library Prep Kit V2 | 12194ES | Tumor/enzymetisk metode | |
| Hieff NGS OnePot II DNA Library Prep Kit til Illumina | 13490ES | Patogen/enzymetisk/normal tid (140 min) | |
| Hieff NGS OnePot Flash DNA Library Prep Kit | 12316ES | Patogen/enzymetisk/ultrahurtig (100 min) | |
| Hieff NGS DNA&RNA Library Co-Prep Kit V2 | 12305ES | Pathgen/ Enzymetic/ DNA & RNA Co-Prep | |
| RNA | Hieff NGS Ultima Dual-mode mRNA Library Prep Kit | 12308ES | Uden oligo dT magnetiske perler, 11 rør |
| Hieff NGS Ultima Dual-mode mRNA Library Prep Kit | 12309ES | oligo dT magnetiske perler plus, 14 rør | |
| Hieff NGS® Ultima Dual-mode RNA Library Prep Kit | 12310ES | Forblandet version, 5 rør | |
| Hieff NGS ® EvoMax RNA Library Prep Kit (forblandet version) (actinomycin D Gratis) | 12340ES | Forblandet version, (Actinomycin D Gratis) | |
| Hieff NGS® MaxUp rRNA-udtømningssæt (plante) | 12254ES | Plante | |
| Hieff NGS® MaxUp Human rRNA Depletion Kit (rRNA & ITS/ETS) | 12257ES | Human |
Referencer:
[1] Mardis, Elaine R. Næste generations sekvenseringsplatforme[J]. Annual Review of Analytical Chemistry, 2013, 6(1):287-303.
[2] Gulilat M, Lamb T, Teft WA, et al. Målrettet næste generations sekvensering som et værktøj til recision medicin[J]. BMC Medical Genomics, 2019, 12(1):81.
[3] Lundberg KS, Dan DS, Adams M, et al. High-fidelity amplifikation ved hjælp af en termostabil DNA-polymerase isoleret fra Pyrococcus furiosus[J]. Gene, 1991, 108(1):1-6.
[4] Miyazaki K. Tilfældig DNA-fragmentering med endonuklease V: anvendelse til DNA-shuffling[J]. Nucleic Acids Research, 2002, 30(24):e139.
[5] Baldwin A, Morris AR, Mukherjee N. En nem, omkostningseffektiv og skalerbar metode til at udtømme humant ribosomalt RNA for RNA-seq[J]. Current Protocols, 2021, 1(6):e176.

