Deep Learning der wichtigsten Enzymrohstoffe in der PCR-Nachweis von SARS-CoV-2
Seit März 2022 hat der heimtückische Omikron-Mutantenstamm erneut das friedliche Leben der Menschen gestört. Die neuartigen Coronavirus-Epidemien brachen im ganzen Land aus und betrafen 30 Provinzen (autonome Regionen und Gemeinden). Als wirksames Mittel zur präzisen Prävention und Kontrolle der SARS-CoV-2-Epidemie ist die Nukleinsäureerkennung zu einer normalen Lebensweise geworden. „Haben Sie heute den Nukleinsäuretest gemacht?“ ist auch zu einer täglichen Begrüßung der Menschen geworden. Apropos, wissen Sie, welche Kernrohstoffe für die Nukleinsäureerkennung benötigt werden? In diesem Artikel werden die kritischen Kernrohstoffe für das Nukleinsäureerkennungsenzym vorgestellt.
1. Nukleinsäure-Nachweisverfahren für SARS-CoV-2
2. Kernenzyme bei der Nukleinsäureextraktion
3. Kernenzyme während der RT-qPCR
4. Kernenzyme der SARS-CoV-2-Nukleinsäureerkennung von
1. Nukleinsäure-Nachweisverfahren für SARS-CoV-2
Ein Enzym ist eine äußerst wichtige Klasse von Biokatalysatoren mit hoher katalytischer Effizienz und Reaktionsspezifität. Die meisten biochemischen Reaktionen erfordern die Beteiligung von Enzymen. Im Prozess der Nukleinsäureerkennung von 2019-nCoV (siehe Abbildung 1) spielen verschiedene Arten molekularer Enzyme in verschiedenen experimentellen Phasen wie der Nukleinsäureextraktion und RT-qPCR eine wichtige Rolle. Als Nächstes werden wir gemäß den verschiedenen experimentellen Verbindungen bei der Nukleinsäureerkennung die wichtigsten Enzymrohstoffe sortieren, die im Nukleinsäureerkennungsprozess verwendet werden.
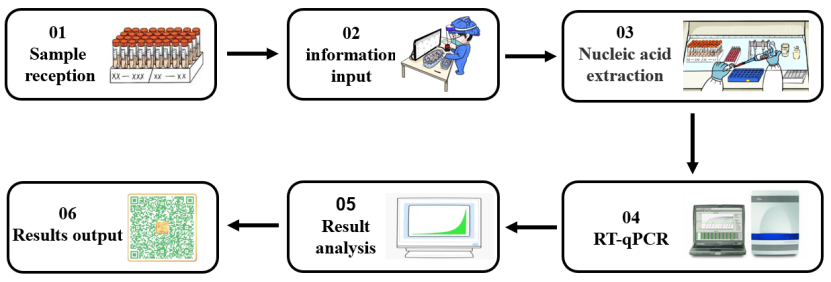
Abbildung 1. Nukleinsäure-Nachweisverfahren für SARS-CoV-2
2. Kernenzyme bei der Nukleinsäureextraktion
Der Extraktionsprozess der Nukleinsäure des neuen Coronavirus umfasst hauptsächlich zwei Schritte: Lyse und Reinigung. Bei der Lyse wird die Zellstruktur der Probe zerstört, sodass die Nukleinsäure in der Probe im Lysesystem frei ist. Bei der Reinigung wird die Nukleinsäure vollständig von anderen Komponenten im Lysesystem getrennt, wie z. B. Protein, Salz und anderen Verunreinigungen. Der Reaktionsprozess erfordert die Beteiligung von Proteinase K, Desoxyribonuklease I und RNase-Inhibitoren.
2.1 Protease K
Proteinase K ist eine Serinprotease mit breiter Spaltungsaktivität. Die Spaltungsstellen sind die carboxyterminalen Peptidbindungen von aliphatischen und aromatischen Aminosäuren (Abbildung 2). Im Prozess der Nukleinsäureextraktion kann Proteinase K Histone abbauen, die eng mit Nukleinsäuren verbunden sind, die Trennung von Nukleinsäuren fördern und die Extraktion von Nukleinsäureproben erleichtern. Darüber hinaus kann Proteinase K die Aktivität der RNA-Hydrolase (RNase) abbauen und die RNase-Hydrolyse von Matrizen-RNA hemmen.
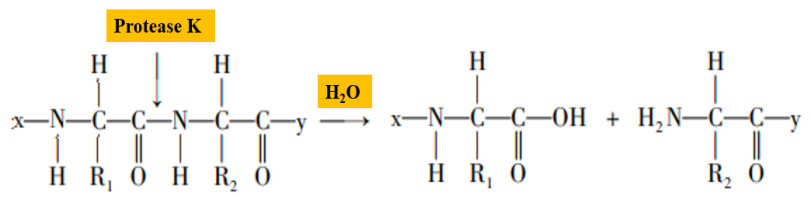
Abbildung 2. Schematische Darstellung der Proteinase K, die Peptidbindungen hydrolysiert
2.2 Desoxyribonuklease I
Desoxyribonuklease I (DNase I) kann verschiedene Formen von DNA katalysieren, gezielt Phosphodiesterbindungen neben Pyrimidinen spalten und Polynukleotide mit einer Phosphatgruppe am 5'-Ende und einer Hydroxylgruppe am 3'-Ende erzeugen. Das durchschnittliche Verdauungsprodukt ist das kleinste Polytetranukleotid.Bei der SARS-CoV-2-Nukleinsäureextraktion wird DNase I hauptsächlich verwendet, um genomische Verunreinigungen in RNA-Proben zu entfernen, DNA-Rückstände in RNA-Vorlagen zu vermeiden und die Reinheit der Vorlagen zu verbessern.
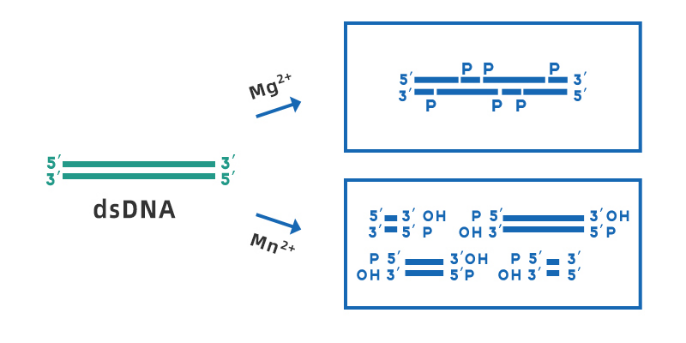
Abb. 3. Schematische Darstellung der Spaltung von dsDNA durch DNase I in Gegenwart von Mg2+ und Mn2+
2.3 RNase-Inhibitor
Beim Nachweis von SARS-CoV-2-Nukleinsäure kann es bei der Extraktion und Reinigung der Nukleinsäureprobe oder bei der Vorbereitung des experimentellen Reaktionssystems zu einer Kontamination mit Ribonuklease (RNase) kommen, was zum Abbau der RNA-Vorlage führt. Um eine RNase-Kontamination zu vermeiden, ist ein RNase-Inhibitor erforderlich.
RNase-Inhibitor ist ein spezifischer RNase-Inhibitor in der menschlichen Plazenta, der spezifisch RNase binden kann, um einen Komplex mit einer nicht-kovalenten Bindung zu bilden und RNase zu inaktivieren.
3. Kernenzyme während der RT-qPCR
Nachdem die Nukleinsäureextraktion der SARS-CoV-2-Probe abgeschlossen ist, kann die Nukleinsäureerkennung durch RT-qPCR abgeschlossen werden. Im Verlauf dieser Experimente sind DNA-Polymerase, Reverse Transkriptase und Uracil-DNA-Glycosylase allesamt wichtige Kernenzymrohstoffe.
3.1 Reverse Transkriptase
Nach der Extraktion und Reinigung benötigt die SARS-CoV-2-RNA eine Reverse Transkriptase, um die dNTP-Polymerisation zu katalysieren und eine cDNA-Sequenz zu erzeugen, die komplementär zur Matrizen-RNA ist (Abbildung 4). Für die RT-qPCR-Reaktion sollte eine hochtemperaturbeständige Reverse Transkriptase ausgewählt werden. Derzeit wird die MMLV-Reverse Transkriptase am häufigsten verwendet, da sie aufgrund ihrer fehlenden DNA-Endonuklease-Aktivität und ihrer geringen RNase H-Aktivität mehr Vorteile bei der Anwendung der cDNA-Klonierung bietet.
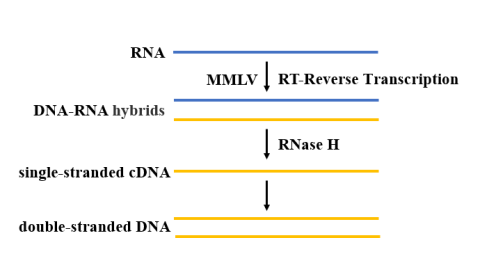
Abbildung 4. Schematische Darstellung des Reverse-Transkriptionsprozesses
3.2 DNA-Polymerase
Nachdem der Reverse-Transkriptionsprozess der Vorlage zur Erzeugung doppelsträngiger cDNA abgeschlossen ist, muss die „Seelenspieler“-DNA-Polymerase in der PCR-Reaktion in Erscheinung treten. Durch Polymerisation freier Desoxyribonukleotide wird die DNA-Kette verlängert und eine große Menge an Vorlagen-DNA wird in vitro amplifiziert, um den Zweck der Erkennung viraler Nukleinsäuren zu erreichen.
Die in RT-qPCR-Reaktionen üblicherweise verwendete DNA-Polymerase ist die Hot-Start-Taq-DNA-Polymerase. Dieser Enzymtyp ist bei Raumtemperatur inaktiv.Es weist nur nach dem Hot-Start eine Polymerisationsaktivität auf, wodurch die Entstehung von Hintergrundsignalen minimiert werden kann. Es löst die Probleme der unspezifischen Amplifikation, die durch Primer-Dimer-Erzeugung oder Fehlpaarung bei herkömmlichen PCR-Reaktionen verursacht werden. Derzeit umfassen die häufig verwendeten Hot-Start-Modifikationsmethoden für DNA-Polymerasen hauptsächlich chemische Modifikation, Ligandenmodifikation und Antikörpermodifikation. Die Prinzipien der verschiedenen Hot-Start-Modifikationsmethoden sind in Abbildung 5 dargestellt.
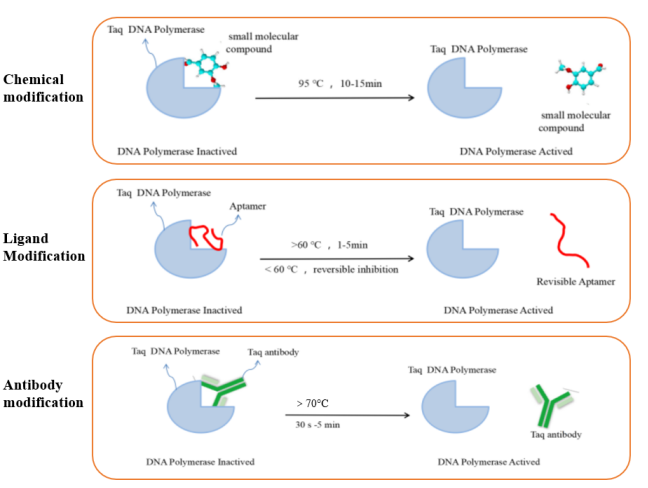
Abbildung 5. Schematische Darstellung verschiedener Arten modifizierter Hot-Start-Enzyme
3.3 Uracil-DNA-Glycosylase
Beim Nachweis neuer Coronavirus-Nukleinsäuren ist die Aerosolverschmutzung in der Betriebsumgebung der häufigste Faktor, der zu falsch positiven PCR-Ergebnissen führt. Durch Zugabe des UDG-Enzyms (Uracil-DNA-Glycosylase, Uracil-DNA-Glycosylase) zum Amplifikationssystem können die im PCR-System eingemischten Amplifikationsrestschadstoffe (meist in Form von Aerosolen) wirksam eliminiert werden, um die Genauigkeit der Amplifikationsergebnisse sicherzustellen. Das Anti-Verschmutzungsprinzip des UDG-Enzyms ist in Abbildung 6 dargestellt.
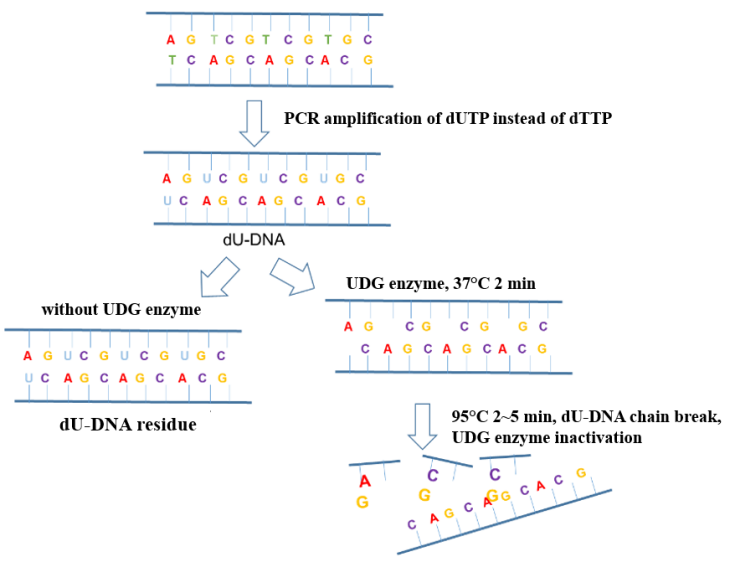
Abbildung 6. Schematische Darstellung des Anti-Pollution-Prinzips des UDG-Enzyms
4.Kernenzyme der SARS-CoV-2-Nukleinsäureerkennung von Yeasen
| Verfahren | Beschreibung | Produktname | Artikelnummer |
| Probenverarbeitung | Proteinverdauung | 10401ES | |
| RNA-Extraktion | Rekombinant DNase I (RNase-frei) (Anfragen) | 10325ES | |
| RNase-Hemmung | Muriner RNase-Inhibitor (40 Einheiten/µl) | 10603ES | |
| Umgekehrte Transkription | Geeignet für RT-qPCR | Hifair™ V Reverse Transkriptase (200 Einheiten/µl) | 11300ES |
| HifairTM V Reverse Transkriptase (600U/ μL) GLyceroL-frei (Anfrage) | 11301ES | ||
| PCR-Amplifikation | Hot-Start-DNA-Polymerase | 10726ES | |
| Thermisches UDG | Uracil-DNA-Glykosylase (UDG/UNG), hitzelabil, 1 U/μL | 10303ES |
Zum Thema Lesen:
Auswahl der Reverse-Transkriptase
YEASEN Hitzelabiles UDG—— Aerosolverschmutzung leicht kontrollieren
Murine RNase-Inhibitoren - RNase-Kontamination erfolgreich eliminieren und RNA erhalten
