ハイスループット シーケンシングは、次世代シーケンシング (NGS) 技術とも呼ばれ、サンガー シーケンシングなどの初期の DNA シーケンシング方法から大きく進歩したものです。NGS では、何十万、あるいは何百万もの核酸分子配列を同時にプロファイリングできます。その利点には、優れたスループット、コスト効率、拡張性、幅広い用途などがあり、世界中で主流のシーケンシング技術としての地位を確立しています。
NGS シーケンシング ワークフローには、サンプルの準備、ライブラリの構築、シーケンシング、データ分析という 4 つの主要な段階があります。ライブラリ構築の中心となるのは、標準化された NGS プラットフォーム アダプター シーケンスを断片化されたゲノム DNA の両端に付加することです。このステップの目的は、PCR 増幅によって NGS 機器でシーケンシングできるように準備されたライブラリ核酸分子を十分に生成することです。サンプルの性質に応じて、NGS ライブラリ構築は DNA ライブラリ構築と RNA ライブラリ構築に分類できます。酵素は、これらの相互接続された実験で極めて重要な役割を果たします。では、ライブラリ構築のプロセスに関係する主要な酵素は何でしょうか。
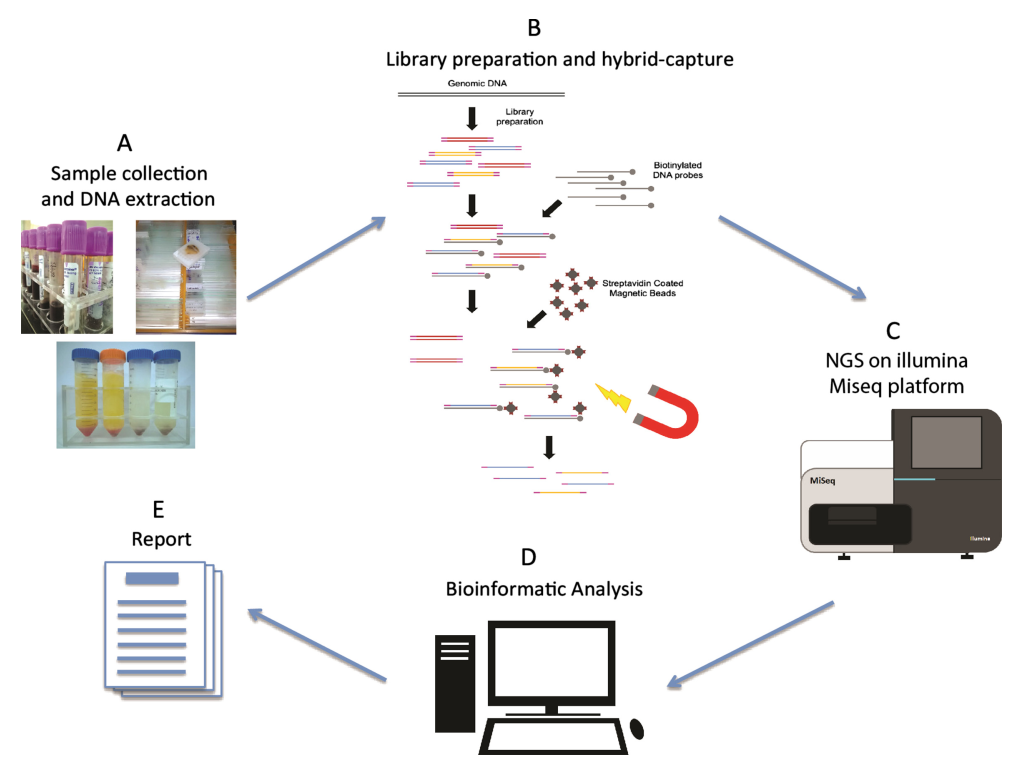
図1. 次世代シーケンシングワークフロー[2]
1. DNAライブラリ構築とその鍵となる酵素
2. RNAライブラリ構築とその鍵酵素
3. DNAおよびRNAライブラリ構築におけるNGSコア酵素のガイドライン
1. DNAライブラリ構築とその鍵となる酵素
DNA ライブラリ構築のプロセスでは、TA クローンライゲーションアダプターライブラリ構築が現在最も一般的に使用されている技術手段です。主なライブラリ構築プロセスは次のとおりです。

図2. DNAライブラリ構築プロセス(イルミナ)
1.1 DNAの断片化
現在のシーケンサーのシーケンシング長は通常 150 ~ 500 塩基対 (bp) の範囲です。そのため、大きなゲノム DNA 断片を小さな断片に分解するには、機械的または酵素的断片化法を使用する必要があります。機械的断片化ではサンプル損失が比較的大きくなり、より複雑な操作プロセスが必要になります。一方、酵素消化はゲノム DNA の断片化によく使用される方法です。機械的方法と比較すると、酵素消化はより費用対効果が高く、断片化酵素の添加後、一定時間反応するだけで済みます。
現在、主に 2 種類のフラグメントが使用されています。1 つはトランスポゾンの原理に基づく Tn5 トランスポザーゼに依存し、もう 1 つはエンドヌクレアーゼの混合物を使用します。ただし、これらのフラグメントの有効性は、DNA の GC 含有量と塩基の好みによって左右される可能性があります。対照的に、
1.2 端部修復、dAテーリング
断片化された DNA は 5'/3' 粘着末端と平滑末端 DNA を生成し、3' オーバーハングの除去と 5' 突出 DNA 末端の埋め戻しを含め、すべての粘着末端を平滑末端に変換する必要があります。アダプター連結に TA 連結を使用する場合、DNA 断片も 5' 末端でリン酸化され、3' 末端に「A」が追加されて、「T」粘着末端を持つアダプターと相補的になります。上記のプロセスは、T4 DNA ポリメラーゼ、T4 ポリヌクレオチド キナーゼ、およびTaq DNA ポリメラーゼの連携によって完了します。
T4 DNAポリメラーゼ(カタログ番号12901) 5'→3' DNAポリメラーゼ活性を持ち、5'→3'方向に沿ったDNA合成を触媒し、5'突出末端を埋めることができます。同時に、この酵素は3'→5'エキソヌクレアーゼ活性も持ち、3'突出末端を切断し、粘着末端を含むDNA断片を平滑末端DNAに変換します。
合成 PCR プライマーおよびアダプターの 5' 末端は通常、リン酸基ではなくヒドロキシル基です。したがって、アダプター連結の次のステップに備えて、ATP の存在下でオリゴヌクレオチド鎖の 5' ヒドロキシル末端への ATP γ リン酸基の転移を触媒するには、T4 ポリヌクレオチド キナーゼ (Cat#12902) が必要です。
S-Taq DNA ポリメラーゼ(Cat#13486) は、5'→3' ポリメラーゼ活性を持ち、5'→3' 方向から DNA を合成できます。また、デオキシヌクレオチド転移酵素活性を持ち、PCR 産物の 3' 末端にヌクレオチド「A」を追加できます。

図3.末端修復プロセスには複数の酵素が関与している
図 4. S-taq は、キャピラリー電気泳動で検出された遺伝子セグメントの 3' 末端の ATCG の 4 つの塩基に A を付加する効率が非常に高い。
1.3 アダプターライゲーション
アダプターはライブラリの重要なコンポーネントを構成します。イルミナ シーケンシングの分野では、一般的に使用される Y 型アダプターには、P5/P7、インデックス、および Rd1/Rd2 SP シーケンスが含まれます。これらのうち、P5/P7 シーケンスは、シーケンシング チップ上に存在するシーケンスとペアリングする目的で使用され、分析するフラグメントをフロー セルに固定してブリッジ増幅を実行します。インデックス シーケンスは、シーケンシングの対象となる混合ライブラリ内の異なるサンプルを区別するために使用され、Rd1/Rd2 SP は、Read1 および Read2 シーケンシング プライマーを結合する領域を示します。
アダプター連結のタスクでは、 T4 DNA リガーゼ (Cat#12996) が標準的な選択肢です。このリガーゼは、二本鎖 DNA に見られる一本鎖の切れ目を修復し、隣接するヌクレオチドを再接続する機能を備えています。
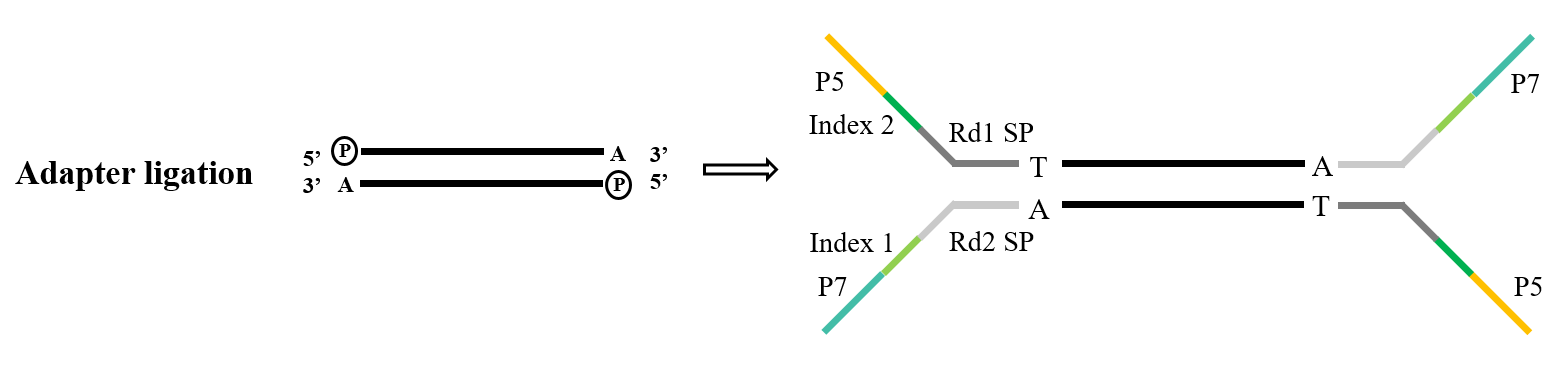
図5. 一般的なアダプターライゲーションプロセス(イルミナ)

図 6. 170 bp DNA を 80 bp アダプターと連結することによる T4 DNA リガーゼ変異体の検証。
1.4 PCR増幅
PCR反応により、アダプター付きの十分なDNA配列を取得し、サンプル核酸配列のシークエンシングを機械で完了します。PCRでよく使用されるHieff Canace TM Pro High-Fidelity DNAポリメラーゼ(カタログ番号13476)は、5'→3'ポリメラーゼ活性を持ち、5'→3'方向のDNAを合成できます。さらに、3'→5'エキソヌクレアーゼ活性も持ち、増幅プロセス中に誤った塩基の組み込みを修正し、DNA断片を迅速かつ忠実に増幅できます。
2. RNAライブラリ構築とその鍵酵素
RNA の種類に応じて、RNA ライブラリの構築は、mRNA ライブラリ、LncRNA ライブラリなどに分けられます。従来の RNA ライブラリには、次のプロセスが含まれます。
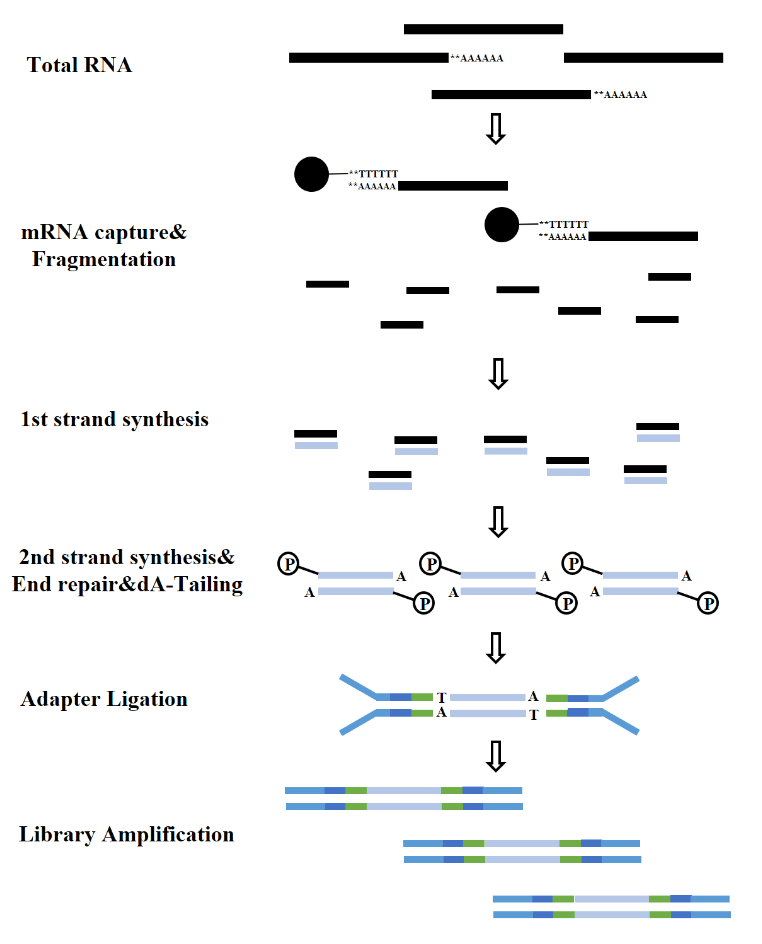
図7. mRNAライブラリ構築プロセス(イルミナ)
2.1 RNA濃縮
真核生物でも原核生物でも、リボソーム RNA (rRNA) は最も豊富な RNA として際立っており、総 RNA 含有量の最大 80% を占めています。サンプルの総 RNA を直接シーケンスすると、シーケンス データの大部分が rRNA に関連します。この干渉を軽減するには、RNA エンリッチメント法を使用する必要があります。これには、オリゴ dT に基づく mRNA エンリッチメント法と rRNA 除去法という 2 つの主な方法があります。
真核生物では、mRNA は 3' 末端に明確なポリ (A) 構造を示します。オリゴ dT ビーズを使用すると、サンプルから転写されたすべての mRNA を捕捉できるため、特に高品質の RNA サンプルでは転写分析に適しています。一方、rRNA 除去法ではサンプル品質の要件が緩やかで、低品質サンプル (FFPE サンプルなど) と高品質 RNA サンプルの両方、および原核生物サンプルに適用できます。一般的に使用されている商業的アプローチでは、以下の特定の手順に従って RNase H 消化を使用して rRNA を除去します。
- rRNA に結合するように設計された特定のオリゴヌクレオチドプローブを合成します。
- RNA-DNA ハイブリッド鎖内の RNA を分解できる RNase H (Cat#12906) を使用して、プローブに結合した rRNA を選択的に除去します。
- 最後に、DNA プローブをDNase I (Cat#10325) で消化します。これにより、一本鎖 DNA と二本鎖 DNA の両方が分解され、rRNA が効果的に除去されます。DNase I の詳細については、このリンクを参照してください。
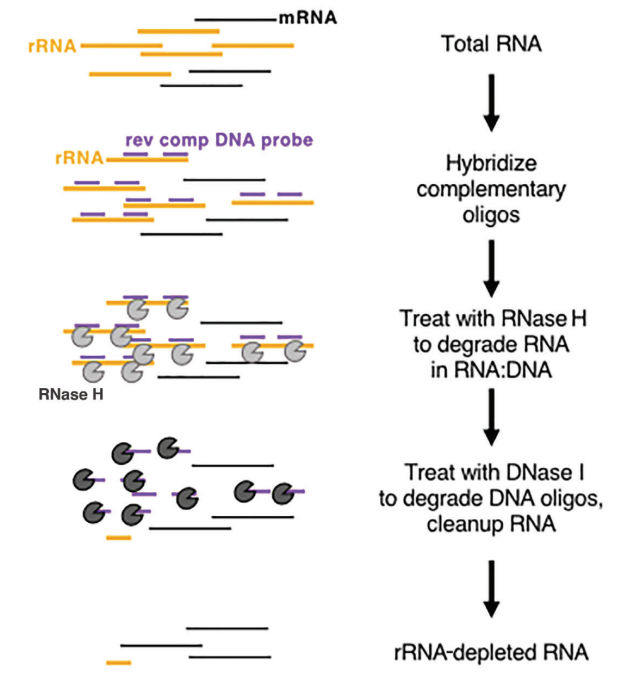
図8: 酵素ベースのrRNA除去の模式図[5]
2.2 RNAの断片化
通常、二価金属カチオンと高温の作用により、RNA の大きな断片は小さな断片に分解されます。
2.3 1st 鎖 cDNA 合成
得られたターゲット RNA を cDNA の第一鎖に逆転写します。RNA は環境中に存在する RNase によって容易に分解されるため、逆転写中にRNase 阻害剤 (Cat#14672)を使用すると、これらの酵素の活性を阻害し、RNA を RNase 分解から保護できます。同時に、逆転写酵素 (Cat#11112)を使用して、テンプレート RNA を cDNA に逆転写しました。逆転写酵素は RNA 依存性 DNA ポリメラーゼ活性を持ち、RNA をテンプレートとして使用して 5'→3' 方向に cDNA を合成できます。DNA の一本鎖は RNA テンプレートと相補的です。
1st ストランド cDNA 合成中にアクチノマイシン D を組み込むことで、ストランド特異的ライブラリの構築が明らかに改善され、鎖特異性が大幅に向上しました。この革新により、実験プロセスが合理化され、研究者にとって簡素化されました。
しかし、アクチノマイシン D には欠点もあります。毒性があり、光から保護する必要があります。プレミックス キットやプレート ライブラリ構築キットの需要が高まっている今日では、光から保護する必要があるため、プレート キットの進歩に制限が生じます。
幸いなことに、

図9: MMLVのエンジニアリングにより、Standed RNA-seqに寄与する可能性のあるMMLV変異体を特定
2.4 2本鎖cDNA合成
逆転写によって生成された一本鎖 cDNA は非常に不安定であるため、DNA ポリメラーゼ I の影響下で cDNA の 2 番目の鎖を直ちに合成する必要があります。この 2 番目の鎖の合成中に、RNase H が作用して RNA 鎖を RNA-DNA ハイブリッド構造から除去します。RNase H はDNA ポリメラーゼ I (Cat#12903)と連携して、相補的な cDNA の 2 番目の鎖の触媒合成を促進します。DNA ポリメラーゼ I は 5'→3' DNA ポリメラーゼ活性を持ち、テンプレートとプライマーによって誘導され、5'→3' 方向で一本鎖 cDNA を補完する配列を合成します。
プロセスのその後のステップには、末端修復、dA テーリング、アダプター連結、PCR 増幅が含まれますが、これらはすべて DNA ライブラリ構築手順で詳しく説明されているため、ここで繰り返す必要はありません。逆転写が完了したら、核酸フラグメントをさらに断片化する必要がないことに注意してください。
3. DNAおよびRNAライブラリ構築におけるNGSコア酵素のガイドライン
表1. DNAおよびRNAライブラリ構築におけるNGSコア酵素のガイドライン
|
タイプ |
製品の位置付け |
製品名 |
猫# |
|
RNAライブラリの構築 |
rRNA除去/2本鎖cDNA合成 |
12906ES |
|
|
rRNAの枯渇 |
10325ES |
||
|
1st 鎖 cDNA 合成 |
14672ES |
||
|
11112ES |
|||
|
2本鎖cDNA合成 |
12903ES |
||
|
RNAライブラリ構築とDNAライブラリ構築 |
修理終了 |
12901ES |
|
|
12902ES |
|||
|
dA-テーリング |
13486ES |
||
|
アダプターライゲーション |
10301ES |
||
|
PCR増幅 |
12621ES |
表2. DNA & RNA ライブラリ調製キット
| 名前 | 猫# | 注記 | |
| DNA | Hieff NGS DNA ライブラリ準備キット | 13577ES | 腫瘍/メカニック法 |
| Hieff NGS OnePot Pro DNA ライブラリ準備キット V2 | 12194ES | 腫瘍/酵素法 | |
| Hieff NGS OnePot II DNA ライブラリ準備キット (Illumina 用) | 13490ES | Pathgen/酵素/通常時間(140分) | |
| Hieff NGS OnePot フラッシュ DNA ライブラリ準備キット | 12316ES | Pathgen/酵素/超高速(100分) | |
| Hieff NGS DNA&RNA ライブラリー Co-Prep キット V2 | 12305ES | Pathgen/酵素/DNAとRNAの共同調製 | |
| RNA | Hieff NGS Ultima デュアルモード mRNA ライブラリ調製キット | 12308ES | オリゴdT磁気ビーズなし、チューブ11本 |
| Hieff NGS Ultima デュアルモード mRNA ライブラリ調製キット | 12309ES | オリゴ dT 磁気ビーズ プラス、14 チューブ | |
| Hieff NGS® Ultima デュアルモード RNA ライブラリ調製キット | 12310ES | プレミックスバージョン、5本入り | |
| Hieff NGS ® EvoMax RNA ライブラリ調製キット(プレミックス版)(アクチノマイシン D フリー) | 12340ES | プレミックスバージョン(アクチノマイシンDフリー) | |
| Hieff NGS® MaxUp rRNA 除去キット (植物) | 12254ES | 植物 | |
| Hieff NGS® MaxUp ヒト rRNA 除去キット (rRNA & ITS/ETS) | 12257ES | 人間 |
参考文献:
[1] Mardis, Elaine R. 次世代シーケンシングプラットフォーム[J]. Annual Review of Analytical Chemistry, 2013, 6(1):287-303.
[2] Gulilat M、Lamb T、Teft WA、et al. 切除医療のためのツールとしての標的次世代シーケンシング[J]。BMCメディカルゲノミクス、2019、12(1):81。
[3] Lundberg KS, Dan DS, Adams M, et al. Pyrococcus furiosusから単離された耐熱性DNAポリメラーゼを用いた高忠実度増幅[J]. Gene, 1991, 108(1):1-6.
[4] 宮崎 功. エンドヌクレアーゼVによるランダムDNA断片化:DNAシャッフリングへの応用[J]. 核酸研究、2002、30(24):e139。
[5] Baldwin A、Morris AR、Mukherjee N. RNA-seqのためのヒトリボソームRNAを枯渇させる簡単で費用対効果が高くスケーラブルな方法[J]。Current Protocols、2021、1(6):e176。

