고처리량 시퀀싱은 종종 차세대 시퀀싱(NGS) 기술이라고도 하며, 샌저 시퀀싱과 같은 초기 DNA 시퀀싱 방법에서 크게 도약한 것입니다. NGS는 수백만 개가 아니라면 수십만 개의 핵산 분자 시퀀스를 동시에 프로파일링할 수 있습니다. 이 기술의 장점으로는 뛰어난 처리량, 비용 효율성, 확장성, 광범위한 응용 분야가 있으며, 이를 통해 전 세계적으로 우세한 시퀀싱 기술로 자리매김했습니다.
NGS 시퀀싱 워크플로는 샘플 준비, 라이브러리 구축, 시퀀싱, 데이터 분석의 네 가지 주요 단계로 구성됩니다. 라이브러리 구축의 핵심은 표준화된 NGS 플랫폼 어댑터 시퀀스를 단편화된 게놈 DNA의 양쪽 끝에 부착하는 것입니다. 이 단계는 PCR 증폭을 통해 NGS 기기에서 시퀀싱을 위해 준비된 충분한 라이브러리 핵산 분자를 생성하는 것을 목표로 합니다. 샘플의 특성에 따라 NGS 라이브러리 구축은 DNA 라이브러리 구축과 RNA 라이브러리 구축으로 분류할 수 있습니다. 효소는 이러한 상호 연결된 실험에서 중요한 역할을 합니다. 그렇다면 라이브러리 구축 과정에는 어떤 핵심 효소가 관여할까요?
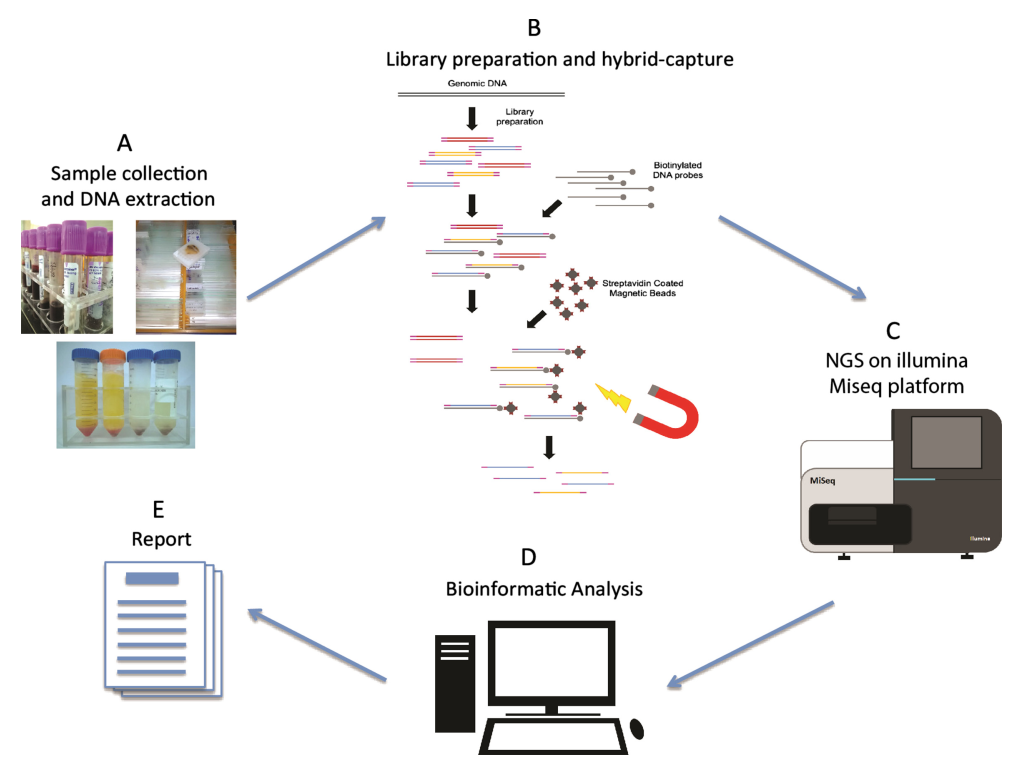
그림 1. 차세대 시퀀싱 워크플로우 [2]
1. DNA 라이브러리 구축 및 핵심 효소
2. RNA 라이브러리 구축 및 주요 효소
3. DNA & RNA 라이브러리 구축 시 NGS 코어 효소에 대한 가이드라인
1. DNA 라이브러리 구축 및 핵심 효소
DNA 라이브러리 구축 과정에서 TA 클론 라이게이션 어댑터 라이브러리 구축은 현재 가장 일반적으로 사용되는 기술 수단입니다. 주요 라이브러리 구축 프로세스는 다음과 같습니다.

그림 2. DNA 라이브러리 구축 과정 (Illumina)
1.1 DNA 단편화
현재 시퀀서는 일반적으로 150-500 염기쌍(bp) 범위의 시퀀싱 길이를 갖습니다. 결과적으로 큰 게놈 DNA 단편을 더 작은 단편으로 분해하기 위해 기계적 또는 효소적 단편화 방법을 채택해야 합니다. 기계적 단편화는 비교적 높은 샘플 손실로 이어질 수 있으며 더 복잡한 운영 프로세스를 수반합니다. 반면, 효소적 소화는 게놈 DNA를 단편화하는 데 일반적으로 사용되는 방법입니다. 기계적 방법과 비교할 때, 효소적 소화는 더 비용 효율적이고 간단하며, 단편화 효소를 첨가한 후 반응에 일정 기간만 필요합니다.
현재 주로 두 가지 유형의 단편이 사용되고 있습니다. 하나는 트랜스포존 원리에 기반한 Tn5 트랜스포사제에 의존하는 반면, 다른 하나는 엔도뉴클레아제 혼합물을 활용합니다. 그러나 이러한 단편의 효과는 DNA의 GC 함량과 염기 선호도에 의해 영향을 받을 수 있습니다. 반면,
1.2 끝단수리, dA-테일링
단편화된 DNA는 5'/3' 스티키 엔드와 블런트 엔드 DNA를 생성하며, 모든 스티키 엔드는 3' 오버행을 제거하고 5'-돌출된 DNA 엔드를 채워서 블런트 엔드로 변환해야 합니다. 어댑터 라이게이션에 TA 라이게이션을 사용할 때, DNA 단편은 5' 엔드에서 인산화되어야 하며, "T" 스티키 엔드가 있는 어댑터와 상보적이 되도록 3' 엔드에 "A"를 추가해야 합니다. 위의 과정은 T4 DNA 중합효소, T4 폴리뉴클레오티드 키나제, Taq DNA 중합효소의 협력으로 완료됩니다.
T4 DNA 중합효소(Cat#12901) 5'→3' DNA 중합효소 활성을 가지고 있어 5'→3' 방향으로 DNA 합성을 촉진하고 5' 돌출부를 채울 수 있습니다. 동시에 이 효소는 3'→5' 엑소뉴클레아제 활성도 가지고 있어 3' 돌출부를 절단하여 끈적끈적한 끝을 포함하는 DNA 단편을 평활한 끝 DNA로 변환합니다.
합성 PCR 프라이머와 어댑터의 5' 말단은 일반적으로 인산기 대신 히드록실기입니다. 따라서 T4 폴리뉴클레오티드 키나제(Cat#12902)는 ATP가 존재하는 상태에서 올리고뉴클레오티드 사슬의 5'-히드록실 말단으로 ATP γ-인산기를 전달하는 것을 촉매하여 다음 단계인 어댑터 결찰을 준비하는 데 필요합니다.
S-Taq DNA 중합효소 (Cat#13486)는 5'→3' 중합효소 활성을 가지고 있어 5'→3' 방향에서 DNA를 합성할 수 있습니다. 한편, 데옥시뉴클레오티딜 전이효소 활성을 가지고 있어 PCR 생성물의 3' 끝에 뉴클레오타이드 "A"를 추가할 수 있습니다.

그림 3. 최종 복구 과정에는 여러 효소가 관여합니다.
그림 4. S-taq는 모세관 전기영동으로 검출된 유전자 단편의 3' 말단의 ATCG의 4개 염기에 A를 첨가하는 효율이 매우 높습니다.
1.3 어댑터 결찰
어댑터는 라이브러리의 중요한 구성 요소입니다. Illumina 시퀀싱의 영역 내에서 일반적으로 사용되는 Y형 어댑터는 P5/P7, Index 및 Rd1/Rd2 SP 시퀀스를 포함합니다. 이 중 P5/P7 시퀀스는 시퀀싱 칩에 있는 시퀀스와 페어링하여 분석할 조각을 유동 셀에 고정하여 브릿지 증폭을 실행하는 목적을 제공합니다. Index 시퀀스는 시퀀싱을 거친 혼합 라이브러리 내의 서로 다른 샘플을 구별하는 데 사용되는 반면 Rd1/Rd2 SP는 Read1 및 Read2 시퀀싱 프라이머를 결합하는 영역을 나타냅니다.
어댑터 라이게이션 작업의 경우 T4 DNA 리가제(Cat#12996)가 표준 선택입니다. 이는 이중 가닥 DNA에서 발견되는 단일 가닥 틈을 복구하고 인접한 뉴클레오티드를 다시 연결하는 기능을 보여줍니다.
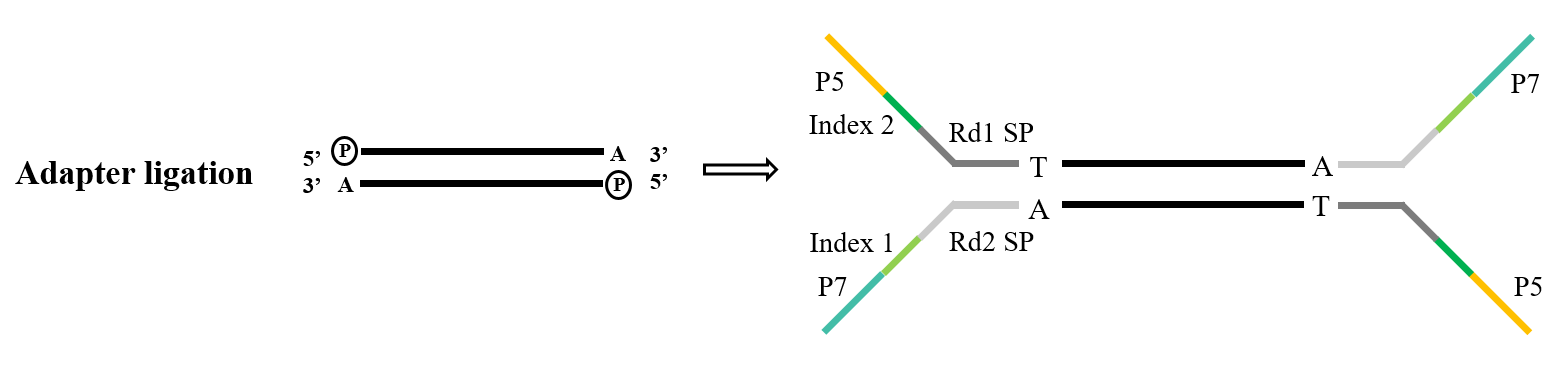
그림 5. 일반적인 어댑터 결찰 과정(Illumina)

그림 6. 80bp 어댑터로 170bp DNA를 연결하여 T4 DNA 리가제 돌연변이체 확인.
1.4 PCR 증폭
PCR 반응을 통해 어댑터로 충분한 DNA 시퀀스를 얻고, 기계에서 샘플 핵산 시퀀스의 시퀀싱을 완료합니다. PCR에서 일반적으로 사용되는 Hieff Canace TM Pro High-Fidelity DNA Polymerase(Cat#13476)는 5'→3' 중합효소 활성을 가지고 있으며 5'→3' 방향으로 DNA를 합성할 수 있습니다. 또한 3'→5' 엑소뉴클레아제 활성도 있어 증폭 과정에서 잘못된 염기 삽입을 교정하여 DNA 단편을 빠르고 높은 충실도로 증폭할 수 있습니다.
2. RNA 라이브러리 구축 및 주요 효소
RNA의 종류에 따라 RNA 라이브러리의 구성은 mRNA 라이브러리, LncRNA 라이브러리 등으로 나눌 수 있다. 기존의 RNA 라이브러리는 다음과 같은 과정을 거친다.
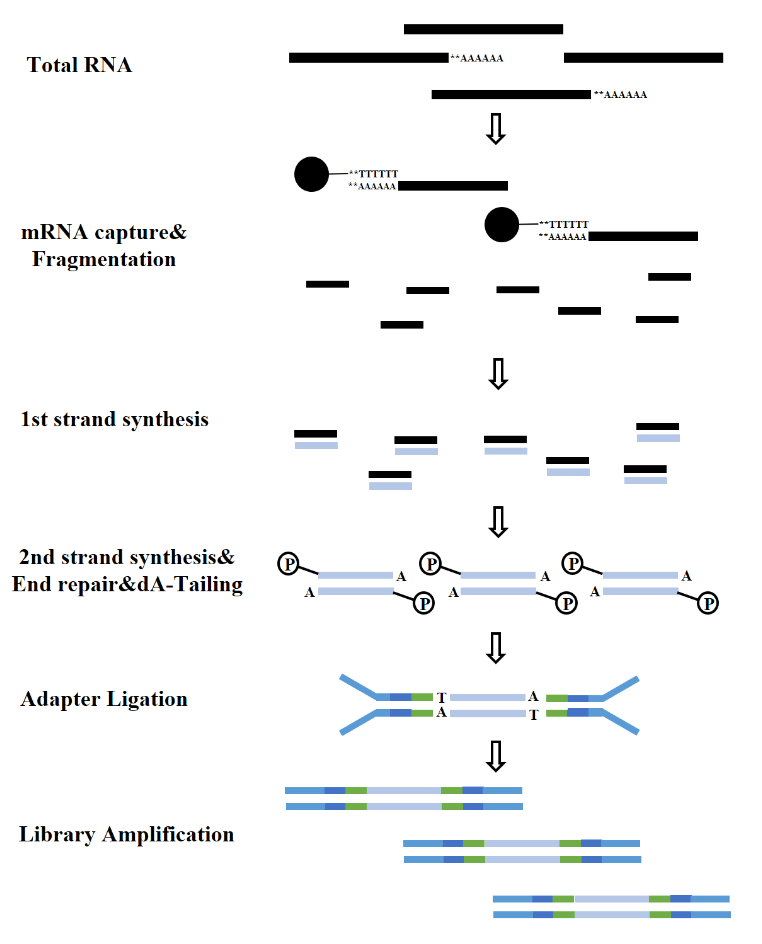
그림 7. mRNA 라이브러리 구축 과정(Illumina)
2.1 RNA 풍부화
진핵생물이든 원핵생물이든 리보솜 RNA(rRNA)는 가장 풍부한 RNA로 두드러지며, 총 RNA 함량의 최대 80%를 구성합니다. 샘플의 총 RNA를 직접 시퀀싱할 때 시퀀싱 데이터의 상당 부분은 rRNA와 관련이 있습니다. 이러한 간섭을 완화하려면 RNA 농축 방법을 사용해야 합니다. 이를 위한 두 가지 주요 방법이 있습니다. 올리고-dT 기반 mRNA 농축과 rRNA 고갈 방법입니다.
진핵생물에서 mRNA는 3' 말단에서 뚜렷한 폴리(A) 구조를 보인다. Oligo-dT 비드는 샘플에서 전사된 모든 mRNA를 포획하는 데 사용될 수 있으므로, 특히 고품질 RNA 샘플의 경우 전사 분석에 적합하다. 반면, rRNA 고갈 방법은 샘플 품질에 대한 요구 사항이 더 관대하며 저품질 샘플(예: FFPE 샘플)과 고품질 RNA 샘플, 그리고 원핵생물 샘플에 모두 적용할 수 있다. 일반적으로 사용되는 상업적 접근 방식은 RNase H 소화를 사용하여 rRNA를 제거하는 것으로, 다음과 같은 특정 단계를 따른다.
- rRNA에 결합하도록 설계된 특정 올리고뉴클레오티드 프로브를 합성합니다.
- RNA-DNA 하이브리드 가닥에서 RNA를 분해할 수 있는 RNase H(Cat#12906)를 사용하여 프로브에 결합된 rRNA를 선택적으로 제거합니다.
- 마지막으로, DNA 프로브를 DNase I(Cat#10325)로 소화합니다. 이는 단일 및 이중 가닥 DNA를 모두 분해하여 rRNA를 효과적으로 제거할 수 있습니다. DNase I에 대한 자세한 내용은 이 링크를 따라가면 됩니다.
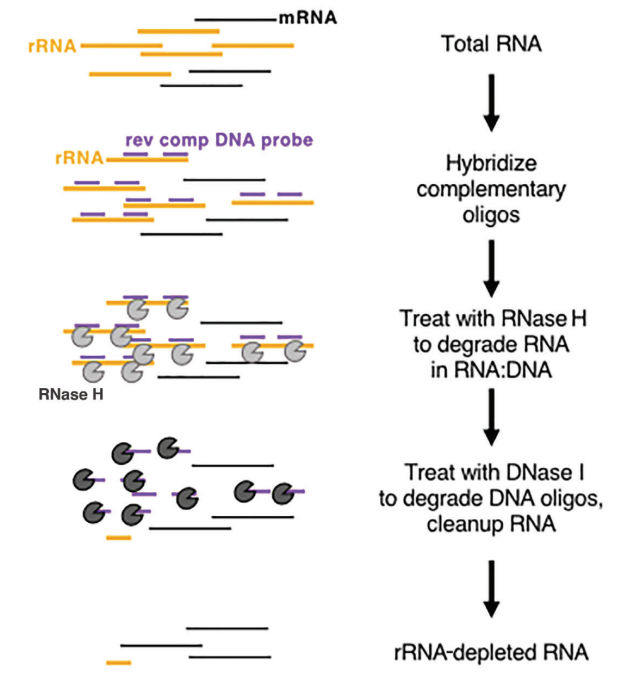
그림 8: 효소 기반 rRNA 고갈의 개략도 [5]
2.2 RNA 단편화
일반적으로 2가 금속 양이온과 고온의 작용으로 큰 RNA 조각이 작은 조각으로 분해됩니다.
2.3 1st strand cDNA 합성
획득한 타겟 RNA를 cDNA의 첫 번째 가닥으로 역전사합니다. RNA는 환경에 존재하는 RNase에 의해 쉽게 분해되기 때문에 역전사 중에 RNase Inhibitor(Cat#14672) 를 사용하면 이러한 효소의 활성을 억제하고 RNA를 RNase 분해로부터 보호할 수 있습니다. 동시에 역전사효소(Cat#11112)를 사용하여 템플릿 RNA를 cDNA로 역전사했습니다. 역전사효소는 RNA 의존성 DNA 중합효소 활성을 가지고 있으며 RNA를 템플릿으로 사용하여 5'→3' 방향으로 cDNA를 합성할 수 있습니다. DNA의 단일 가닥은 RNA 템플릿과 상보적입니다.
1차 가닥 cDNA 합성 중에 액티노마이신 D를 첨가하면 가닥 특이적 라이브러리의 구성이 확실히 개선되어 사슬 특이성이 크게 향상되었습니다. 이 혁신은 실험 과정을 간소화하여 연구자들에게 단순화를 제공했습니다.
그러나 액티노마이신 D에는 단점이 있습니다. 독성이 있고 빛으로부터 보호해야 합니다. 오늘날 사전 혼합 및 플레이트 라이브러리 구축 키트에 대한 수요가 증가하는 상황에서 빛으로부터 보호해야 할 필요성은 플레이트 키트 발전에 제한을 가합니다.
다행히도

그림 9: Standed RNA-seq에 기여할 수 있는 MMLV 돌연변이를 식별하기 위한 MMLV 엔지니어링
2.4 2nd strand cDNA 합성
역전사를 통해 생성된 단일 가닥 cDNA는 매우 불안정하여 DNA 중합효소 I의 영향 하에 두 번째 cDNA 가닥을 즉시 합성해야 합니다. 이 두 번째 가닥 합성 중에 RNase H가 RNA 가닥을 RNA-DNA 하이브리드 구조에서 제거하여 작용합니다. DNA 중합효소 I(Cat#12903) 과 협력하여 보완적인 두 번째 cDNA 가닥의 촉매 합성을 용이하게 합니다. DNA 중합효소 I은 5'→3' DNA 중합효소 활성을 가지고 있으며, 템플릿과 프라이머의 안내를 받아 5'→3' 방향으로 단일 가닥 cDNA를 보완하는 서열을 합성합니다.
이 과정의 후속 단계에는 말단 복구, dA-테일링, 어댑터 라이게이션, PCR 증폭이 포함되며, 이는 모두 DNA 라이브러리 구축 절차에 자세히 설명되어 있으므로 여기서 반복할 필요가 없습니다. 역전사가 완료되면 핵산 단편을 더 이상 단편화할 필요가 없다는 점에 유의해야 합니다.
3. DNA & RNA 라이브러리 구축 시 NGS 코어 효소에 대한 가이드라인
표 1. DNA & RNA 라이브러리 구축 시 NGS 코어 효소에 대한 가이드라인
|
유형 |
제품 포지셔닝 |
제품 이름 |
고양이# |
|
RNA 라이브러리 구축 |
rRNA 고갈/2nd strand cDNA 합성 |
12906ES |
|
|
rRNA 고갈 |
10325ES |
||
|
1번째 가닥 cDNA 합성 |
14672ES |
||
|
11112ES |
|||
|
2nd strand cDNA 합성 |
12903ES |
||
|
RNA 라이브러리 구축 & DNA 라이브러리 구축 |
수리 종료 |
12901ES |
|
|
12902ES |
|||
|
dA-테일링 |
13486ES |
||
|
어댑터 결찰 |
10301ES |
||
|
PCR 증폭 |
12621ES |
표2. DNA & RNA 라이브러리 준비 키트
| 이름 | 고양이# | 노트 | |
| DNA | Hieff NGS DNA 라이브러리 준비 키트 | 13577ES | 종양/기계적 방법 |
| Hieff NGS OnePot Pro DNA 라이브러리 준비 키트 V2 | 12194ES | 종양/효소법 | |
| Illumina용 Hieff NGS OnePot II DNA 라이브러리 준비 키트 | 13490ES | 병원체/효소/정규시간(140분) | |
| Hieff NGS OnePot Flash DNA 라이브러리 준비 키트 | 12316ES | 병원체/효소/초고속(100분) | |
| Hieff NGS DNA&RNA 라이브러리 공동 준비 키트 V2 | 12305ES | 병원체/ 효소/ DNA & RNA 공동 준비 | |
| RNA | Hieff NGS Ultima 듀얼모드 mRNA 라이브러리 준비 키트 | 12308ES | 올리고 dT 자기 비드 없음, 11개 튜브 |
| Hieff NGS Ultima 듀얼모드 mRNA 라이브러리 준비 키트 | 12309ES | 올리고 dT 자기 비드 플러스, 14개 튜브 | |
| Hieff NGS® Ultima 듀얼모드 RNA 라이브러리 준비 키트 | 12310ES | 사전 혼합 버전, 5개 튜브 | |
| Hieff NGS ® EvoMax RNA 라이브러리 준비 키트(사전 혼합 버전)(액티노마이신 D 무함유) | 12340ES | 사전 혼합 버전(Actinomycin D Free) | |
| Hieff NGS® MaxUp rRNA 고갈 키트(식물) | 12254ES | 식물 | |
| Hieff NGS® MaxUp 인간 rRNA 고갈 키트(rRNA 및 ITS/ETS) | 12257ES | 인간 |
참고문헌:
[1] Mardis, Elaine R. 차세대 시퀀싱 플랫폼[J]. 분석 화학 연례 리뷰, 2013, 6(1):287-303.
[2] Gulilat M, Lamb T, Teft WA, et al. 정밀 의학을 위한 도구로서의 표적형 차세대 시퀀싱[J]. BMC Medical Genomics, 2019, 12(1):81.
[3] Lundberg KS, Dan DS, Adams M, et al. Pyrococcus furiosus에서 분리한 열안정 DNA 중합효소를 사용한 고충실도 증폭[J]. Gene, 1991, 108(1):1-6.
[4] Miyazaki K. 엔도뉴클레아제 V를 이용한 무작위 DNA 단편화: DNA 셔플링에 대한 응용[J]. Nucleic Acids Research, 2002, 30(24):e139.
[5] Baldwin A, Morris AR, Mukherjee N. RNA-seq[J]를 위한 인간 리보솜 RNA를 고갈시키는 쉽고 비용 효율적이며 확장 가능한 방법. Current Protocols, 2021, 1(6):e176.

