高通量测序通常被称为新一代测序 (NGS) 技术,与最初的 DNA 测序方法(例如桑格测序)相比,它代表了一次重大飞跃。NGS 可以同时分析数十万甚至数百万个核酸分子序列。它的优点包括卓越的通量、成本效益、可扩展性和广泛的应用范围,使其成为全球主要的测序技术。
NGS 测序工作流程包括四个主要阶段:样本制备、文库构建、测序和数据分析。文库构建的核心是将标准化的 NGS 平台接头序列连接到片段化基因组 DNA 的两端。此步骤旨在生成充足的文库核酸分子,通过 PCR 扩增为 NGS 仪器上的测序做好准备。根据样本的性质,NGS 文库构建可分为 DNA 文库构建和 RNA 文库构建。酶在这些相互关联的实验中起着关键作用。那么,哪些关键酶参与了文库构建过程?
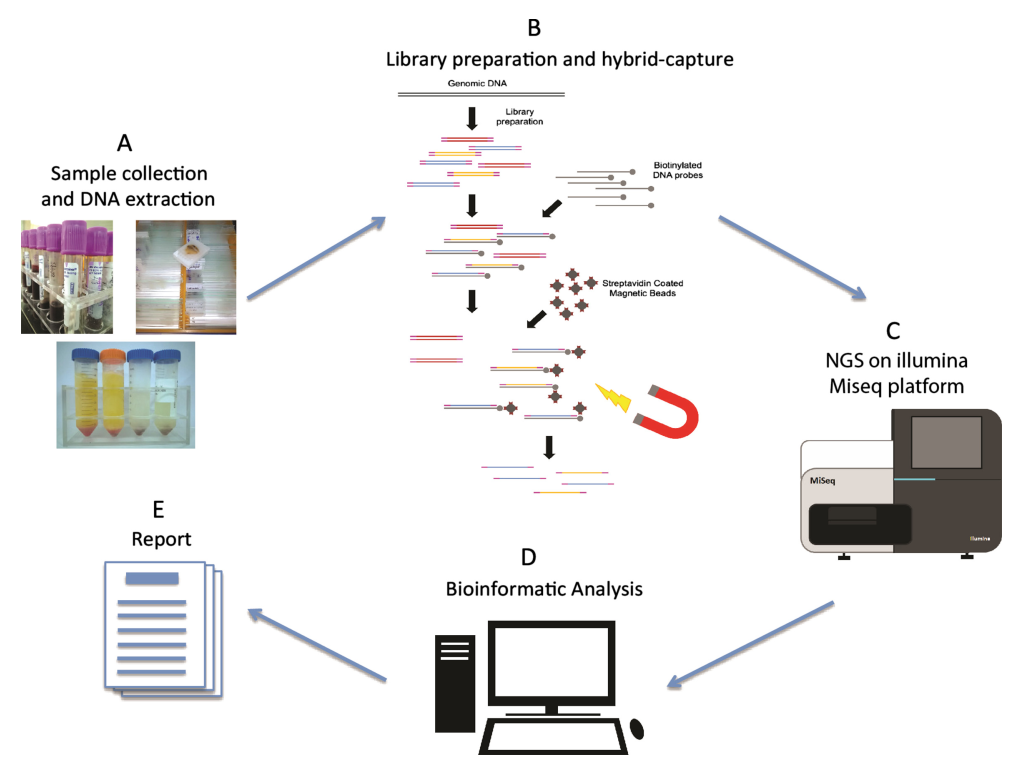
图 1. 新一代测序工作流程[2]
1. DNA文库构建及其关键酶
2. RNA文库构建及其关键酶
3. DNA & RNA 文库构建中NGS核心酶的使用指南
1. DNA文库构建及其关键酶
在DNA文库构建过程中,TA克隆连接接头文库构建是目前最常用的技术手段,其主要建库流程如下:

图2. DNA文库构建过程(Illumina)
1.1 DNA 碎片
目前测序仪的测序长度一般在150-500个碱基对(bp)范围内。因此,必须采用机械或酶促碎裂方法将较大的基因组DNA片段分解成较小的片段。机械碎裂会导致相对较高的样品损失,并且涉及更复杂的操作过程。另一方面,酶促消化是碎裂基因组DNA的常用方法。与机械方法相比,酶促消化更具成本效益且更简单,反应只需在加入碎裂酶后一段时间即可完成。
目前主要使用两种类型的片段。一种依赖于基于转座子原理的 Tn5 转座酶,另一种利用内切酶混合物。然而,这些片段的有效性可能受到 DNA 的 GC 含量和碱基偏好的影响。相比之下,由
1.2 末端修复,dA-Tailing
片段化的DNA会产生5'/3'粘性末端及平端DNA,所有粘性末端均需要转化为平端,包括去除3'突出端和填补5'突出端的DNA末端。使用TA连接进行接头连接时,DNA片段也需要在5'端进行磷酸化,并在3'端添加“A”以与带有“T”粘性末端的接头互补。上述过程是在T4 DNA聚合酶、T4多核苷酸激酶和 塔克 DNA 聚合酶。
T4 DNA 聚合酶(目录号:12901) 具有5'→3'DNA聚合酶活性,可催化DNA沿5'→3'方向合成,并填充5'突出端;同时该酶还具有3'→5'外切酶活性,可裂解3'突出端,从而将含有粘性末端的DNA片段转化为平端DNA。
由于合成的PCR引物和接头的5’端通常是羟基而不是磷酸基,因此需要T4多核苷酸激酶(Cat#12902)在ATP存在下催化ATPγ-磷酸基转移到寡核苷酸链的5’-羟基端,为下一步接头连接做准备。
硫脲 DNA聚合酶 (Cat#13486)具有5'→3'聚合酶活性,可以从5'→3'方向合成DNA,同时具有脱氧核苷酸转移酶活性,可以在PCR产物的3'端添加一个核苷酸“A”。

图3. 多种酶参与末端修复过程
图4. 毛细管电泳检测发现,S-taq能非常高效地在基因片段3’端ATCG的四个碱基上添加A。
1.3 接头连接
Adapter是文库的重要组成部分,在Illumina测序领域,常用的Y型Adapter有P5/P7、Index、Rd1/Rd2 SP序列。其中P5/P7序列的作用是与测序芯片上的序列配对,从而将待分析的片段锚定到Flow Cell上进行桥式扩增;Index序列用于区分混合文库中待测序的不同样本;Rd1/Rd2 SP表示Read1和Read2测序引物的结合区域。
对于接头连接任务, T4 DNA 连接酶(目录号#12996) 是标准选择。它具有修复双链 DNA 中发现的单链缺口并重新连接相邻核苷酸的能力。
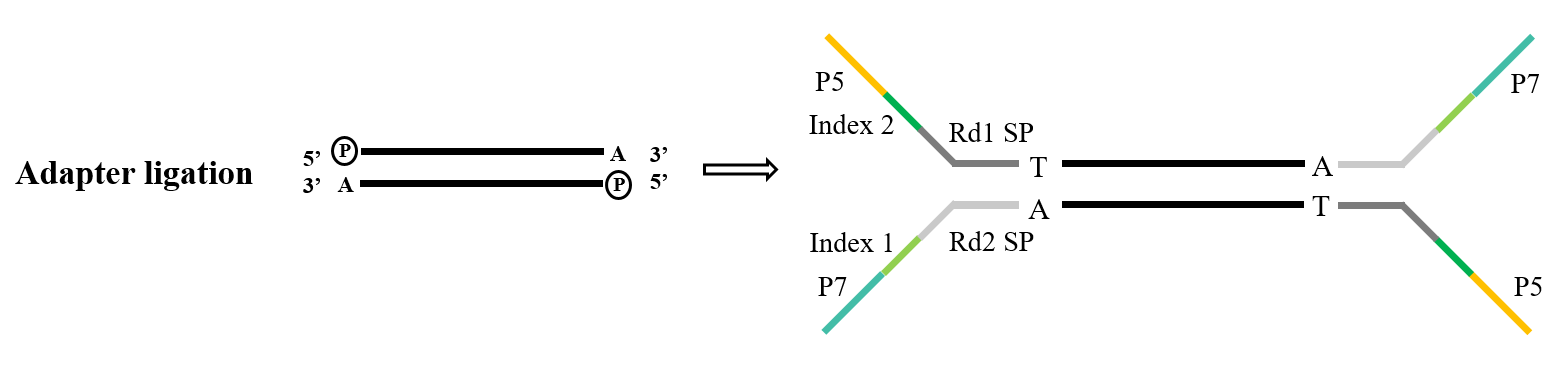
图5. 通用接头连接流程(Illumina)

图 6. 通过将 170-bp DNA 与 80-bp 接头连接来验证 T4 DNA 连接酶突变体。
1.4 PCR扩增
通过PCR反应获得足够数量的带有接头的DNA序列,在机上完成样本核酸序列的测序。Hieff Canace商标 PCR常用的Pro High-Fidelity DNA Polymerase(Cat#13476)具有5'→3'聚合酶活性,可按5'→3'方向合成DNA;同时还具有3'→5'外切酶活性,可纠正扩增过程中碱基的错误掺入,从而快速、高保真地扩增DNA片段。
2. RNA文库构建及其关键酶
根据RNA的种类,RNA文库的构建可分为mRNA文库、LncRNA文库等。常规的RNA文库构建包括以下几个过程:
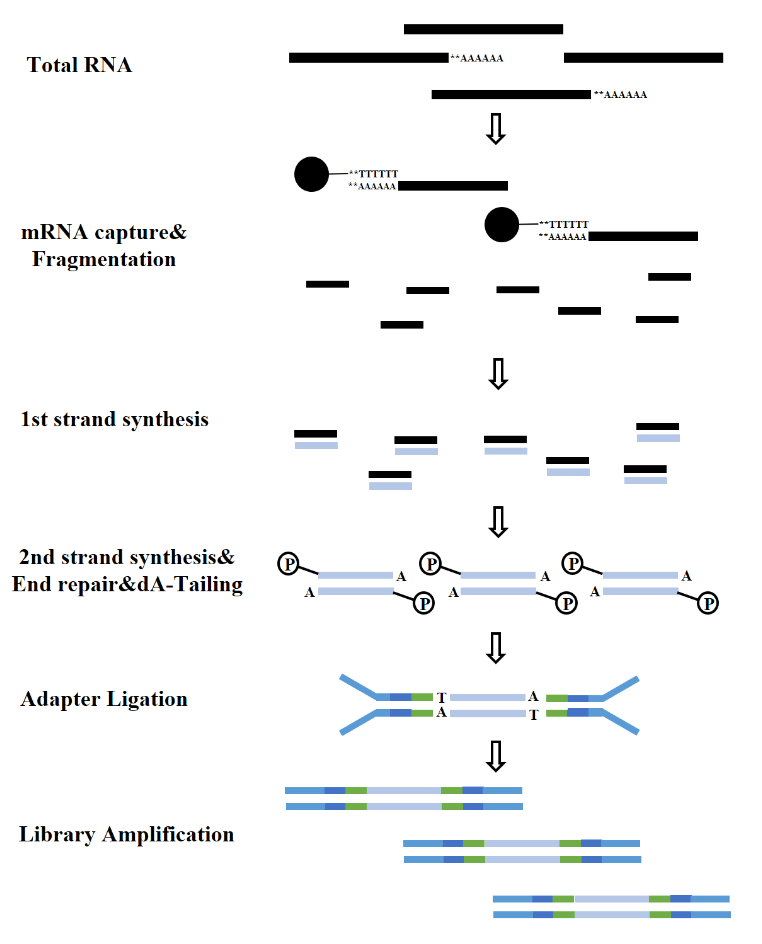
图7. mRNA文库构建流程(Illumina)
2.1 RNA富集
无论是真核生物还是原核生物,核糖体 RNA (rRNA) 都是最丰富的 RNA,占总 RNA 含量的 80%。直接对样本的总 RNA 进行测序时,很大一部分测序数据将与 rRNA 相关。为了减轻这种干扰,必须采用 RNA 富集方法。主要有两种方法:基于寡聚 dT 的 mRNA 富集和 rRNA 消耗方法。
在真核生物中,mRNA 在 3' 端表现出明显的 poly(A) 结构。Oligo-dT 珠可用于捕获从样本中转录的所有 mRNA,使其适合于转录分析,尤其是对于高质量的 RNA 样本。另一方面,rRNA 消耗方法对样本质量的要求更宽松,既可以应用于低质量样本(例如 FFPE 样本),也可以应用于高质量的 RNA 样本,以及原核生物样本。常用的商业方法是使用 RNase H 消化去除 rRNA,具体步骤如下:
- 合成旨在与 rRNA 结合的特定寡核苷酸探针。
- 使用能够降解 RNA-DNA 混合链中的 RNA 的 RNase H(Cat#12906)来选择性地去除与探针结合的 rRNA。
- 最后,用 DNase I(目录号 10325), 它可以降解单链和双链 DNA,有效消除 rRNA。有关 DNase I 的更多信息,您可以关注此链接。
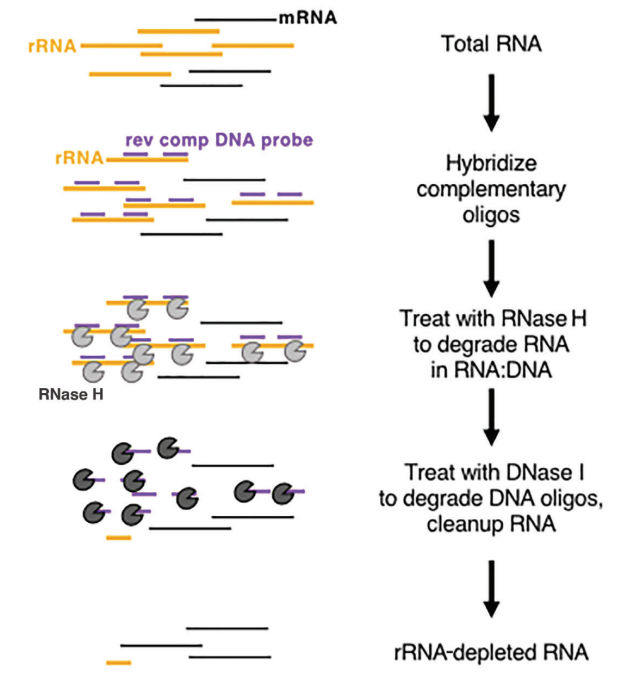
图8:基于酶的rRNA消耗示意图[5]
2.2 RNA碎片
通常在二价金属阳离子和高温的作用下,RNA的大片段会断裂成小片段。
2.3 第一链 cDNA 合成
将所得靶RNA逆转录成cDNA的第一链。由于RNA很容易被环境中的RNases降解,因此使用 RNase 抑制剂(Cat#14672) 在逆转录过程中,可以抑制这些酶的活性,保护 RNA 免受 RNase 降解。同时, 逆转录酶(目录号#11112) 用于将模板RNA逆转录成cDNA。逆转录酶具有RNA依赖性的DNA聚合酶活性,可以以RNA为模板,以5'→3'方向合成cDNA。DNA单链与RNA模板互补。
在第一 链 cDNA 合成中,放线菌素 D 的加入无疑改善了链特异性文库的构建,显著提高了链特异性。这一创新简化了实验过程,为研究人员简化了实验过程。
然而,放线菌素 D 也有缺点:它具有毒性,需要避光。在当今对预混和板库构建试剂盒的需求不断增长的背景下,避光的必要性限制了板库试剂盒的进步。
幸运的是,

图 9: 对 MMLV 进行工程改造,以识别可能有助于标准 RNA 测序的 MMLV 突变体
2.4 第二链 cDNA 合成
通过逆转录产生的单链 cDNA 非常不稳定,需要在 DNA 聚合酶 I 的影响下立即合成 cDNA 的第二链。在第二链合成过程中,RNase H 发挥作用,从 RNA-DNA 杂交结构中去除 RNA 链。它与 DNA聚合酶I(目录号12903) 以促进互补的第二条 cDNA 链的催化合成。DNA 聚合酶 I 具有 5'→3' DNA 聚合酶活性,在模板和引物的引导下,合成一条以 5'→3' 方向与单链 cDNA 互补的序列。
后续步骤包括末端修复、dA-Tailing、接头连接、PCR扩增等,这些步骤在DNA文库构建步骤中有详细说明,在此不再赘述。值得注意的是,逆转录完成后,无需对核酸片段进行进一步的片段化。
3. DNA & RNA 文库构建中NGS核心酶的使用指南
表 1.DNA 和 RNA 文库构建中 NGS 核心酶指南
| 类型 | 产品定位 | 产品名称 | 猫# |
| RNA文库 建造 | 核糖体RNA 消耗/第二链 cDNA 合成 | 12906ES | |
| 核糖体RNA 消耗 | 10325ES | ||
| 第一链 cDNA 合成 | 14672ES | ||
| 11112ES | |||
| 第二链 cDNA 合成 | 12903ES | ||
| RNA文库 建造 & DNA 图书馆 建造 | 结束修复 | 12901ES | |
| 12902ES | |||
| dA 尾矿 | 13486ES | ||
| 接头连接 | 10301ES | ||
| 聚合酶链反应 放大 | 12621ES |
表2.DNA 和 RNA 文库 准备工具包
| 姓名 | 猫# | 笔记 | |
| 脱氧核糖核酸 | Hieff NGS DNA 文库制备试剂盒 | 13577ES | 肿瘤/机械方法 |
| Hieff NGS OnePot Pro DNA 文库制备试剂盒 V2 | 12194ES | 肿瘤/酶法 | |
| Hieff NGS OnePot Illumina II DNA 文库制备试剂盒 | 13490ES | 病原体/酶/常规时间(140分钟) | |
| Hieff NGS OnePot Flash DNA 文库制备试剂盒 | 12316ES | 病原体/ 酶/ 超快 (100分钟) | |
| Hieff NGS DNA&RNA 文库共制备试剂盒 V2 | 12305ES | 病原体/酶/DNA 和 RNA 共制备 | |
| 核糖核酸 | Hieff NGS Ultima 双模式 mRNA 文库制备试剂盒 | 12308ES | 不含寡核苷酸 dT 磁珠,11 管 |
| Hieff NGS Ultima 双模式 mRNA 文库制备试剂盒 | 12309ES | oligo dT 磁珠 plus,14 管 | |
| Hieff NGS® Ultima 双模式 RNA 文库制备试剂盒 | 12310ES | 预混版,5 管 | |
| Hieff NGS ® EvoMax RNA Library Prep Kit(预混版)(放线菌素D 自由的) | 12340ES | 预混版本, (放线菌素 D 自由的) | |
| Hieff NGS® MaxUp rRNA 耗竭试剂盒(植物) | 12254ES | 植物 | |
| Hieff NGS® MaxUp 人类 rRNA 耗竭试剂盒(rRNA 和 ITS/ETS) | 12257ES | 人类 |
参考:
[1] Mardis, Elaine R. 新一代测序平台[J]. 分析化学年鉴, 2013, 6(1):287-303。
[2] Gulilat M, Lamb T, Teft WA 等。靶向下一代测序作为切除医学的工具[J]。BMC Medical Genomics,2019,12(1):81。
[3] Lundberg KS, Dan DS, Adams M 等. 利用从Pyrococcus furiosus分离的耐热DNA聚合酶进行高保真扩增[J]. 基因, 1991, 108(1):1-6。
[4] Miyazaki K. 用内切酶V进行随机DNA片段化:在DNA改组中的应用[J]. 核酸研究,2002,30(24):e139。
[5] Baldwin A, Morris AR, Mukherjee N. 一种用于 RNA 测序的简单、经济有效且可扩展的消耗人类核糖体 RNA 的方法 [J]。Current Protocols, 2021, 1(6):e176。

