高通量定序,通常稱為下一代定序 (NGS) 技術,代表了從最初的 DNA 定序方法(例如桑格定序)向前邁出的重大飛躍。 NGS可以同時分析數十萬甚至數百萬個核酸分子序列。其優點包括卓越的通量、成本效益、可擴展性和廣泛的應用範圍,使其成為全球主要的定序技術。
NGS 定序工作流程包括四個主要階段:樣本準備、文庫建構、定序和資料分析。文庫建構的核心是將標準化的NGS平台接頭序列附著到片段化的基因組DNA的兩端。此步驟旨在產生充足的文庫核酸分子,準備透過 PCR 擴增在 NGS 儀器上進行定序。根據樣本性質,NGS文庫建構可分為DNA文庫建構和RNA文庫建構。酶在這些相互關聯的實驗中起著關鍵作用。那麼,文庫建構過程中有哪些關鍵酵素參與呢?
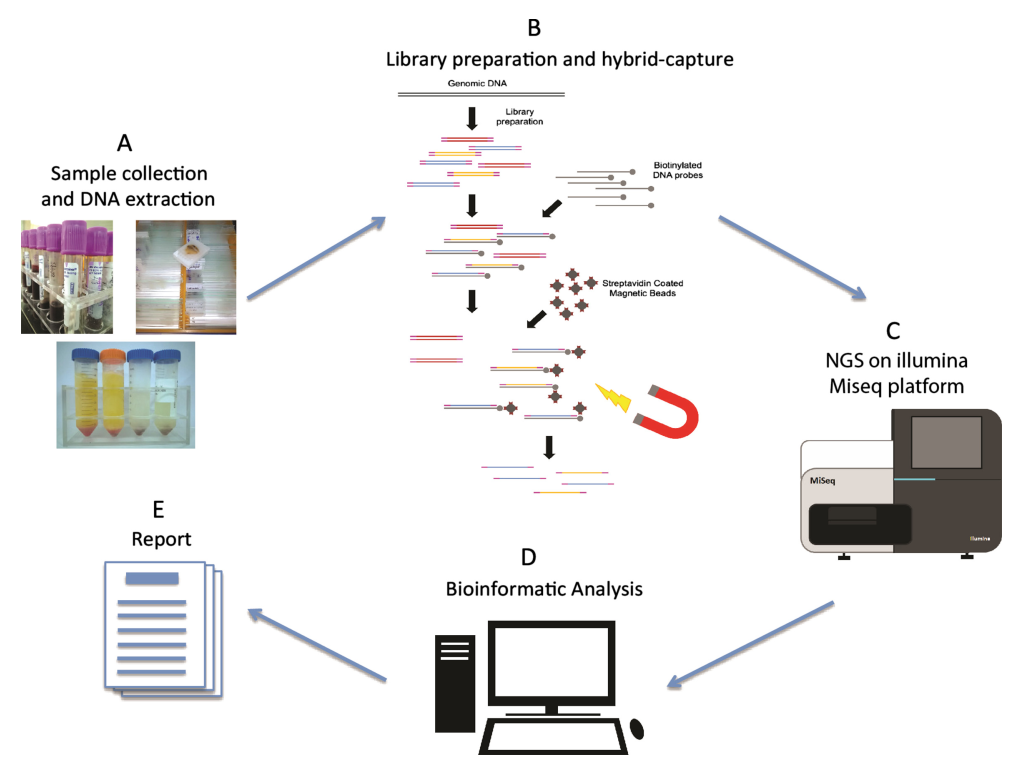
圖 1. 新一代定序工作流程[2]
1. DNA文庫建構及其關鍵酶
2. RNA文庫建構及其關鍵酶
3. DNA & RNA 文庫建構中NGS核心酵素的使用指南
1. DNA文庫建構及其關鍵酶
在DNA文庫建構過程中,TA克隆連接接頭文庫建構是目前最常用的技術手段。主要建庫流程如下:

圖2. DNA文庫建構過程(Illumina)
1.1 DNA 碎片
目前的定序儀的定序長度通常在150-500個鹼基對(bp)的範圍內。因此,有必要採用機械或酵素碎片化方法將較大的基因組 DNA 片段分解成較小的片段。機械破碎會導致相對較高的樣品損失,並涉及更複雜的操作過程。另一方面,酵素消化是片段化基因組 DNA 的常用方法。與機械方法相比,酵素消化更經濟、更簡單,加入碎裂酶後僅需一段時間即可完成反應。
目前,主要使用的碎片有兩種類型。一種依賴基於轉座子原理的 Tn5 轉座酶,而另一種則利用核酸內切酶的混合物。然而,這些片段的有效性會受到 DNA 的 GC 含量和鹼基偏好的影響。相比之下,
1.2 端修復,dA-Tailing
片段化的DNA會產生5'/3'粘端及平端DNA,所有粘端均需要轉化為平端,包括去除3'突出端和填補5'突出端的DNA末端。上述過程是在T4 DNA聚合酶、T4多核苷酸激酶和 塔克 DNA 聚合酶。
T4 DNA 聚合酶(目錄編號:12901) 具有5'→3'DNA聚合酶活性,能催化DNA沿5'→3'方向的合成,並填充5'突出端。同時,此酵素還具有3'→5'外切酶活性,可切割3'懸垂末端,從而將含有黏性末端的DNA片段轉化為平端DNA。
由於合成的PCR引子和接頭的5'端通常是羥基而不是磷酸基。因此,需要T4多核苷酸激酶(Cat#12902)在ATP存在下催化ATPγ-磷酸基轉移到寡核苷酸鏈的5'-羥基端,為下一步的接頭連接做準備。
硫脲 DNA聚合酶 (Cat#13486)具有5'→3'聚合酶活性,可以從5'→3'方向合成DNA。同時,它具有脫氧核苷酸轉移酶活性,可以在PCR產物的3’端添加一個核苷酸「A」。

圖3. 多種酵素參與末端修復過程
圖4. 毛細管電泳檢測發現,S-taq能非常有效率地在基因片段3’端ATCG的四個鹼基上添加A。
1.3 接頭連接
適配器是該庫的重要組成部分。在Illumina定序領域,常用的Y型接頭包括P5/P7、Index、Rd1/Rd2 SP序列。其中P5/P7序列的作用是與定序晶片上的序列配對,從而將需要分析的片段錨定到流動槽上,進行橋式擴增。 Index序列用於區分混合文庫中需要定序的不同樣本,Rd1/Rd2 SP表示Read1和Read2定序引子的結合區域。
對於接頭連接任務, T4 DNA 連接酶(目錄編號#12996) 是標準選擇。它具有修復雙股 DNA 中單股切口和重新連接相鄰核苷酸的能力。
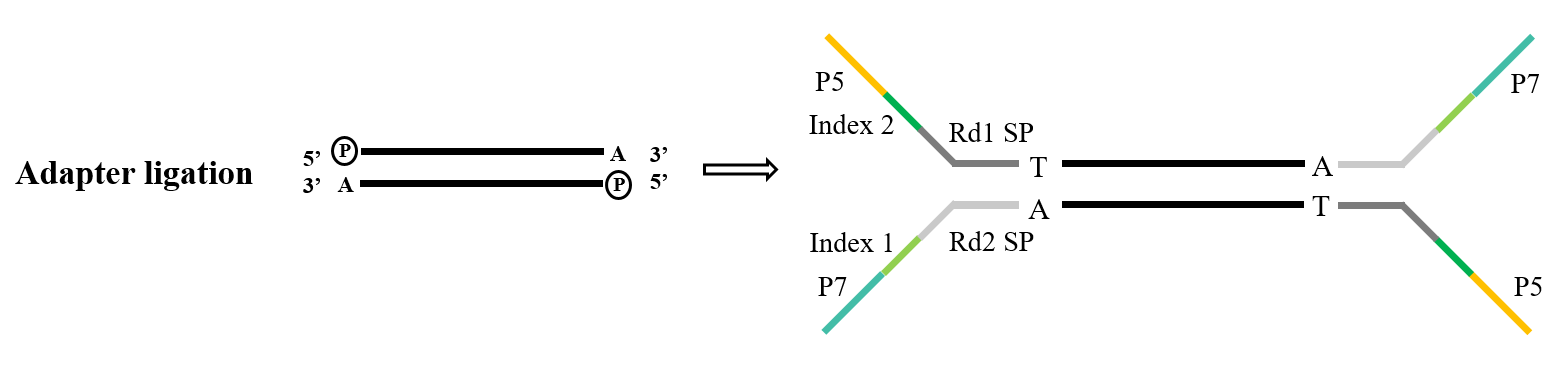
圖5. 通用接頭連接流程(Illumina)

圖 6. 將 170-bp DNA 與 80-bp 接頭連接來驗證 T4 DNA 連接酶突變體。
1.4 PCR擴增
透過PCR反應獲得足夠的帶有接頭的DNA序列,在機器上完成樣本核酸序列的定序。希夫·卡納斯商標 PCR中常用的Pro High-Fidelity DNA Polymerase(Cat#13476)具有5'→3'聚合酶活性,可依5'→3'方向合成DNA。此外其還具有3'→5'外切酶活性,能夠糾正擴增過程中鹼基的錯誤摻入,從而快速、高保真地擴增DNA片段。
2. RNA文庫建構及其關鍵酶
根據RNA的種類,RNA文庫的建構可分為mRNA文庫、LncRNA文庫等。
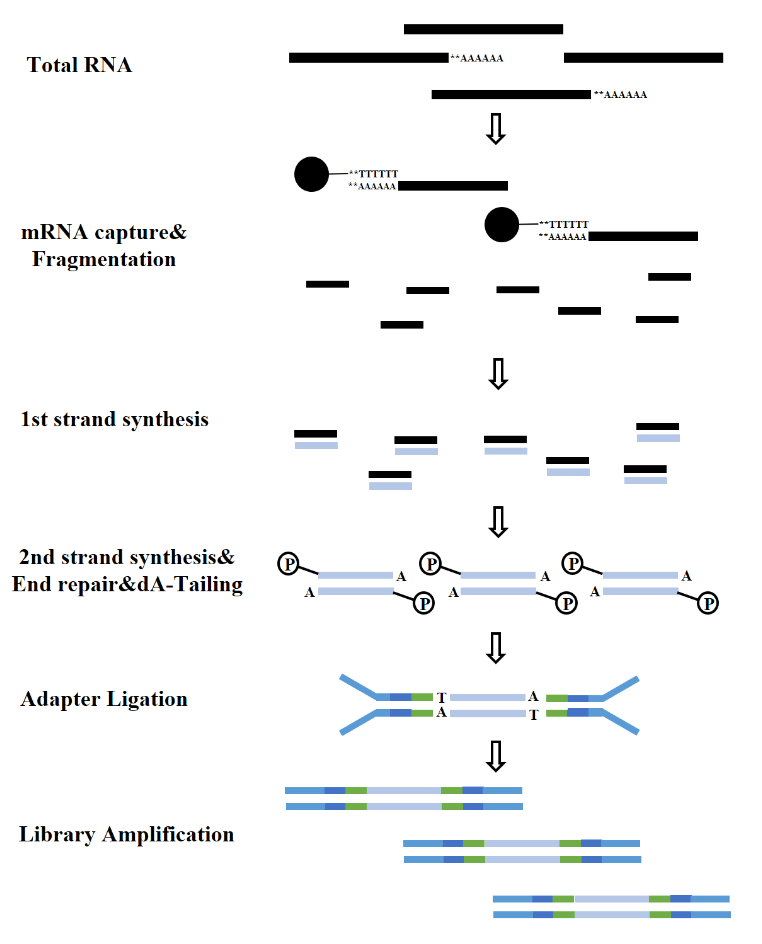
圖7. mRNA文庫建構流程(Illumina)
2.1 RNA富集
無論是真核生物或原核生物,核醣體RNA(rRNA)都是最豐富的RNA,佔總RNA含量的80%。直接對樣本的總 RNA 進行定序時,相當一部分定序資料將與 rRNA 相關。為了減輕這種幹擾,必須採用 RNA 富集的方法。主要有兩種方法:基於寡聚dT的mRNA富集方法和rRNA消耗方法。
在真核生物中,mRNA 在 3' 端表現出獨特的 poly(A) 結構。 Oligo-dT 珠可用於捕獲從樣本中轉錄的所有 mRNA,使其適用於轉錄分析,尤其是高品質的 RNA 樣本。另一方面,rRNA消耗方法對樣本品質的要求更為寬鬆,既可以應用於低品質樣本(例如FFPE樣本),也可以應用於高品質的RNA樣本,以及原核生物樣本。常用的商業方法是使用 RNase H 消化去除 rRNA,具體步驟如下:
- 合成旨在與 rRNA 結合的特定寡核苷酸探針。
- 使用能夠降解 RNA-DNA 混合鏈中的 RNA 的 RNase H(Cat#12906)來選擇性地去除與探針結合的 rRNA。
- 最後,用 DNase I(目錄編號 10325), 它可以降解單鍊和雙股DNA,有效消除rRNA。有關 DNase I 的更多信息,您可以點擊此連結。
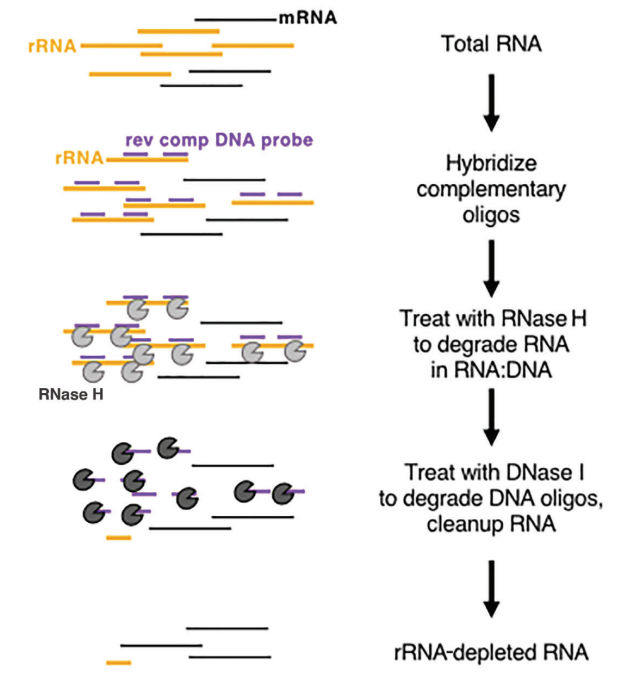
圖8:基於酵素的rRNA消耗示意圖[5]
2.2 RNA碎片
通常在二價金屬陽離子和高溫的作用下,RNA的大片段會斷裂成小片段。
2.3 第一鏈 cDNA 合成
將所得的目標RNA逆轉錄為cDNA第一鏈。由於 RNA 容易被環境中的 RNases 降解,因此使用 RNase 抑制劑(Cat#14672) 在逆轉錄過程中可以抑制這些酵素的活性,保護RNA免受RNase降解。同時, 逆轉錄酶(目錄號#11112) 用於將模板RNA逆轉錄為cDNA。逆轉錄酶具有RNA依賴性的DNA聚合酶活性,可以以RNA為模板,以5'→3'方向合成cDNA。 DNA單股與RNA模板互補。
在第一 鏈 cDNA 合成中,放線菌素 D 的加入無疑改善了鏈特異性文庫的構建,顯著提高了鏈特異性。這項創新簡化了實驗流程,為研究人員簡化了實驗過程。
然而,放線菌素 D 也有其缺點:它具有毒性並且需要避光。在當今對預混和板庫建構套件的需求不斷增加的背景下,避光的必要性對板套件的進步造成了限制。
幸運的是,

圖 9: 對 MMLV 進行工程改造,以識別可能有助於標準 RNA 定序的 MMLV 突變體
2.4 第二鏈 cDNA 合成
透過逆轉錄產生的單鏈 cDNA 極不穩定,需要在 DNA 聚合酶 I 的影響下立即合成 cDNA 的第二鏈。它與 DNA聚合酶I(目錄號12903) 促進互補的第二條 cDNA 鏈的催化合成。 DNA聚合酶I具有5'→3'DNA聚合酶活性,在模板和引子的引導下,合成與5'→3'方向的單股cDNA互補的序列。
該過程的後續步驟包括末端修復、dA-Tailing、接頭連接和 PCR 擴增,所有這些步驟均在 DNA 文庫建構程序中詳細說明,在此無需重申。值得注意的是,一旦逆轉錄完成,就不需要進一步分解核酸片段。
3. DNA & RNA 文庫建構中NGS核心酵素的使用指南
表 1.DNA 和 RNA 文庫建構 NGS 核心酵素指南
| 類型 | 產品定位 | 產品名稱 | 貓# |
| RNA文庫 建造 | 核醣體RNA 消耗/第二鏈 cDNA 合成 | 12906ES | |
| 核醣體RNA 消耗 | 10325ES | ||
| 第一鏈 cDNA 合成 | 14672ES | ||
| 11112ES | |||
| 第二鏈 cDNA 合成 | 12903ES | ||
| RNA文庫 建造 & DNA 圖書館 建造 | 結束脩復 | 12901ES | |
| 12902ES | |||
| dA 尾礦 | 13486ES | ||
| 接頭連接 | 10301ES | ||
| 聚合酶連鎖反應 放大 | 12621ES |
表2.DNA 和 RNA 文庫 準備工具包
| 姓名 | 貓# | 筆記 | |
| 脫氧核糖核酸 | Hieff NGS DNA 文庫製備試劑盒 | 13577ES | 腫瘤/機械方法 |
| Hieff NGS OnePot Pro DNA 文庫製備試劑盒 V2 | 12194ES | 腫瘤/酵素法 | |
| Hieff NGS OnePot Illumina II DNA 文庫製備試劑盒 | 13490ES | 病原體/酵素/常規時間(140分鐘) | |
| Hieff NGS OnePot Flash DNA 文庫製備試劑盒 | 12316ES | 病原體/ 酵素/ 超快 (100分鐘) | |
| Hieff NGS DNA&RNA 文庫共製備試劑盒 V2 | 12305ES | 病原體/酵素/DNA 和 RNA 共製備 | |
| 核糖核酸 | Hieff NGS Ultima 雙模式 mRNA 文庫製備試劑盒 | 12308ES | 不含寡核苷酸 dT 磁珠,11 管 |
| Hieff NGS Ultima 雙模式 mRNA 文庫製備試劑盒 | 12309ES | oligo dT 磁珠 plus,14 管 | |
| Hieff NGS® Ultima 雙模式 RNA 文庫製備試劑盒 | 12310ES | 預混版,5 管 | |
| Hieff NGS ® EvoMax RNA Library Prep Kit(預混版)(放線菌素D 自由的) | 12340ES | 預混版本, (放線菌素 D 自由的) | |
| Hieff NGS® MaxUp rRNA 耗竭試劑盒(植物) | 12254ES | 植物 | |
| Hieff NGS® MaxUp 人類 rRNA 耗竭試劑盒(rRNA 和 ITS/ETS) | 12257ES | 人類 |
參考:
[1] Mardis,Elaine R.次世代定序平台[J]。分析化學年鑑,2013,6(1):287-303。
[2] Gulilat M、Lamb T、Teft WA 等人。靶向下一代定序作為切除醫學的工具[J]。 BMC Medical Genomics,2019,12(1):81。
[3] Lundberg KS、Dan DS、Adams M 等人。利用從激烈火球菌中分離的耐熱 DNA 聚合酶進行高保真擴增[J]。基因,1991,108(1):1-6。
[4] Miyazaki K. 利用內切酶V進行隨機DNA片段化:在DNA重組的應用[J]。核酸研究,2002,30(24):e139。
[5] Baldwin A、Morris AR、Mukherjee N. 一種用於 RNA 定序的簡單、經濟有效且可擴展的消耗人類核醣體 RNA 的方法[J]。當前協議,2021,1(6):e176。

