Le séquençage à haut débit, souvent appelé technologie de séquençage de nouvelle génération (NGS), représente une avancée significative par rapport aux méthodes initiales de séquençage de l'ADN, telles que le séquençage Sanger. Le NGS permet de profiler simultanément des centaines de milliers, voire des millions, de séquences de molécules d'acide nucléique. Ses mérites comprennent un débit exceptionnel, une rentabilité, une évolutivité et un large spectre d'applications, ce qui en fait la technologie de séquençage prédominante dans le monde.
Le flux de travail de séquençage NGS comprend quatre phases principales : la préparation des échantillons, la construction de la bibliothèque, le séquençage et l'analyse des données. Au cœur de la construction de la bibliothèque se trouve la fixation de séquences d'adaptateur de plateforme NGS standardisées aux deux extrémités de l'ADN génomique fragmenté. Cette étape vise à générer une réserve suffisante de molécules d'acide nucléique de la bibliothèque, préparées pour le séquençage sur l'instrument NGS par amplification PCR. Selon la nature de l'échantillon, la construction de la bibliothèque NGS peut être classée en construction de bibliothèque d'ADN et construction de bibliothèque d'ARN. Les enzymes jouent un rôle essentiel dans ces expériences interconnectées. Alors, quelles enzymes clés sont impliquées dans le processus de construction de la bibliothèque ?
Figure 1. Flux de travail de séquençage de nouvelle génération[2]
1. Construction d'une bibliothèque d'ADN et de ses enzymes clés
2. Construction d'une bibliothèque d'ARN et ses enzymes clés
3. Directives pour les enzymes de base NGS dans la construction de bibliothèques d'ADN et d'ARN
1. Construction d'une bibliothèque d'ADN et de ses enzymes clés
Dans le processus de construction d'une bibliothèque d'ADN, la construction d'une bibliothèque d'adaptateurs de ligature de clones TA est le moyen technologique le plus couramment utilisé à l'heure actuelle. Le principal processus de construction de la bibliothèque est le suivant :

Figure 2. Processus de construction d'une bibliothèque d'ADN (Illumina)
1.1 Fragmentation de l'ADN
Les séquenceurs actuels ont généralement une longueur de séquençage comprise entre 150 et 500 paires de bases (pb). Par conséquent, il devient nécessaire d'utiliser des méthodes de fragmentation mécaniques ou enzymatiques pour décomposer les gros fragments d'ADN génomique en fragments plus petits. La fragmentation mécanique peut entraîner une perte d'échantillon relativement élevée et implique un processus opérationnel plus complexe. D'autre part, la digestion enzymatique est une méthode couramment utilisée pour fragmenter l'ADN génomique. Par rapport aux méthodes mécaniques, la digestion enzymatique est plus rentable et plus simple, la réaction ne nécessitant qu'une période définie après l'ajout de l'enzyme de fragmentation.
Actuellement, il existe principalement deux types de fragments utilisés. L'un repose sur la transposase Tn5, basée sur les principes du transposon, tandis que l'autre utilise un mélange d'endonucléases. Cependant, l'efficacité de ces fragments peut être influencée par la teneur en GC et les préférences de base de l'ADN. En revanche, les fragments développés par Yeasen (Cat#12917) offrent un effet de digestion stable et présentent une préférence de site nettement inférieure à celle de la transposase Tn5. Ils donnent systématiquement d'excellents résultats de séquençage pour divers types d'échantillons d'ADN, y compris ceux provenant d'échantillons FFPE.
1.2 Réparation d'extrémité, dA-Tailing
L'ADN fragmenté générera des extrémités collantes 5'/3' et de l'ADN à extrémités franches, et toutes les extrémités collantes doivent être converties en extrémités franches, y compris les surplombs 3' supprimés et les extrémités d'ADN saillantes 5' remplies. Lors de l'utilisation de la ligature TA pour la ligature de l'adaptateur, le fragment d'ADN doit également être phosphorylé à l'extrémité 5' et ajouter « A » à l'extrémité 3' pour être complémentaire de l'adaptateur avec l'extrémité collante « T ».Le processus ci-dessus est complété par la coopération de l'ADN polymérase T4, de la polynucléotide kinase T4 et Taq ADN polymérase.
ADN polymérase T4 (Cat#12901) L'enzyme possède une activité d'ADN polymérase 5'→3', qui peut catalyser la synthèse d'ADN dans le sens 5'→3' et combler l'extrémité saillante 5'. En même temps, l'enzyme possède également une activité exonucléase 3'→5' pour cliver les extrémités 3' en surplomb, transformant ainsi les fragments d'ADN contenant des extrémités collantes en ADN à extrémités franches.
Étant donné que les extrémités 5' des amorces et adaptateurs PCR synthétiques sont généralement des groupes hydroxyles au lieu de groupes phosphates, la polynucléotide kinase T4 (Cat#12902) est nécessaire pour catalyser le transfert des groupes ATP γ-phosphates à l'extrémité 5'-hydroxyle de la chaîne oligonucléotidique en présence d'ATP, en préparation de l'étape suivante de la ligature de l'adaptateur.
S-Taq ADN polymérase (Cat#13486) possède une activité polymérase 5'→3', qui lui permet de synthétiser l'ADN dans la direction 5'→3'. Parallèlement, il possède une activité de transférase de désoxynucléotidyle, qui peut ajouter un nucléotide « A » à l'extrémité 3' du produit PCR.

Figure 3. Plusieurs enzymes sont impliquées dans le processus de réparation terminale
Figure 4. S-taq a une très grande efficacité pour ajouter A aux quatre bases de l'ATCG de l'extrémité 3' des segments de gènes détectés par électrophorèse capillaire.
1.3 Ligature de l'adaptateur
Les adaptateurs constituent un élément essentiel de la bibliothèque. Dans le domaine du séquençage Illumina, les adaptateurs de type Y couramment utilisés englobent les séquences P5/P7, Index et Rd1/Rd2 SP. Parmi celles-ci, la séquence P5/P7 sert à s'associer à la séquence présente sur la puce de séquençage, ancrant ainsi les fragments à analyser sur la cellule d'écoulement pour exécuter l'amplification en pont. La séquence Index est utilisée pour distinguer les différents échantillons au sein de la bibliothèque mixte soumise au séquençage, tandis que Rd1/Rd2 SP désigne les régions de liaison des amorces de séquençage Read1 et Read2.
Pour la tâche de ligature de l'adaptateur, ADN ligase T4 (Cat#12996) est le choix standard. Il présente la capacité de réparer les entailles monocaténaires présentes dans l'ADN double brin et de reconnecter les nucléotides adjacents.
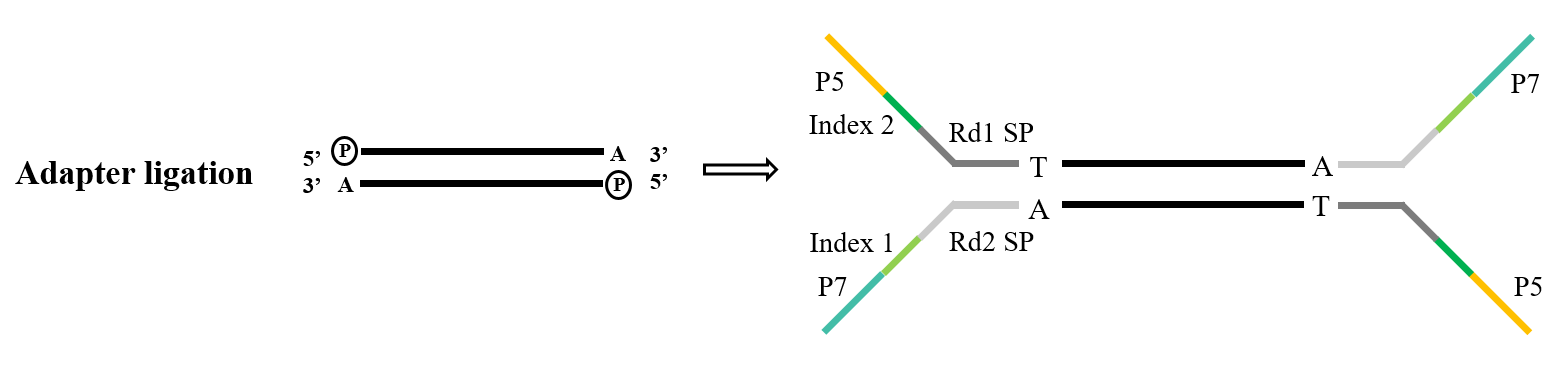
Figure 5. Processus général de ligature de l'adaptateur (Illumina)

Figure 6. Vérification des mutants de la ligase ADN T4 en ligaturant l'ADN de 170 pb avec des adaptateurs de 80 pb.
1.4 Amplification par PCR
Obtenez suffisamment de séquences d'ADN avec des adaptateurs grâce à la réaction PCR et terminez le séquençage de la séquence d'acide nucléique de l'échantillon sur la machine. Hieff CanaceMT L'ADN polymérase haute fidélité Pro (Cat#13476) couramment utilisée en PCR possède une activité polymérase 5'→3' et peut synthétiser l'ADN dans le sens 5'→3'. De plus, elle possède également l'activité d'exonucléase 3'→5', qui peut corriger l'incorporation incorrecte de bases pendant le processus d'amplification, pour amplifier les fragments d'ADN rapidement et avec une grande fidélité.
2. Construction d'une bibliothèque d'ARN et ses enzymes clés
Selon les types d'ARN, la construction d'une bibliothèque d'ARN peut être divisée en bibliothèque d'ARNm, bibliothèque d'ARNnc, etc. Une bibliothèque d'ARN conventionnelle comprend les processus suivants :
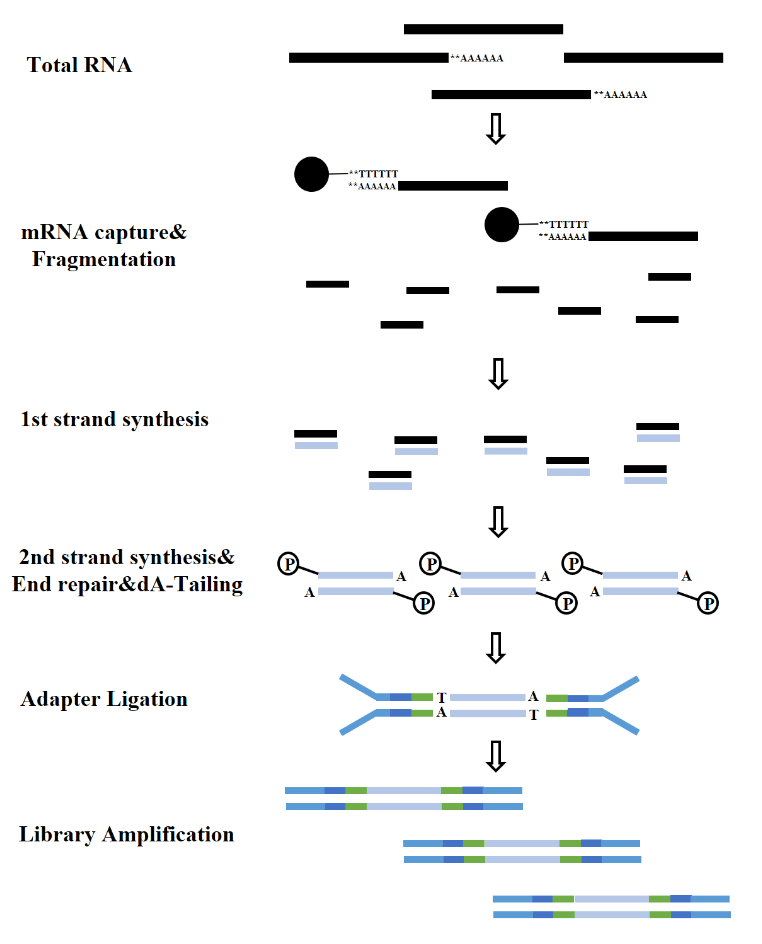
Figure 7. Processus de construction d'une bibliothèque d'ARNm (Illumina)
2.1 Enrichissement en ARN
Qu'il s'agisse d'eucaryotes ou de procaryotes, l'ARN ribosomique (ARNr) se distingue comme l'ARN le plus abondant, constituant jusqu'à 80 % du contenu total en ARN. Lors du séquençage direct de l'ARN total d'un échantillon, une partie substantielle des données de séquençage sera liée à l'ARNr. Pour atténuer cette interférence, la méthode d'enrichissement de l'ARN doit être utilisée. Il existe deux méthodes principales pour cela : l'enrichissement de l'ARNm basé sur l'oligo-dT et les méthodes d'appauvrissement de l'ARNr.
Chez les eucaryotes, l'ARNm présente une structure poly(A) distincte à l'extrémité 3'. Les billes d'oligo-dT peuvent être utilisées pour capturer tout l'ARNm transcrit à partir de l'échantillon, ce qui le rend adapté à l'analyse transcriptionnelle, en particulier avec des échantillons d'ARN de haute qualité. D'autre part, les méthodes d'épuisement de l'ARNr ont des exigences plus souples en matière de qualité de l'échantillon et peuvent être appliquées à la fois aux échantillons de faible qualité (par exemple, les échantillons FFPE) et aux échantillons d'ARN de haute qualité, ainsi qu'aux échantillons procaryotes. L'approche commerciale couramment utilisée implique l'utilisation de la digestion par RNase H pour éliminer l'ARNr, en suivant ces étapes spécifiques :
- Synthétiser des sondes oligonucléotidiques spécifiques conçues pour se lier à l'ARNr.
- Utilisez la RNase H (Cat#12906), capable de dégrader l'ARN dans le brin hybride ARN-ADN, pour éliminer sélectivement l'ARNr lié aux sondes.
- Enfin, digérez les sondes d’ADN avec DNase I (Cat#10325), qui peut dégrader l'ADN simple et double brin, éliminant ainsi efficacement l'ARNr. Pour plus d'informations sur la DNase I, vous pouvez suivre ce lien.
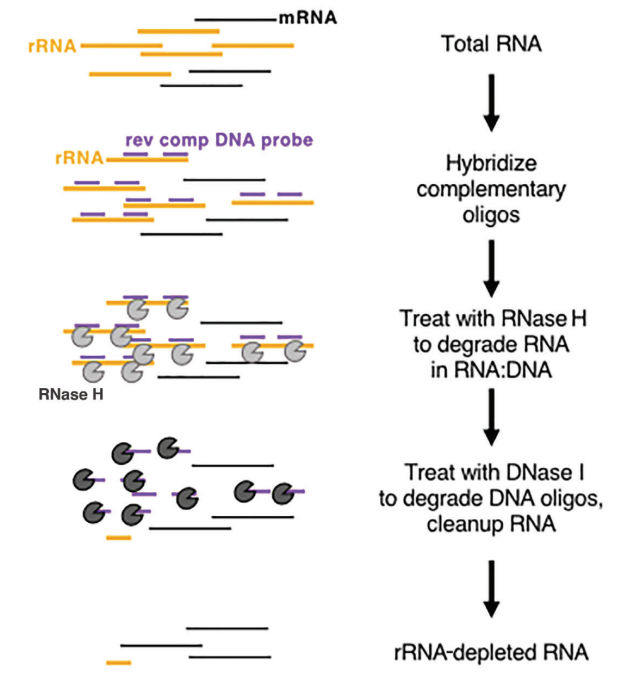
Figure 8 : Schéma de l'épuisement de l'ARNr par les enzymes[5]
2.2 Fragmentation de l'ARN
Habituellement, sous l'action des cations métalliques divalents et de la température élevée, les gros fragments d'ARN sont brisés en petits fragments.
2.3 Synthèse du premier brin d'ADNc
Transcription inverse de l'ARN cible obtenu dans le premier brin d'ADNc. L'ARN étant facilement dégradé par les ARNases présentes dans l'environnement, l'utilisation de Inhibiteur de RNase (Cat#14672) pendant la transcription inverse peut inhiber l'activité de ces enzymes et protéger l'ARN de la dégradation par la RNase. Dans le même temps, transcriptase inverse (Cat#11112) a été utilisée pour effectuer la transcription inverse de l'ARN modèle en ADNc. La transcriptase inverse a une activité d'ADN polymérase dépendante de l'ARN et peut utiliser l'ARN comme matrice pour synthétiser un ADNc dans la direction 5'→3'. Le brin simple d'ADN est complémentaire de la matrice d'ARN.
Au cours du 1er Dans la synthèse d'ADNc à brins multiples, l'incorporation d'actinomycine D a indéniablement amélioré la construction de bibliothèques spécifiques à chaque brin, améliorant considérablement la spécificité de la chaîne. Cette innovation a rationalisé le processus expérimental, le simplifiant pour les chercheurs.
Cependant, l'actinomycine D présente des inconvénients : elle est toxique et nécessite une protection contre la lumière. Dans le contexte actuel de demande croissante de kits de construction de bibliothèques pré-mélangées et de plaques, la nécessité de se protéger de la lumière limite les avancées des kits de plaques.
Heureusement, la plateforme Yeasen ZymeEditor a introduit un mutant révolutionnaire de l'enzyme MMLV (Inquiry) qui remplace la fonction de l'actinomycine D. Un nouveau Le kit (Cat : 12340ES) a été développé avec des ingrédients inodores, non toxiques et nj'ai besoin d'éviter Léger. Il offre une spécificité de chaîne supérieure, éliminant ainsi les problèmes liés à la santé et à la sensibilité à la lumière.

2.4 Synthèse du 2ème brin d'ADNc
L'ADNc monocaténaire produit par transcription inverse est très instable, ce qui nécessite la synthèse immédiate du second brin d'ADNc sous l'influence de l'ADN polymérase I. Au cours de cette synthèse du second brin, la RNase H entre en jeu en retirant le brin d'ARN de la structure hybride ARN-ADN. Elle fonctionne de concert avec ADN polymérase I (Cat#12903) pour faciliter la synthèse catalytique du deuxième brin complémentaire de l'ADNc. L'ADN polymérase I possède une activité ADN polymérase 5'→3' et, guidée par un modèle et une amorce, synthétise une séquence qui complète l'ADNc simple brin dans la direction 5'→3'.
Les étapes suivantes du processus comprennent la réparation des extrémités, la dA-Tailing, la ligature de l'adaptateur et l'amplification par PCR, qui sont toutes détaillées dans la procédure de construction de la bibliothèque d'ADN et n'ont pas besoin d'être réitérées ici. Il convient de noter qu'une fois la transcription inverse terminée, il n'est pas nécessaire de fragmenter davantage le fragment d'acide nucléique.
3. Directives pour les enzymes de base NGS dans la construction de bibliothèques d'ADN et d'ARN
Yeasen est une société de biotechnologie spécialisée dans la recherche, le développement, la production et la vente de trois principaux réactifs biologiques : les molécules, les protéines et les cellules. La société Yeasen Biotech produit une variété d'enzymes liées à la construction de bibliothèques NGS. Vous pouvez choisir le produit de construction de bibliothèque le plus adapté dans le tableau ci-dessous.
Tableau 1.Guide pour les enzymes de base NGS dans la construction de bibliothèques d'ADN et d'ARN
| Taper | Positionnement du produit | Nom du produit | Chat# |
| Bibliothèque d'ARN construction | ARNr épuisement/synthèse du 2e brin d'ADNc | 12906ES | |
| ARNr épuisement | 10325ES | ||
| Synthèse du 1er brin d'ADNc | 14672ES | ||
| 11112ES | |||
| Synthèse du 2ème brin d'ADNc | 12903ES | ||
| Bibliothèque d'ARN construction & ADN bibliothèque construction | Réparation de fin | 12901ES | |
| 12902ES | |||
| dA-Queues | 13486ES | ||
| Ligature de l'adaptateur | 10301ES | ||
| PCR amplification | 2×Super Canace® II Mix haute fidélité pour amplification de bibliothèque | 12621ES |
Tableau 2.Bibliothèque d'ADN et d'ARN Kit de préparation
| Nom | Chat# | Remarques | |
| ADN | Kit de préparation de bibliothèque d'ADN Hieff NGS | 13577ES | Méthode tumorale/mécanique |
| Kit de préparation de bibliothèque d'ADN Hieff NGS OnePot Pro V2 | 12194ES | Tumeur/Méthode enzymatique | |
| Système Hieff NGS OnePot Kit de préparation de bibliothèque d'ADN II pour Illumina | 13490ES | Pathgen/Enzymatique/temps régulier (140min) | |
| Kit de préparation de bibliothèque d'ADN Flash OnePot NGS Hieff | 12316ES | Pathgen/ Enzymatique/ Ultrarapide (100min) | |
| Kit de co-préparation de bibliothèque d'ADN et d'ARN Hieff NGS V2 | 12305ES | Pathgen/Co-préparation enzymatique/ADN et ARN | |
| ARN | Kit de préparation de bibliothèque d'ARNm bimode Hieff NGS Ultima | 12308ES | Sans billes magnétiques oligo dT, 11 tubes |
| Kit de préparation de bibliothèque d'ARNm bimode Hieff NGS Ultima | 12309ES | billes magnétiques oligo dT plus, 14 tubes | |
| Kit de préparation de bibliothèque d'ARN bimode Hieff NGS® Ultima | 12310ES | Version pré-mélangée, 5 tubes | |
| Kit de préparation de bibliothèque d'ARN Hieff NGS ® EvoMax (version prémélangée) (actinomycine D Gratuit) | 12340ES | Version pré-mélangée, (Actinomycine D Gratuit) | |
| Kit de déplétion d'ARNr Hieff NGS® MaxUp (plante) | 12254ES | Usine | |
| Kit de déplétion d'ARNr humain Hieff NGS® MaxUp (ARNr et ITS/ETS) | 12257ES | Humain |
Références :
[1] Mardis, Elaine R. Plateformes de séquençage de nouvelle génération[J]. Revue annuelle de chimie analytique, 2013, 6(1):287-303.
[2] Gulilat M, Lamb T, Teft WA, et al. Le séquençage ciblé de nouvelle génération comme outil de médecine de précision[J]. BMC Medical Genomics, 2019, 12(1):81.
[3] Lundberg KS, Dan DS, Adams M, et al. Amplification haute fidélité utilisant une ADN polymérase thermostable isolée de Pyrococcus furiosus[J]. Gene, 1991, 108(1):1-6.
[4] Miyazaki K. Fragmentation aléatoire de l'ADN avec l'endonucléase V : application au brassage de l'ADN[J]. Nucleic Acids Research, 2002, 30(24):e139.
[5] Baldwin A, Morris AR, Mukherjee N. Une méthode simple, rentable et évolutive pour épuiser l'ARN ribosomique humain pour l'ARN-seq[J]. Protocoles actuels, 2021, 1(6):e176.

