Giải trình tự thông lượng cao, thường được gọi là công nghệ giải trình tự thế hệ tiếp theo (NGS), là một bước tiến đáng kể so với các phương pháp giải trình tự DNA ban đầu, chẳng hạn như giải trình tự Sanger. NGS cho phép lập hồ sơ đồng thời hàng trăm nghìn, nếu không muốn nói là hàng triệu, trình tự phân tử axit nucleic. Ưu điểm của nó bao gồm thông lượng đặc biệt, hiệu quả về chi phí, khả năng mở rộng và phổ ứng dụng rộng, đưa nó trở thành công nghệ giải trình tự chiếm ưu thế trên toàn thế giới.
Quy trình giải trình tự NGS bao gồm bốn giai đoạn chính: chuẩn bị mẫu, xây dựng thư viện, giải trình tự và phân tích dữ liệu. Trọng tâm của việc xây dựng thư viện là gắn các trình tự bộ điều hợp nền tảng NGS chuẩn hóa vào cả hai đầu của DNA bộ gen bị phân mảnh. Bước này nhằm mục đích tạo ra nguồn cung cấp dồi dào các phân tử axit nucleic thư viện, được chuẩn bị để giải trình tự trên thiết bị NGS thông qua khuếch đại PCR. Tùy thuộc vào bản chất của mẫu, việc xây dựng thư viện NGS có thể được phân loại thành xây dựng thư viện DNA và xây dựng thư viện RNA. Các enzyme đóng vai trò quan trọng trong các thí nghiệm có sự kết nối này. Vậy, những enzyme chính nào tham gia vào quá trình xây dựng thư viện?
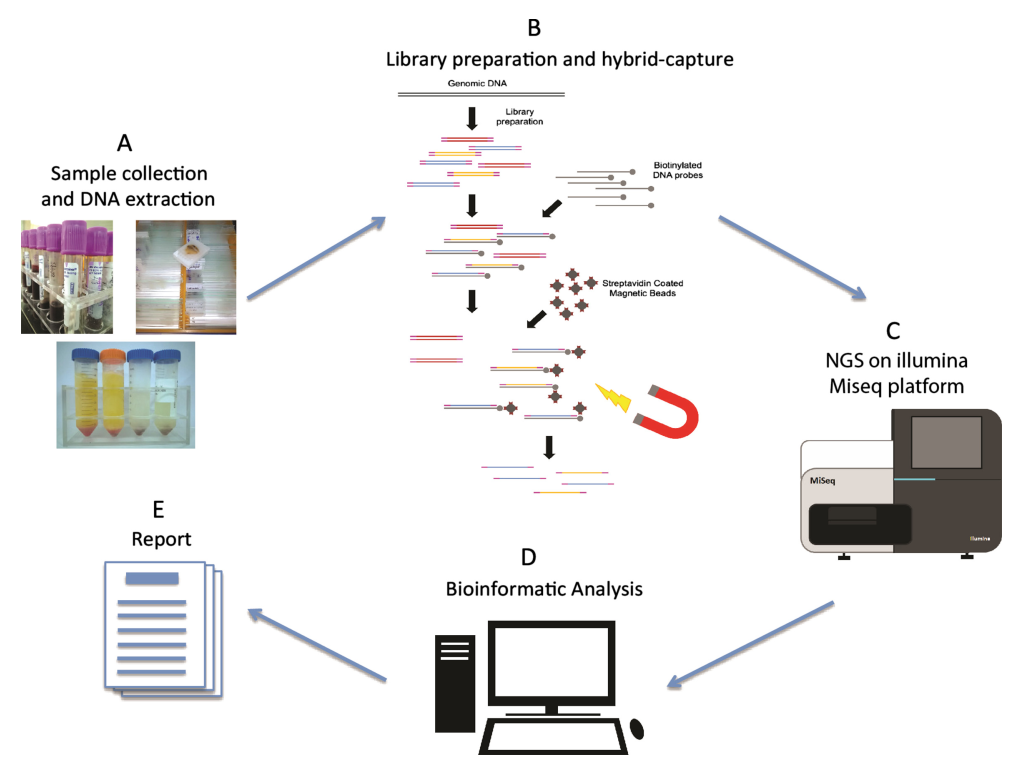
Hình 1. Quy trình giải trình tự thế hệ tiếp theo[2]
1. Xây dựng thư viện DNA và các enzyme chính của nó
2. Xây dựng thư viện RNA và các enzyme chính của nó
3. Hướng dẫn cho các enzyme cốt lõi NGS trong xây dựng thư viện DNA & RNA
1. Xây dựng thư viện DNA và các enzyme chính của nó
Trong quá trình xây dựng thư viện DNA, xây dựng thư viện bộ điều hợp nối bản sao TA là phương tiện công nghệ được sử dụng phổ biến nhất hiện nay. Quá trình xây dựng thư viện chính như sau:

Hình 2. Quá trình xây dựng thư viện DNA (Illumina)
1.1 Phân mảnh DNA
Các máy giải trình tự hiện tại thường có độ dài giải trình tự trong khoảng 150-500 cặp bazơ (bp). Do đó, cần phải sử dụng các phương pháp phân mảnh cơ học hoặc enzym để phá vỡ các đoạn DNA bộ gen lớn thành các đoạn nhỏ hơn. Phân mảnh cơ học có thể dẫn đến mất mẫu tương đối cao và liên quan đến một quy trình vận hành phức tạp hơn. Mặt khác, tiêu hóa bằng enzym là một phương pháp thường được sử dụng để phân mảnh DNA bộ gen. So với các phương pháp cơ học, tiêu hóa bằng enzym hiệu quả hơn về mặt chi phí và đơn giản hơn, với phản ứng chỉ cần một khoảng thời gian nhất định sau khi thêm enzym phân mảnh.
Hiện nay, chủ yếu có hai loại mảnh đang được sử dụng. Một loại dựa trên transposase Tn5, dựa trên các nguyên tắc transposon, trong khi loại còn lại sử dụng hỗn hợp các endonuclease. Tuy nhiên, hiệu quả của các mảnh này có thể bị ảnh hưởng bởi hàm lượng GC và sở thích cơ sở của DNA. Ngược lại, các mảnh được phát triển bởi
1.2 Sửa chữa cuối, dA-Tailing
DNA phân mảnh sẽ tạo ra đầu dính 5'/3' và DNA đầu tù, và tất cả các đầu dính cần được chuyển thành đầu tù, bao gồm cả phần nhô ra 3' được loại bỏ và đầu DNA nhô ra 5' được lấp đầy. Khi sử dụng phương pháp thắt TA để thắt bộ chuyển đổi, đoạn DNA cũng cần được phosphoryl hóa ở đầu 5' và thêm "A" ở đầu 3' để bổ sung cho bộ chuyển đổi với đầu dính "T".Quá trình trên được hoàn thành nhờ sự hợp tác của T4 DNA polymerase, T4 polynucleotide kinase và Taq ADN polymerase.
T4 DNA polymerase (Mã số 12901) có hoạt tính DNA polymerase 5'→3', có thể xúc tác quá trình tổng hợp DNA theo hướng 5'→3' và lấp đầy đầu nhô ra 5'. Đồng thời, enzyme cũng có hoạt tính exonuclease 3'→5' để cắt đầu nhô ra 3', do đó biến đổi các đoạn DNA có đầu dính thành DNA đầu cụt.
Vì đầu 5' của mồi PCR tổng hợp và bộ điều hợp thường là nhóm hydroxyl thay vì nhóm phosphate. Do đó, T4 polynucleotide kinase (Cat#12902) là cần thiết để xúc tác quá trình chuyển nhóm γ-phosphate ATP đến đầu 5'-hydroxyl của chuỗi oligonucleotide khi có ATP, để chuẩn bị cho bước tiếp theo của quá trình gắn bộ điều hợp.
S-Taq ADN polymerase (Cat#13486) có hoạt tính polymerase 5'→3', có thể tổng hợp DNA theo hướng 5'→3'. Đồng thời, có hoạt tính deoxynucleotidyl transferase, có thể thêm nucleotide "A" vào đầu 3' của sản phẩm PCR.

Hình 3. Nhiều loại enzyme tham gia vào quá trình sửa chữa cuối cùng
Hình 4. S-taq có hiệu quả rất cao trong việc thêm A vào bốn bazơ ATCG ở đầu 3' của các đoạn gen được phát hiện bằng phương pháp điện di mao quản.
1.3 Thắt ống dẫn
Bộ điều hợp tạo nên một thành phần quan trọng của thư viện. Trong phạm vi giải trình tự Illumina, các bộ điều hợp loại Y thường được sử dụng bao gồm các trình tự SP P5/P7, Index và Rd1/Rd2. Trong số này, trình tự P5/P7 có mục đích ghép nối với trình tự có trên chip giải trình tự, do đó neo các đoạn cần phân tích vào tế bào dòng chảy để thực hiện khuếch đại cầu nối. Trình tự Index được sử dụng để phân biệt giữa các mẫu khác nhau trong thư viện hỗn hợp được giải trình tự, trong khi Rd1/Rd2 SP biểu thị các vùng để liên kết các đoạn mồi giải trình tự Read1 và Read2.
Đối với nhiệm vụ thắt ống dẫn, T4 DNA ligase (Mã số: 12996) là lựa chọn tiêu chuẩn. Nó thể hiện khả năng sửa chữa các vết nứt mạch đơn được tìm thấy trong DNA mạch đôi và kết nối lại các nucleotide liền kề.
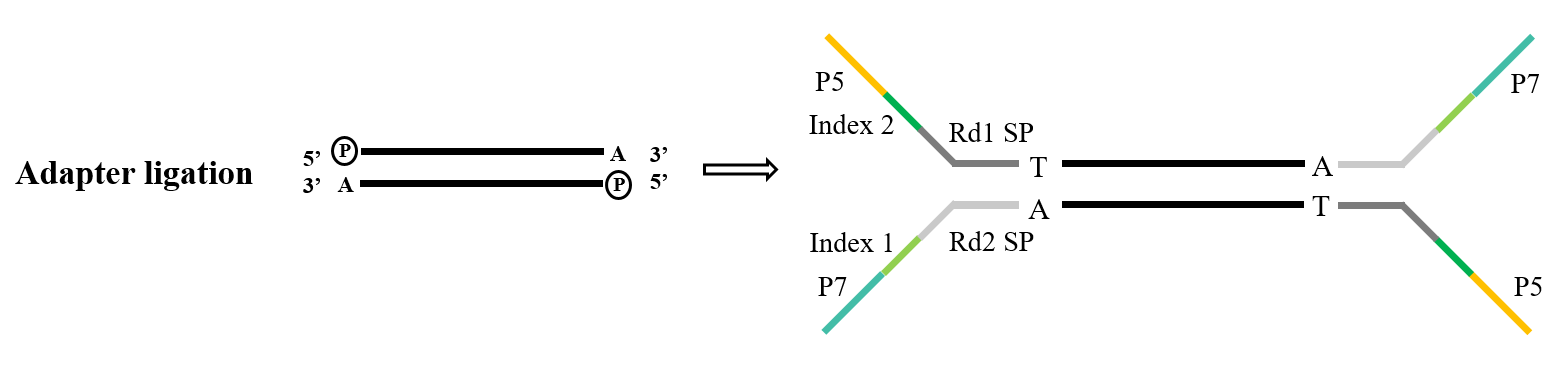
Hình 5. Quy trình thắt ống dẫn chung (Illumina)

Hình 6. Xác minh đột biến T4 DNA Ligase bằng cách gắn DNA 170-bp với bộ điều hợp 80-bp.
1.4 khuếch đại PCR
Thu thập đủ trình tự DNA bằng bộ điều hợp thông qua phản ứng PCR và hoàn tất việc giải trình tự chuỗi axit nucleic mẫu trên máy. Hieff CanaceTM Pro High-Fidelity DNA Polymerase (Cat#13476) thường được sử dụng trong PCR có hoạt tính polymerase 5'→3' và có thể tổng hợp DNA theo hướng 5'→3'. Ngoài ra, nó còn có hoạt tính của exonuclease 3'→5', có thể sửa lỗi kết hợp sai các bazơ trong quá trình khuếch đại, để khuếch đại các đoạn DNA nhanh chóng và có độ trung thực cao.
2. Xây dựng thư viện RNA và các enzyme chính của nó
Theo loại RNA, quá trình xây dựng thư viện RNA có thể được chia thành thư viện mRNA, thư viện LncRNA, v.v. Thư viện RNA thông thường bao gồm các quá trình sau:
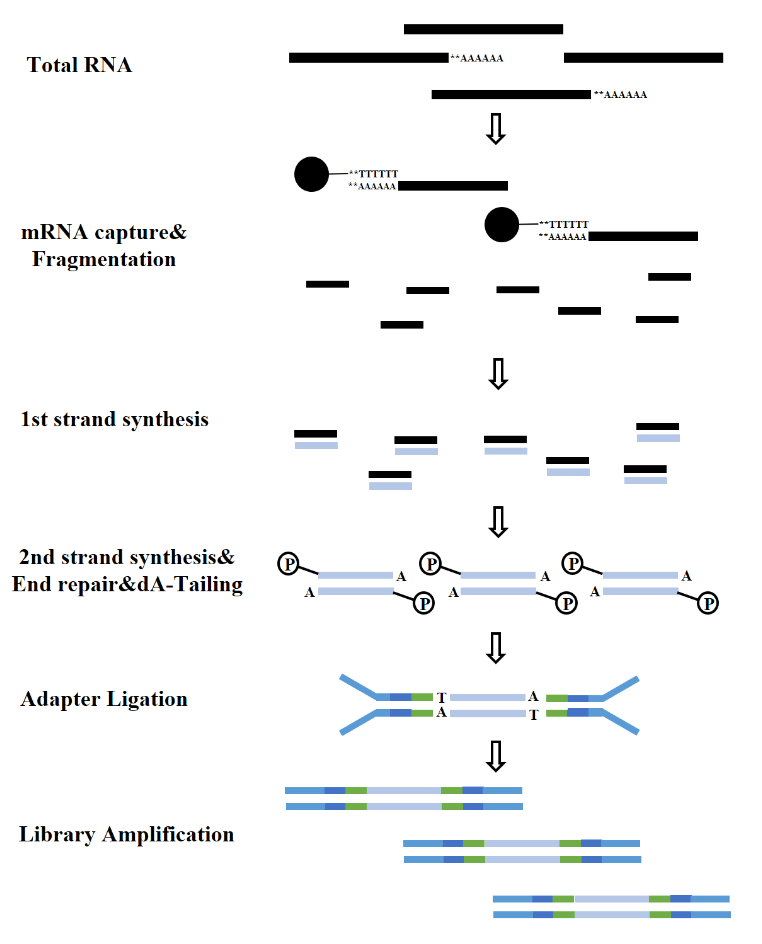
Hình 7. Quy trình xây dựng thư viện mRNA (Illumina)
2.1 Làm giàu RNA
Cho dù là sinh vật nhân chuẩn hay sinh vật nhân sơ, RNA ribosome (rRNA) nổi bật là RNA dồi dào nhất, chiếm tới 80% tổng hàm lượng RNA. Khi giải trình tự trực tiếp tổng RNA của một mẫu, một phần đáng kể dữ liệu giải trình tự sẽ liên quan đến rRNA. Để giảm thiểu sự can thiệp này, phương pháp làm giàu RNA phải được sử dụng. Có hai phương pháp chính cho việc này: làm giàu mRNA dựa trên phương pháp oligo-dT và phương pháp làm cạn kiệt rRNA.
Ở sinh vật nhân chuẩn, mRNA thể hiện cấu trúc poly(A) riêng biệt ở đầu 3'. Các hạt Oligo-dT có thể được sử dụng để thu thập tất cả mRNA được phiên mã từ mẫu, làm cho nó phù hợp để phân tích phiên mã, đặc biệt là với các mẫu RNA chất lượng cao. Mặt khác, các phương pháp làm cạn kiệt rRNA có yêu cầu dễ dãi hơn về chất lượng mẫu và có thể áp dụng cho cả mẫu chất lượng thấp (ví dụ: mẫu FFPE) và mẫu RNA chất lượng cao, cũng như các mẫu sinh vật nhân sơ. Phương pháp thương mại thường được sử dụng bao gồm việc sử dụng tiêu hóa RNase H để loại bỏ rRNA, theo các bước cụ thể sau:
- Tổng hợp các đầu dò oligonucleotide cụ thể được thiết kế để liên kết với rRNA.
- Sử dụng RNase H (Cat#12906), có khả năng phân hủy RNA trong chuỗi lai RNA-DNA, để loại bỏ có chọn lọc rRNA liên kết với các đầu dò.
- Cuối cùng, tiêu hóa các đầu dò DNA với DNase I (Mã số 10325), có thể phân hủy cả DNA sợi đơn và sợi đôi, loại bỏ hiệu quả rRNA. Để biết thêm thông tin về DNase I, bạn có thể theo liên kết này.
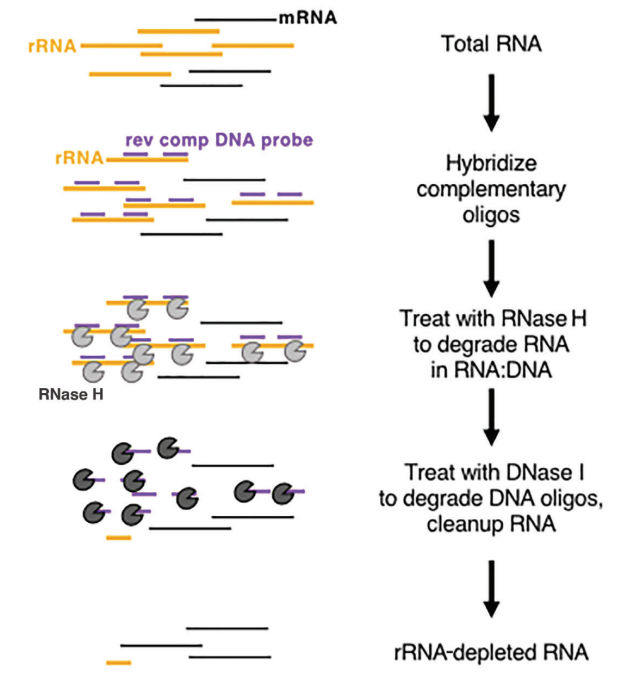
Hình 8: Sơ đồ mô tả sự suy giảm rRNA dựa trên enzyme[5]
2.2 Phân mảnh RNA
Thông thường, dưới tác dụng của cation kim loại hóa trị hai và nhiệt độ cao, các mảnh RNA lớn bị phá vỡ thành các mảnh nhỏ hơn.
2.3 Tổng hợp cDNA sợi thứ nhất
Phiên mã ngược của RNA mục tiêu thu được thành chuỗi đầu tiên của cDNA. Vì RNA dễ bị phân hủy bởi RNase có trong môi trường, nên việc sử dụng Chất ức chế RNase (Cat#14672) trong quá trình phiên mã ngược có thể ức chế hoạt động của các enzyme này và bảo vệ RNA khỏi sự phân hủy RNase. Đồng thời, phiên mã ngược (Cat#11112) được sử dụng để phiên mã ngược RNA khuôn mẫu thành cDNA. Phiên mã ngược có hoạt động DNA polymerase phụ thuộc RNA và có thể sử dụng RNA làm khuôn mẫu để tổng hợp cDNA theo hướng 5'→3'. Sợi đơn của DNA bổ sung cho khuôn mẫu RNA.
Trong ngày 1 tổng hợp cDNA sợi, việc kết hợp actinomycin D chắc chắn đã cải thiện việc xây dựng các thư viện đặc hiệu sợi, tăng cường đáng kể tính đặc hiệu của chuỗi. Sự đổi mới này đã hợp lý hóa quy trình thử nghiệm, đơn giản hóa quy trình cho các nhà nghiên cứu.
Tuy nhiên, actinomycin D có nhược điểm: nó có độc tính và cần được bảo vệ khỏi ánh sáng. Trong bối cảnh nhu cầu ngày càng tăng đối với bộ dụng cụ xây dựng thư viện đĩa và trộn sẵn hiện nay, nhu cầu che chắn khỏi ánh sáng đặt ra những hạn chế đối với sự phát triển của bộ dụng cụ đĩa.
May mắn thay,

Hình 9: Kỹ thuật MMLV để xác định các đột biến MMLV có thể đóng góp vào Standed RNA-seq
2.4 Tổng hợp cDNA sợi thứ 2
cDNA sợi đơn được tạo ra thông qua phiên mã ngược rất không ổn định, đòi hỏi phải tổng hợp ngay sợi cDNA thứ hai dưới ảnh hưởng của DNA polymerase I. Trong quá trình tổng hợp sợi thứ hai này, RNase H phát huy tác dụng bằng cách loại bỏ sợi RNA khỏi cấu trúc lai RNA-DNA. Nó hoạt động phối hợp với DNA polymerase I (Mã số 12903) để tạo điều kiện cho quá trình tổng hợp xúc tác của sợi thứ hai bổ sung của cDNA. DNA polymerase I có hoạt động DNA polymerase 5'→3' và được hướng dẫn bởi khuôn mẫu và mồi, tổng hợp một trình tự bổ sung cho cDNA sợi đơn theo hướng 5'→3'.
Các bước tiếp theo trong quy trình bao gồm sửa chữa đầu, dA-Tailing, gắn bộ điều hợp và khuếch đại PCR, tất cả đều được trình bày chi tiết trong quy trình xây dựng thư viện DNA và không cần phải nhắc lại ở đây. Cần lưu ý rằng sau khi phiên mã ngược hoàn tất, không cần phải phân mảnh thêm đoạn axit nucleic.
3. Hướng dẫn cho các enzyme cốt lõi NGS trong xây dựng thư viện DNA & RNA
Bảng 1.Hướng dẫn cho các enzyme cốt lõi NGS trong xây dựng thư viện DNA & RNA
| Kiểu | Vị trí sản phẩm | Tên sản phẩm | Con mèo# |
| Thư viện RNA sự thi công | rARN suy giảm/tổng hợp cDNA sợi thứ 2 | 12906ES | |
| rARN sự cạn kiệt | 10325ES | ||
| Tổng hợp cDNA sợi thứ nhất | 14672ES | ||
| 11112ES | |||
| Tổng hợp cDNA sợi thứ 2 | 12903ES | ||
| Thư viện RNA sự thi công & ADN thư viện sự thi công | Kết thúc sửa chữa | 12901ES | |
| 12902ES | |||
| dA-Đuôi | 13486ES | ||
| Thắt ống dẫn | 10301ES | ||
| PCR sự khuếch đại | 2×Super Canace® II High-Fidelity Mix cho khuếch đại thư viện | 12621ES |
Bảng 2.Thư viện DNA & RNA Bộ dụng cụ chuẩn bị
| Tên | Con mèo# | Ghi chú | |
| ADN | Bộ dụng cụ chuẩn bị thư viện DNA Hieff NGS | 13577ES | Phương pháp khối u/ cơ học |
| Bộ dụng cụ chuẩn bị thư viện DNA Hieff NGS OnePot Pro V2 | 12194ES | Phương pháp khối u/Enzym | |
| Hieff NGS OnePot II Bộ dụng cụ chuẩn bị thư viện DNA cho Illumina | 13490ES | Pathgen/Enzymetic/thời gian thường xuyên (140 phút) | |
| Bộ dụng cụ chuẩn bị thư viện DNA Flash OnePot NGS của Hieff | 12316ES | Pathgen/Enzyme/Siêu nhanh (100 phút) | |
| Hieff NGS DNA&RNA Library Co-Prep Kit V2 | 12305ES | Pathgen/Enzyme/DNA & RNA Co-Prep | |
| ARN | Bộ dụng cụ chuẩn bị thư viện mRNA chế độ kép Hieff NGS Ultima | 12308ES | Không có hạt từ tính oligo dT, 11 ống |
| Bộ dụng cụ chuẩn bị thư viện mRNA chế độ kép Hieff NGS Ultima | 12309ES | hạt từ tính oligo dT plus, 14 ống | |
| Bộ dụng cụ chuẩn bị thư viện RNA chế độ kép Hieff NGS® Ultima | 12310ES | Phiên bản pha sẵn, 5 ống | |
| Bộ dụng cụ chuẩn bị thư viện RNA Hieff NGS ® EvoMax (Phiên bản trộn sẵn) (actinomycin D Miễn phí) | 12340ES | Phiên bản đã trộn sẵn, (Actinomycin D Miễn phí) | |
| Bộ dụng cụ làm suy giảm rRNA Hieff NGS® MaxUp (Thực vật) | 12254ES | Thực vật | |
| Bộ dụng cụ làm suy giảm rRNA của người Hieff NGS® MaxUp (rRNA & ITS/ETS) | 12257ES | Nhân loại |
Tài liệu tham khảo:
[1] Mardis, Elaine R. Nền tảng giải trình tự thế hệ tiếp theo[J]. Tạp chí đánh giá thường niên về hóa học phân tích, 2013, 6(1):287-303.
[2] Gulilat M, Lamb T, Teft WA, et al. Giải trình tự thế hệ tiếp theo được nhắm mục tiêu như một công cụ cho y học cắt bỏ[J]. BMC Medical Genomics, 2019, 12(1):81.
[3] Lundberg KS, Dan DS, Adams M, et al. Khuếch đại độ trung thực cao sử dụng DNA polymerase chịu nhiệt được phân lập từ Pyrococcus furiosus[J]. Gene, 1991, 108(1):1-6.
[4] Miyazaki K. Phân mảnh DNA ngẫu nhiên với endonuclease V: ứng dụng vào việc xáo trộn DNA[J]. Nghiên cứu axit nucleic, 2002, 30(24):e139.
[5] Baldwin A, Morris AR, Mukherjee N. Một phương pháp dễ dàng, tiết kiệm chi phí và có thể mở rộng để làm cạn kiệt RNA ribosome của con người cho RNA-seq[J]. Giao thức hiện tại, 2021, 1(6):e176.

